Yüksek hızlı süper çözünürlük hızı mikroskobu canlı birincil Cilium içinde uygulanması
Summary
Son zamanlarda ulaşım yolları çeşitli proteinlerin canlı hücrelerdeki birincil kirpikler içinde translocating için üç boyutlu (3D) kayma yerlerini eşleştirilmiş. Burada deneysel kurulum, biyolojik örneklerin işlem ve veri analizleri yaklaşım yeni görüntüleme 3D süper çözünürlük floresan için uygulanan bu kağıt ayrıntıları birincil kirpikler yaşıyorum.
Abstract
Birincil cilium bir Mikrotubul tabanlı çıkıntı yüzeyi birçok ökaryotik hücre ve proteinlerin benzersiz bir tamamlayıcı bu işlev eleştirel hücre hareketliliği ve sinyal içerir. Kirpikler kendi protein sentezleme aciz olduğundan, 200’e yakın benzersiz silier proteinler sitozol ve birincil kirpikler ticareti gerekir. Ancak, hala taşıma yolları bu proteinler arasında şu an mevcut olan sınırlamaları nedeniyle canlı birincil kirpikler için üç boyutlu (3D) yerlerini eşlemek için teknik bir sorun olduğunu teknikleri. Meydan fethetmek için son zamanlarda biz geliştirdik ve tek-noktaya kenar-uyarma alt kırınım (hız) mikroskopi, taşıma yolları için 3D uzaysal konumunu belirlemek için olarak adlandırdığı bir yüksek hızlı sanal 3D süper çözünürlük mikroskobu istihdam hem sitozolik ve birincil kirpikler canlı hücre membran proteinlerinin. Bu makalede, biz hız mikroskobu, silier protein Floresans protein etiketli, gerçek zamanlı tek molekül izleme canlı cilium bireysel proteinlerin ve başarı ifade hücreleri hazırlanması ayrıntılı kurulum gösterecektir 3D mekansal olasılık yoğunluk haritaları silier proteinler için ulaşım yollarının.
Introduction
Ernst Abbe tarafından 1873 yılında belirtilen beri konvansiyonel ışık mikroskobu çözünürlüğe objektif1,2ışık kırınım nedeniyle sınırlı yaklaşık 200 nm olduğuna inanılan. Şu anda, süper çözümleme ışık mikroskobu teknikleri bu sınırlama kırmak ve alt kırınım (< 200 nm) çözünürlük ile dinamik görüntü yakalama sağlar. Teknikleri, genellikle iki geniş kategoriye ayrılır: uyarılmış alt kırınım Aydınlatma Cilt fluorophores doğrusal olmayan optik tepki nedeniyle örnekleri3‘ te; oluşturmak emisyon tükenmesi (STED) mikroskopi dayalı yaklaşımlar ve photoactivated ışık mikroskobu (PALM) ve Stokastik optik imar mikroskobu (fırtına)-fluorophores cisimlerin yerelleştirilmesine ve bu cisimlerin yeniden oluşturmak için Matematiksel işlevler kullanmak süper çözümleme teknikleri dayalı süper çözünürlük görüntüleri4,5oluşturmak için. Şu anda, nispeten basit optik Kur nedeniyle PALM ve fırtına geniş yalnızca küçük bir alt kümesine fluorophores biyolojik hazırlanması uzun bir videonun her çerçevede etkinleştirerek istihdam edilmektedir. Bu daha doğru yerelleştirme için floresan spot, 2D Gauss montaj tarafından noktaya yayılmış işlevi (PSF) olarak adlandırdığı video her çerçevede fluorescently etiketli proteinlerin sağlar. Her fluorescently etiketli molekül 2D konumunu sonra biyolojik hazırlık1,2süper çözünürlük görüntü üretmek için tek bir görüntüleme uçağa bindirilen. Bu tek molekül yerelleştirme sırasında mikroskobu süper çözünürlük yaklaşımlar kesinlikle nasıl görüntüleme biyolojik örneklerin gerçekleştirilen devrim, hala aşılması gereken sorunlar vardır. Örneğin, fırtına ve PALM onların en iyi uzamsal çözünürlük sonra biyolojik örneklerin fiksasyonu elde etmek ve böylece fluorescently etiketli proteinler, statik bir temsili olan elektron mikroskobu benzer bir kısıtlamasıdır mevcut. Ayrıca, canlı hücrelerdeki her fluorescently etiketli protein için yüksek Uzaysal çözünürlük elde etmek için örnekleri protein dynamics yakalamak mümkün değildir çok uzun framerate yansıması gerekir. Bu nedenle, bu ana teknik engellerin üstesinden gelmek gereklidir.
Çok hızlı hareket eden protein veya RNA’ların canlı hücrelerde algılamak için uygundur yüksek bir kronolojik zamanmekansal çözünürlük elde etmek için biz süper çözünürlük hızı mikroskobu bizim laboratuvar (şekil 1)6,7‘, gelişen 8. hız mikroskobu birkaç önemli teknik gelişmeler bizi küçük moleküllerin nucleocytoplasmic taşıma başarılı bir şekilde izlemek daha önce etkinleştirdiyseniz, proteinler, mRNA ve virüs-den geçerek yerli nükleer gözenek kompleksleri (NPCs)6, 7 , 8. kısaca, hız mikroskobu, aşağıdaki özellikleri hızlı hareket eden oluştururlar alt mikrometre rotasyonel simetrik yapıları NPCs ve birincil kirpikler gibi canlı hücrelerdeki üzerinden izlemek için kullanılan: (1) bir eğimli veya bir Dikey aydınlatma PSF odak düzlemi (şekil 1); küçük kırınım-limit hacmindeki içinde tek moleküllerin uyarma sağlar (2) eğimli PSF büyük ölçüde odak floresan önlemek ve böylece sinyal-gürültü oranı geliştirmek. (3) 100-500 kW optik yoğunluk / cm2 aydınlatma PSF’hızlı algılama hızı (> 500 Hz) ile tek fluorophores üzerinden tahsil edilecek fotonlar binlerce sağlar. (4) hızlı algılama hızı da büyük ölçüde azaltır tek molekül kayma yerelleştirme hata (< 10 nm) moleküler Difüzyon önemli faktörlerden biri olduğu için canlı hücrelerde, floresan molekülleri hareket kayma yörüngeleri belirlemede molekülleri hareket etmek için tek molekül yerelleştirme kusurları neden. (5) well-established 2D 3D dönüştürme algoritmaları molekülleri NPC veya birincil cilium 3D mekansal olasılık yoğunluk haritalar ulaşım yollarının sağlamamıza olanak sağlar. Bu bizim dönüştürme işlemi Kartezyen ve silindirik koordinasyon sistemi arasında bir 3D mekansal olasılık yoğunluk 3D yerine harita tek molekül izleme (Şekil 2) oluşturmak için kullanılan dikkat çekicidir. Daha önce elektron mikroskobu veri-si olmak kâşif NPC9,10 ve11 birincil cilium hem de rotasyonel simetrik bir yapıya sahip. Prensip olarak, rasgele NPC veya birincil cilium aracılığıyla hareketli moleküllerin Difüzyon rotasyonel simetrik dağıtımları da olmalıdır. Şekil 2‘ de gösterildiği gibi rastgele silindir içinde moleküllerin Difüzyon yüksek bir dizi bu kesit görünümü, rotasyonel simetrik dağıtımları daha fazla bir yaklaşık Tekdüzen kaynaklanan NPC oluşturmak kayma Dağıtım iki komşu yüzük (Şekil 2E) arasında çok küçük her alt bölge içerisinde. Bu tekdüze dağılım silindirik sistemdeki θ boyuttaki kayma dağıtım sabittir yol açar. Sonra 3D koordinatları (R, X, θ)-ebilmek var olmak kolaylaştırmak-2B koordinatları (R, X, Sabit) olmak. Aslında, bizim dönüştürme işlemi Kartezyen ve silindirik sistemleri arasındaki 2D (R, X, Sabit) 2D (X, Y) etmektir. Sürekli θ, Şekil 2Emekansal yoğunluk p başvurduğu, ADenklem kullanılarak hesaplanır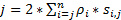 .
.
Sonuçta, tek molekül izleme geniş uygulama Biyolojik araştırma vardır, böylece, bu teknikleri bir bolluk belirli biyolojik nişler12,13,14doldurmak için geliştirilecek doğaldır. Böyle hız mikroskobu ile durumdur. Daha önce bir 3B dönüştürme algoritması ile birleştiğinde, moleküller NPCs, alt diffraction ölçekli ve rotasyonel simetrik biyolojik yapısı6üzerinden transit, 3D ulaşım yolları çözmek için bu teknik geliştirilmiştir. Bu yazıda, birincil kirpikler mükemmel model organelleri de olduğu gösterilmiştir. Birincil kirpikler en memeli hücreleri15,16,17yüzeyinden Project’in silindirik, anten gibi organellerin (~ 125 nm RADIUS) vardır. Onlar dış sinyalleri alma ve genellikle büyüme ve metabolizma15,16ile ilgili hücre içi bir yanıt gönderme için sorumludur. Bu nedenle, yapısal proteinlerin akı, geri dönüşüm transmembran reseptörler ve hücre içi haberci iletimini birincil kirpikler önemli sorumlulukları vardır. Dönemde birincil kirpikler ve hücre beden arasındaki geçiş bölgesi veya TZ, hangi aracılığıyla bu protein taşıma11,18,19ortaya çıkması gereken bir kritik seçicilik engel vardır, 20. TZ gating işleve ek olarak, en az iki taşıma işlemi, intraflagellar taşıma ve pasif difüzyon, protein bu bölge16,21, üzerinden hareket sorumlu olduğu düşünülür 22. bir insan sağlığı açısından birincil kirpikler kaybı ve aşağı akım sinyal sonraki deregülasyon birçok kanser özelliğidir. Buna ek olarak, Bardet-Biedl sendromu ve Polikistik böbrek hastalığı, gibi birçok genetik hastalıkların arızalı protein taşıma23ile ilişkilidir. Alt kırınım boyutu sınırı ve seçici protein taşıma TZ aracılığıyla karmaşık süreci birincil kirpikler için bu tekniği ilk hedef olun. Bu yöntemleri yazıda, biz silier transmembran protein, somatostatin reseptörü 3 (SSTR3) izleme gösterecektir24dışarıdan Alexa Fluor 647 ve bir bileşeni olan bir yuvarlak GFP molekül ile etiketli LFT, IFT2025, taşır,.
Protocol
Representative Results
Discussion
Bu iletişim kuralı hız mikroskobu uygulamaya birincil cilium, son derece verimli protein araçta güvenen bir hücresel sinyal organel açıklar. Onlar TZ üzerinde ortalanmış tek nokta aydınlatma yoluyla geçerken hız mikroskobu (< 10 nm) konumları fluorescently etiketli moleküller için yüksek çözünürlüklü sağlayabilir. Daha önce NPC6,7,8ile ticareti protein çalışmaya uygulanmıştır. Ancak, herhangi bir …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Biz Dr Kristen Verhey (Michigan Üniversitesi, Ann Arbor) ve Dr. Gregory Pazour (Massachusetts Üniversitesi Tıp Fakültesi) bazı plazmid verdiğiniz için teşekkür ederiz. Projenin Ulusal Sağlık Enstitüleri (NIH GM097037, GM116204 ve GM122552 W.Y. için) gelen hibe tarafından desteklenmiştir.
Materials
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25×36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
References
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell’s antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).

