Microscopia confocale rivela l'aggregazione di recettore di superficie delle cellule tramite spettroscopia di correlazione di immagine
Summary
Gli anticorpi che si legano ai recettori bersaglio sulla superficie delle cellule possono conferire conformazione e alterazioni clustering. Questi cambiamenti dinamici hanno implicazioni per caratterizzare lo sviluppo di farmaci nelle cellule bersaglio. Questo protocollo utilizza la microscopia confocale e spettroscopia di correlazione di immagine attraverso ImageJ/FIJI per quantificare l’entità del recettore sulla superficie cellulare di clustering.
Abstract
Microscopia confocale fornisce una metodologia accessibile per catturare subcellulare interazioni critiche per la caratterizzazione e l’ulteriore sviluppo di pre-clinici agenti etichettati con sonde fluorescenti. Con gli avanzamenti recenti nella droga citotossica anticorpo basato su sistemi di consegna, comprendere le alterazioni indotte da questi agenti all’interno del Regno di aggregazione del recettore e di interiorizzazione è di importanza critica. Questo protocollo sfrutta la metodologia consolidata di immunocytochemistry fluorescente e la distribuzione di FIJI di open source di ImageJ, con la sua autocorrelazione integrato e funzioni matematiche di immagine, per eseguire la correlazione di immagine spaziale spettroscopia (ICS). Questo protocollo quantitates l’intensità di fluorescenza dei recettori con etichettati in funzione della zona di fascio del microscopio confocale. Questo fornisce una misura quantitativa dello stato di aggregazione della molecola bersaglio sulla superficie delle cellule. Questa metodologia è focalizzata sulla caratterizzazione di celle statiche con potenziale di espandersi in indagini temporale dell’aggregazione del recettore. Questo protocollo presenta una metodologia accessibile per fornire quantificazione degli eventi che si verificano alla superficie delle cellule, utilizzando ben stabilito non specializzati imaging apparato e tecniche di clustering.
Introduction
Lo sviluppo di anticorpi terapeutici ha dimostrato notevole successo nel trattamento del tumore più tipi1. I recenti progressi dei coniugati di anticorpo-farmaco (ADC) come meccanismi di consegna per composti citotossici ha ampliato i requisiti per comprendere le dinamiche delle interazioni anticorpo: recettore dalla cella di superficie2. Dopo il successo di targeting di un anticorpo per il recettore di superficie delle cellule, questi complessi possono indurre modelli di aggregazione simili a quelle osservate nel ligando: anticorpo interazioni3. Alterazioni nell’aggregazione del recettore possono indurre cambiamenti alla membrana e risultato nell’internalizzazione del recettore e la sua rimozione dalla superficie delle cellule. Nel contesto di un anticorpo-farmaco coniugato, questo processo rilascia successivamente il payload citotossico in endosomi interiorizzati e, successivamente, il citoplasma, con conseguente uccisione efficace delle cellule.
Microscopia confocale ha fornito un mezzo efficace di visualizzare queste interazioni importanti degli anticorpi e la loro destinazione recettori4. Per esplorare i cambiamenti di aggregazione delle molecole alla superficie delle cellule questo protocollo utilizza la post-elaborazione di immagini di microscopia confocale via un immagine spaziale correlazione spettroscopia (ICS) tecnica 5,6,7 .
La Fondazione di spettroscopia di correlazione di immagine è l’osservazione che le fluttuazioni di intensità di fluorescenza spaziale condividano un rapporto con lo stato di densità e aggregazione delle strutture con etichettate. Questa relazione è stabilita dopo il calcolo di una funzione di autocorrelazione spaziale di un immagine catturata5.
Tutte le varianti di spettroscopia di correlazione di immagine richiedono il calcolo di un’immagine di autocorrelazione. Questo è seguito inserendo questa funzione ad una curva gaussiana bidimensionale per l’estrazione dei parametri di stato di aggregazione quantitativi contenuti nell’immagine. In termini semplici il calcolo di un’immagine di autocorrelazione implica il confronto tutte le coppie possibili pixel contenuti all’interno di un’immagine e calcolare la probabilità che sia altrettanto brillante come l’altro. Questo viene visualizzato come una funzione della distanza e direzioni di pixel separazione8.
Il quadro teorico per la spettroscopia di correlazione di immagine è stato stabilito e definito da Petersen e Wiseman et al. 5 , 6. nel presente protocollo, vengono eseguiti i calcoli di autocorrelazione in Fiji/ImageJ, nonché un’applicazione di foglio di calcolo, la base per la funzione di autocorrelazione spaziale di intensità fluttuazione può essere descritto come (Eq. 1):
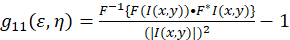
dove F rappresenta la trasformata di Fourier; F− 1 la trasformata di Fourier trasformazione inversa; F * suo complesso coniugato; e il ritardo spaziale variabili ε e η. In ICS spaziale, come descritto in questo protocollo, la funzione di autocorrelazione può essere calcolata usando un 2D fast Fourier transform algoritmo7,9. L’autocorrelazione a zero separazione spaziale, altrimenti noto come zero-lag, g11(0,0), fornisce l’inverso numero medio di particelle presenti per area di larghezza del microscopio. Può essere ottenuto inserendo la funzione di autocorrelazione spaziale a una funzione gaussiana bidimensionale (Eq. 2):
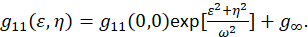
Come i pixel acquisiti all’interno di un’immagine sono contenuti all’interno di una area set e queste misurazioni non si estendono all’infinito, il g∞ di termine è utilizzato come un offset per tenere conto delle correlazioni spaziali a lungo raggio contenute all’interno dell’immagine. Per gli aggregati di dimensioni molecolari, ω è la funzione di punto di diffusione del microscopio e descritto dalla larghezza piena a metà-al massimo la funzione di autocorrelazione spaziale. L’area contenuta all’interno della funzione di diffusione del punto dello strumento può essere calibrato attraverso l’uso di perline fluorescenti Sub-resolution.
Per protocollo di spettroscopia di correlazione di immagine descritto nel presente documento, l’autocorrelazione e funzioni matematiche necessarie per completare ICS vengono effettuate mediante la piattaforma di imaging-elaborazione di open-source, Fiji10, una distribuzione del ImageJ programma11,12. Fiji/ImageJ utilizza la trasformazione di Fourier veloce preinstallata nella funzione FFT matematica. Questa funzione riduce il tempo di calcolo necessario di questo calcolo, riducendo l’intervallo di dati da un fattore di due in ogni dimensione13. Come la funzione di autocorrelazione 2D è approssimativamente simmetrica in x, asse y, una singola linea profilo trama attraverso l’immagine di autocorrelazione può essere utilizzata per misurare l’autocorrelazione crudo come una funzione del GAL spaziale. Qualsiasi rumore zero-lag è rimosso prima dell’ulteriore calcolo, con l’ampiezza risultante di autocorrelazione (valore di picco, g(0)T) corretto per lo sfondo con l’espressione (Eq 3):
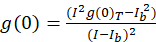
dove hob è l’intensità media da una regione di sfondo esclusa la cella. La densità di cluster, o la densità di oggetti fluorescenti, è definito dal (Eq. 4):
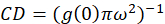
Nel protocollo descritto nel presente documento, inoltre semplicemente il calcolo della densità di cluster (CD), con il presupposto basato sull’osservazione che una funzione di autocorrelazione normalizzato decadrà su un valore che si avvicina 1.0 con ritardo spaziale crescente. Con massimo ritardo spaziale, non c’è più alcuna correlazione di fluorescenza i valori di intensità e così senza una correlazione i calcoli in questa regione sono calcolo del valore di intensità moltiplicato questa intensità che viene successivamente diviso per la Piazza di tale intensità, che per definizione è uguale a 1.0. Così, densità di cluster da una funzione di autocorrelazione normalizzato può essere calcolata sottraendo 1.0 dalla funzione di autocorrelazione normalizzato prima della presa del suo reciproco (Eq 5):
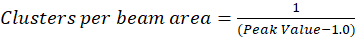
Ulteriore calibrazione della zona fascio può essere effettuata per quantificare il numero di cluster contenute all’interno dell’area della funzione punto diffusione dello strumento. Questa taratura deve essere eseguita utilizzando le stesse condizioni ottiche utilizzate durante l’analisi di spettroscopia di correlazione immagine.
Protocol
Representative Results
Discussion
La tecnica di spettroscopia di correlazione di immagine (ICS) che descriviamo in questo protocollo utilizza microscopi confocali standard senza la necessità di rilevatori specializzati. La tecnica ICS descritta utilizza metodi di immunocitochimica consolidata per fornire per il campionamento rapido di più condizioni di trattamento per l’analisi statistica aumentata. Questa metodologia fa con una leggera riduzione della precisione assoluta rispetto alle tecniche di singola molecola alternativo basato sulla correlazione …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gli autori riconoscono finanziamento da NHMRC (Fellowship 1084178 e borse di studio 1087850, 1030469, 1075898 (AMS)), Cancer Australia, Ludwig Cancer Research, John T Reid si fida, cura Brain Cancer Foundation, La Trobe University e l’agenzia del cancro di Victoria. Finanziamenti dal programma di supporto di infrastrutture operative fornite dal governo vittoriano, Australia è anche riconosciuto.
Materials
| Nunc Lab-Tek II Chambered Coverglass – 8 well | ThermoFisher Scientific | 155409 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Fetal Bovine Serum | ThermoFisher Scientific | 10099141 | |
| TrypLE Express Enzyme (1x), no phenol red | ThermoFisher Scientific | 12604021 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | |
| Goat anti-Human IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | ThermoFisher Scientific | A11013 | |
| TetraSpeck Fluorescent Microsphere Standards 0.1µm | ThermoFisher Scientific | T7279 | |
| Cetuximab | Merck Serono | 3023715501 | |
| Parafilm M 38mx100mm | Merck Millipore | BRND701605 | |
| 16% Paraformaldehyde (formaldehyde) aqueous solution | ProSciTech | C004 | |
| Recombinant Human EGF | R&D System | 236-EG |
References
- Scott, A. M., Wolchok, J. D., Old, L. J. Antibody therapy of cancer. Nature reviews. Cancer. 12 (4), 278-287 (2012).
- Parslow, A. C., Parakh, S., Lee, F. -. T., Gan, H., Scott, A. Antibody-Drug Conjugates for Cancer Therapy. Biomedicines. 4 (3), 14 (2016).
- Sorkin, A., Waters, C. M. Endocytosis of growth factor receptors. BioEssays. 15 (6), 375-382 (1993).
- Pawley, J. . Handbook of Biological Confocal Microscopy. , (2006).
- Petersen, N. O., Höddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophysical Journal. 65 (3), 1135-1146 (1993).
- Wiseman, P. W., Petersen, N. O. Image Correlation Spectroscopy. II. Optimization for Ultrasensitive Detection of Preexisting Platelet-Derived Growth Factor-β Receptor Oligomers on Intact Cells. Biophysical Journal. 76 (2), 963-977 (1999).
- Costantino, S., Comeau, J. W. D., Kolin, D. L., Wiseman, P. W. Accuracy and Dynamic Range of Spatial Image Correlation and Cross-Correlation Spectroscopy. Biophysical Journal. 89 (2), 1251-1260 (2005).
- Claire Robertson, S. C. G. Theory and practical recommendations for autocorrelation-based image correlation spectroscopy. Journal of Biomedical Optics. 17 (8), 080801 (2012).
- Ciccotosto, G. D., Kozer, N., Chow, T. T. Y., Chon, J. W. M., Clayton, A. H. A. Aggregation Distributions on Cells Determined by Photobleaching Image Correlation Spectroscopy. Biophysical Journal. 104 (5), 1056-1064 (2013).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics International. 11 (7), 36-42 (2004).
- Collins, T. J. ImageJ for microscopy. BioTechniques. 43 (1 Suppl), 25-30 (2007).
- Rappaz, B., Wiseman, P. W. Image correlation spectroscopy for measurements of particle densities and colocalization. Current protocols in cell biology. , (2013).
- tferr/Scripts: BAR 1.5.1. Zenodo Available from: https://zenodo.org/record/495245 (2017)
- Elson, E. L. Fluorescence Correlation Spectroscopy: Past, Present, Future. Biophysical Journal. 101 (12), 2855-2870 (2011).
- Jares-Erijman, E. A., Jovin, T. M. Imaging molecular interactions in living cells by FRET microscopy. Current opinion in chemical biology. 10 (5), 409-416 (2006).
- Huang, B., Bates, M., Zhuang, X. Super-Resolution Fluorescence Microscopy. dx.doi.org.ez.library.latrobe.edu.au. 78 (1), 993-1016 (2009).
- Schermelleh, L., Heintzmann, R., Leonhardt, H. A guide to super-resolution fluorescence microscopy. The Journal of Cell Biology. 190 (2), 165-175 (2010).
- Nohe, A., Petersen, N. O. Image Correlation Spectroscopy. Sci. Signal. (417), pl7 (2007).

