Neutronenspin-Echospektroskopie als einzigartige Sonde für Lipidmembrandynamik und Membran-Protein-Wechselwirkungen
Summary
Dieser Artikel beschreibt die Protokolle für die Probenvorbereitung, Datenreduktion und Datenanalyse in Neutronenspinechostudien (NSE) von Lipidmembranen. Eine umsichtige Deuteriummarkierung von Lipiden ermöglicht den Zugang zu unterschiedlichen Membrandynamiken auf mesoskopischen Längen- und Zeitskalen, über die lebenswichtige biologische Prozesse ablaufen.
Abstract
Lipiddoppelschichten bilden die Hauptmatrix der Zellmembranen und sind die primäre Plattform für Nährstoffaustausch, Protein-Membran-Interaktionen und virale Knospung, neben anderen lebenswichtigen zellulären Prozessen. Für eine effiziente biologische Aktivität sollten Zellmembranen starr genug sein, um die Integrität der Zelle und ihrer Kompartimente aufrechtzuerhalten, aber flüssig genug, um Membrankomponenten wie Proteine und funktionelle Domänen zu zerstreuen und interagieren zu lassen. Dieses empfindliche Gleichgewicht zwischen elastischen und flüssigen Membraneigenschaften und deren Einfluss auf die biologische Funktion erfordern ein besseres Verständnis der kollektiven Membrandynamik über mesoskopische Längen- und Zeitskalen wichtiger biologischer Prozesse, z. B. Membranverformungen und Proteinbindungsereignisse. Zu den Techniken, die diesen Dynamikbereich effektiv untersuchen können, gehört die Neutronenspinechospektroskopie (NSE). In Kombination mit der Deuterium-Markierung kann NSE verwendet werden, um direkt auf Biege- und Dickenschwankungen sowie auf die mesoskopische Dynamik ausgewählter Membranmerkmale zuzugreifen. Dieses Papier bietet eine kurze Beschreibung der NSE-Technik und beschreibt die Verfahren zur Durchführung von NSE-Experimenten an liposomalen Membranen, einschließlich Details zur Probenvorbereitung und Deuteration von Schemata sowie Anweisungen zur Datenerhebung und -reduktion. Das Papier stellt auch Datenanalysemethoden vor, die verwendet werden, um wichtige Membranparameter wie den Biegesteifigkeitsmodul, den Flächenkompressibilitätsmodul und die Viskosität in der Ebene zu extrahieren. Um die biologische Bedeutung von NSE-Studien zu veranschaulichen, werden ausgewählte Beispiele für Membranphänomene diskutiert, die von NSE untersucht wurden, nämlich die Wirkung von Additiven auf die Membranbiegesteifigkeit, den Einfluss der Domänenbildung auf Membranfluktuationen und die dynamische Signatur von Membran-Protein-Interaktionen.
Introduction
Das Verständnis von Zellmembranen und ihrer Funktion hat sich in den letzten Jahrzehnten bemerkenswert weiterentwickelt. Die frühere Sichtweise von Zellmembranen als passive Lipiddoppelschichten, die Zellgrenzen definieren und Membranproteine1 beherbergen, hat sich allmählich in ein dynamisches Modell verwandelt, in dem Lipiddoppelschichten eine wichtige Rolle bei der Regulierung lebenswichtiger biologischer Prozesse spielen, einschließlich zellulärer Signalgebung, molekularer Austausch und Proteinfunktion – um nur einige zu nennen2,3,4,5,6. Diese Erkenntnis, dass Zellmembranen hochdynamisch sind und ständig umgestaltet und molekular umverteilt werden, hat wissenschaftliche Erkundungen jenseits der Gleichgewichtsstrukturen der Membranen7,8,9vorgeben. Dementsprechend wurden mehrere Ansätze entwickelt, um die verschiedenen dynamischen Modi in biologischen und bioinspirierten Lipidmembranen zu untersuchen. Bisher konzentrierte sich die Mehrheit dieser Studien hauptsächlich auf diffusive molekulare Bewegungen10 , 11,12,13 und makroskopische Formfluktuationen14,15,16, die eine signifikante Lücke im Verständnis der mittleren Membrandynamik hinterlassen, d.h. kollektive Fluktuationen von Lipidanordnungen, die aus wenigen 10-100s Lipidmolekülen bestehen. Diese Dynamik tritt über Längenskalen von wenigen zehn bis wenigen 100 Å und über Zeitskalen von Sub-ns bis zu einigen hundert ns auf (siehe Abbildung 1),hier als mesoskopische Skalen bezeichnet. Auf diesen Skalen findet die biologische Schlüsselaktivität auf derMembranebene 17statt. Dazu gehören virale Knospen18, Kanal-Gating19und Membran-Protein-Interaktionen20. Es ist auch wichtig darauf hinzuweisen, dass die Energielandschaft der Membranproteine21,22 zeigt, dass Konformationsänderungen in Proteinen – notwendig für ihre regulatorische Rolle – über die ns-Zeitskalen23 kollektiver Membranfluktuationen stattfinden, was die Bedeutung der mesoskopischen Dynamik in der biologischen Funktion von Zellmembranen und ihren bioinspirierten Analoga weiter unterstreicht20. Dieser Beitrag konzentriert sich auf die beiden primären mesoskopischen dynamischen Modi in Lipidmembranen, nämlich Biegeschwankungen und Dickenschwankungen.
Die größte Herausforderung bei der direkten Untersuchung dieser Fluktuationsmodi ist die Schwierigkeit, gleichzeitig auf ihre räumlichen und zeitlichen Skalen mit Standardspektroskopiemethoden zuzugreifen. Die andere Herausforderung besteht darin, dass direkte Kontakttechniken die gleichen Schwankungen beeinflussen könnten, die sie messen sollen16. Dies wird durch die kompositorische und strukturelle Komplexität der biologischen Membranen24,25nochverschärft,was zu inhomogenen Membranmerkmalen führt, einschließlich der Lipiddomänenbildung26,27,28,29,30 und Membranasymmetrie31,32,33– die selektive Sonden erfordern, um die Dynamik verschiedener Membranmerkmale zu verstehen. Glücklicherweise können diese Herausforderungen mit nicht-invasiven Neutronenspektroskopiemethoden wie Neutronenspinecho (NSE) überwunden werden, die von Natur aus auf die erforderlichen Längen- und Zeitskalen zugreifen und weitere Untersuchungen selektiver Membranmerkmale ermöglichen, ohne ihre physikalisch-chemische Umgebung zu verändern34. Tatsächlich hat sich die NSE-Spektroskopie in den letzten Jahren zu einer einzigartigen und leistungsstarken Sonde der kollektiven Membrandynamik entwickelt35. Ergebnisse aus NSE-Studien an Lipidmembranen haben neue Erkenntnisse über mechanische36,37 und viskoelastische38,39 Eigenschaften von Lipidmembranen geliefert und ein neues Licht auf ihre mögliche Rolle in der biologischen Funktion geworfen40,41.
Die NSE-Spektroskopietechnik basiert auf einem interferometrischen Instrumentendesign, das erstmals von Mezei42vorgeschlagen wurde und eine Reihe von Spin-Flippern und Magnetspulen verwendet, um die Präzession des Neutronenspins zu steuern, während Neutronen das Instrument durchqueren. Das Design beruht auf der magnetischen Spiegelung der Magnetfeldelemente in Bezug auf die Probenposition (Abbildung 1A). Dies bedeutet, dass das Neutron in Abwesenheit eines Energieaustauschs zwischen dem Neutron und der Probe in der ersten und zweiten Hälfte des Instruments die gleiche Anzahl von Spin-Präzessionen in entgegengesetzte Richtungen durchführt (beachten Sie den π-Flipper zwischen den beiden Präzessionsspulen). Infolgedessen bleibt der endgültige Spinzustand des Neutrons relativ zum Ausgangszustand unverändert – ein Phänomen, das als Spin-Echo bezeichnet wird (siehe transparentes Neutron in Abbildung 1A). Wenn das Neutron jedoch energetisch mit der Probe wechselwechselt, verändert der Energieaustausch die Anzahl der Spin-Präzessionen in der zweiten Hälfte des Instruments, was zu einem anderen endgültigen Spin-Zustand führt (siehe Abbildung 1A). Dies wird experimentell als Polarisationsverlust nachgewiesen, wie später in diesem Artikel gezeigt wird. Für weitere Einzelheiten zur NSE-Technik wird der Leser auf die speziellen technischen Papiere42,43,44,45verwiesen.
Hier wird eine vereinfachte Beschreibung präsentiert, um eine grobe Schätzung der Längen- und Zeitskalen zu liefern, die mit NSE zugänglich sind. Die Längenskalen werden durch den Bereich der erreichbaren Wellenvektortransfers bestimmt, Q = 4π sin θ/λ, wobei 2θ der Streuwinkel und λ die Neutronenwellenlänge ist. Man kann sehen, dass Q durch den Wellenlängenbereich und das Ausmaß der Rotation des zweiten Arms des Spektrometers eingestellt wird (siehe Abbildung 1A). Ein typischer Q-Bereichauf NSE-Spektrometern ist ~0,02-2 Å-146,47und bis zu 0,01-4 Å-1 mit den jüngsten Upgrades48,49, entsprechend räumlichen Skalen von ~ 1-600 Å. Auf der anderen Seite wird die zugängliche Zeitskala aus dem gesamten Präzessionswinkel (oder der Phase) berechnet, der vom Neutron innerhalb der magnetischen Präzessionsspulen erfasst wird, und es wird festgestellt, dasses 50: 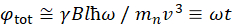 . In diesem Ausdruck ist t die Fourierzeit, definiert als
. In diesem Ausdruck ist t die Fourierzeit, definiert als 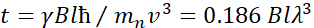 , wobei das
, wobei das 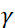 Neutronen-Gyromagnet-Verhältnis,
Neutronen-Gyromagnet-Verhältnis, 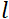 die Spulenlänge und
die Spulenlänge und 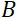 die Stärke des Magnetfeldes der Spule ist. Es ist erwähnenswert, dass die Fourierzeit eine Größe ist, die streng von der Instrumentengeometrie, der Magnetfeldstärke und der Neutronenwellenlänge abhängt. Zum Beispiel wird die Fourierzeit unter Verwendung von Neutronen mit einer Wellenlänge
die Stärke des Magnetfeldes der Spule ist. Es ist erwähnenswert, dass die Fourierzeit eine Größe ist, die streng von der Instrumentengeometrie, der Magnetfeldstärke und der Neutronenwellenlänge abhängt. Zum Beispiel wird die Fourierzeit unter Verwendung von Neutronen mit einer Wellenlänge 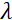 = 8 Å und Instrumenteneinstellungen von
= 8 Å und Instrumenteneinstellungen von 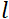 =
= 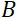 1,2 m und = 0,4 T als t ~ 50 ns berechnet. Experimentell wird die Fourierzeit durch Änderung des Stroms in den Präzessionsspulen (d. H. Magnetfeldstärke) oder unter Verwendung verschiedener Neutronenwellenlängen abgestimmt, was zu typischen NSE-Zeitskalen von ~ 1 ps bis 100 ns führt. Jüngste Upgrades bei NSE-Spektrometern haben jedoch den Zugriff auf längere Fourier-Zeiten ermöglicht, bis zu ~ 400 ns auf dem J-NSE-Phoenix-Spektrometer am Heinz Maier-Leibnitz Zentrum51 und dem SNS-NSE-Spektrometer am Oak Ridge National Lab48und bis zu ~ 1.000 ns am IN15 NSE-Spektrometer am Institut Laue-Langevin (ILL)49.
1,2 m und = 0,4 T als t ~ 50 ns berechnet. Experimentell wird die Fourierzeit durch Änderung des Stroms in den Präzessionsspulen (d. H. Magnetfeldstärke) oder unter Verwendung verschiedener Neutronenwellenlängen abgestimmt, was zu typischen NSE-Zeitskalen von ~ 1 ps bis 100 ns führt. Jüngste Upgrades bei NSE-Spektrometern haben jedoch den Zugriff auf längere Fourier-Zeiten ermöglicht, bis zu ~ 400 ns auf dem J-NSE-Phoenix-Spektrometer am Heinz Maier-Leibnitz Zentrum51 und dem SNS-NSE-Spektrometer am Oak Ridge National Lab48und bis zu ~ 1.000 ns am IN15 NSE-Spektrometer am Institut Laue-Langevin (ILL)49.
Neben dem direkten Zugriff auf die Längen- und Zeitskala der Membrandynamik verfügt NSE über die inhärenten Fähigkeiten der Neutronenisotopenempfindlichkeit52. Insbesondere die Fähigkeit von Neutronen, unterschiedlich mit den Isotopen von Wasserstoff, dem am häufigsten vorkommenden Element in biologischen Systemen, zu interagieren, führt zu einer anderen Neutronenstreulängendichte,34 oder NSLD (das Äquivalent des optischen Brechungsindex50),wenn Protium durch Deuterium ersetzt wird. Dies ermöglicht einen Ansatz, der als Kontrastvariation bekannt ist und häufig verwendet wird, um bestimmte Membranmerkmale hervorzuheben oder andere zu verbergen – das letztere Szenario wird als Kontrastabgleich bezeichnet. Eine häufige Anwendung von Kontrastvariation/Matching ist die Substitution von Wasser (NSLD = -0,56 × 10-6 Å-2) durch schweres Wasser oderD2O (NSLD = 6,4 × 10-6 Å-2), um das Neutronensignal von protiierten Lipidmembranen (NSLD ~ 0 × 10-6 Å-2) zu verstärken. Dieser Ansatz ist bei Untersuchungen der Membranstruktur sehr effektiv, da das Eindringen vonD2Oin den Kopfgruppenbereich der Membran eine genaue Bestimmung der Membrandicken (siehe Abbildung 2A, linkes Feld) und der Lage verschiedener Lipiduntergruppen ermöglicht, wenn anspruchsvollere Modelle angewendet werden53,54. Dieser Artikel hebt einige Beispiele für die Verwendung von Kontrastvariationen für Studien der kollektiven Dynamik in biomimetischen Membranen und ausgewählten Membranmerkmalen hervor.
Hier wird die Wirksamkeit von NSE bei der Bereitstellung einzigartiger Einblicke in dynamische und funktionelle Membraneigenschaften anhand konkreter Beispiele von NSE-Studien zu Modell- und biologisch relevanten Lipidmembransystemen mit Schwerpunkt auf mesoskaliger Dynamik in freistehenden Membranen in Form von liposomalen Suspensionen veranschaulicht. Für NSE-Messungen der In-Plane-Membrandynamik wird der Leser auf spezielle Publikationen zur Grazing-Incidence-Neutronen-Spin-Echo-Spektroskopie (GINSES)55,56 und andere Studien von ausgerichteten multilamellaren Membranstapeln57,58,59,60verwiesen.
Der Einfachheit halber hebt dieser Artikel drei verschiedene Schemata der Membrandeuteration hervor, die auf einem gut untersuchten domänenbildenden oder phasenabtrennenden Lipiddoppelschichtsystem aus 1,2-Dimyristoyl-sn-glycero-3-phosphocholin (DMPC) und 1,2-Distearoyl-sn-glycero-3-phosphocholin (DSPC) -Mischungen61,62dargestellt werden. Die beiden Lipide zeichnen sich durch eine Diskrepanz in ihrer Kohlenwasserstoffkettenlänge (14 Kohlenstoffe / Schwanz in DMPC vs. 18 Kohlenstoffe / Schwanz in DSPC) und ihrer Gel-Fluid-Übergangstemperatur (Tm, DMPC = 23 ° C vs Tm, DSPC = 55 ° C) aus. Dies führt zu einer lateralen Phasentrennung in DMPC:DSPC-Membranen bei Temperaturen zwischen der oberen und unteren Übergangstemperatur des Gemisches63. Die hier betrachteten Deuterationsschemata werden ausgewählt, um die verschiedenen dynamischen Modi zu demonstrieren, die bei NSE-Messungen auf liposomalen Membranen verfügbar sind, nämlich Biegefluktuationen, Dickenschwankungen und selektive Biege-/Dickenschwankungen von lateralen Domänen. Alle Lipidzusammensetzungen werden für DMPC:DSPC-Doppelschichten berichtet, die mit einem Molanteil von 70:30 unter Verwendung handelsüblich erhältlicher protiated und perdeuterated Varianten von DMPC und DSPC hergestellt wurden. Alle Probenvorbereitungsschritte basieren auf 4 ml liposomaler Suspension inD2O mit einer Lipidkonzentration von 50 mg/ml für eine Gesamtlipidmasse von Mtot = 200 mg pro Probe.
Protocol
Representative Results
Discussion
NSE ist eine leistungsstarke und einzigartige Technik zur Messung der mesoskopischen Dynamik von Lipidmembranen unter verschiedenen Bedingungen. Die effektive Nutzung von NSE hängt von der Probenqualität, dem Neutronenkontrast und dem Bereich der zugänglichen Dynamik ab, die für eine bestimmte Probe untersucht werden kann. Daher sind mehrere kritische Schritte erforderlich, um erfolgreiche NSE-Experimente durchzuführen und qualitativ hochwertige Daten zu sammeln. Ein wichtiger Schritt, um die effektive Nutzung der N…
Disclosures
The authors have nothing to disclose.
Acknowledgements
R. Ashkar dankt M. Nagao, L.-R. Stingaciu und P. Zolnierczuk für viele nützliche Diskussionen und für ihre häufige Unterstützung bei NSE-Experimenten auf ihren jeweiligen Beamlines. Die Autoren erkennen den Einsatz von Neutronenspin-Echospektrometern am NIST und ORNL an. Das NSE-Spektrometer am NIST wird vom Center for High Resolution Neutron Scattering unterstützt, einer Partnerschaft zwischen dem National Institute of Standards and Technology und der National Science Foundation unter der Vereinbarung Nr. DMR-1508249. Das NSE-Spektrometer an der Spallations-Neutronenquelle des ORNL wird von der Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy, unterstützt. Das Oak Ridge National Laboratory wird von UT-Battelle, LLC unter der US DOE Contract No. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

