Lipid Membran Dinamiği ve Membran-Protein Etkileşimleri için Benzersiz Bir Prob Olarak Nötron Spin Eko Spektroskopisi
Summary
Bu makalede lipid membranlarının nötron spin eko (NSE) çalışmalarında örnek hazırlama, veri azaltma ve veri analizi protokolleri açıklanmaktadır. Lipitlerin judicious döteryum etiketlemesi, üzerinde hayati biyolojik süreçlerin meydana geldiği mezoskopik uzunluk ve zaman ölçeklerinde farklı membran dinamiklerine erişim sağlar.
Abstract
Lipid bilayerler hücre zarlarının ana matrisini oluşturur ve diğer hayati hücresel süreçlerin yanı sıra besin değişimi, protein-membran etkileşimleri ve viral tomurcuklanma için birincil platformdur. Verimli biyolojik aktivite için, hücre zarları hücrenin ve bölmelerinin bütünlüğünü koruyacak kadar sert, ancak proteinler ve fonksiyonel alanlar gibi membran bileşenlerinin yayılmasına ve etkileşime girmesine izin edecek kadar akışkan olmalıdır. Elastik ve akışkan membran özelliklerinin bu hassas dengesi ve biyolojik işlev üzerindeki etkileri, mezoskopik uzunluk ve zaman ölçekleri üzerinde kolektif membran dinamiklerinin daha iyi anlaşılmasıni gerektirir, örneğin, membran deformasyonları ve protein bağlama olayları. Bu dinamik aralığı etkili bir şekilde araştırabilecek teknikler arasında nötron spin ekosu (NSE) spektroskopisi vardır. Döteryum etiketleme ile birlikte NSE, bükme ve kalınlık dalgalanmalarının yanı sıra seçilen membran özelliklerinin mezoskopik dinamiklerine doğrudan erişmek için kullanılabilir. Bu makale, NSE tekniğinin kısa bir açıklamasını sağlar ve veri toplama ve azaltma talimatlarının yanı sıra örnek hazırlama ve döterasyon şemalarının ayrıntıları da dahil olmak üzere lipozomal membranlar üzerinde NSE deneyleri gerçekleştirme prosedürlerini özetlemektedir. Makale ayrıca bükme sertlik modülü, alan sıkıştırılabilirlik modülü ve düzlem içi viskozite gibi anahtar membran parametrelerini çıkarmak için kullanılan veri analizi yöntemlerini de tanıtır. NSE çalışmalarının biyolojik önemini göstermek için, NSE tarafından araştırılan membran fenomenlerinin seçilmiş örnekleri, yani katkı maddelerinin membran bükme sertliği üzerindeki etkisi, etki alanı oluşumunun membran dalgalanmaları üzerindeki etkisi ve membran-protein etkileşimlerinin dinamik imzası tartışılmaktadır.
Introduction
Hücre zarlarının ve işlevlerinin anlaşılması son birkaç on yılda önemli ölçüde gelişti. Hücre zarlarının hücre sınırlarını tanımlayan pasif lipid bilayerleri olarak eski görüşü ve membran proteinleri1, lipid bilayerlerinin hücresel sinyalizasyon, moleküler değişim ve protein fonksiyonu da dahil olmak üzere hayati biyolojik süreçleri düzenlemede önemli bir rol oynadığı dinamik bir modele dönüştü – birkaç 2,3,4,5,6. Hücre zarlarının son derece dinamik olduğunu, sürekli yeniden şekillendirme ve moleküler yeniden dağıtımdan geçtiğinin farkına varılması, membranların denge yapılarının ötesinde bilimsel keşifler çağrısında bulunmaktadır7,8,9. Buna göre, biyolojik ve biyoinspired lipid membranlarındaki çeşitli dinamik modları incelemek için birden fazla yaklaşım geliştirilmiştir. Bugüne kadar, bu çalışmaların çoğunluğu öncelikle difüzif moleküler hareketler10 , 11,12,13 ve makroskopik şekil dalgalanmaları14,15,16, ara membran dinamiklerini anlamada önemli bir boşluk bırakarak, yani birkaç 10-100’lük lipid moleküllerinden oluşan lipid montajlarının kolektif dalgalanmalarına odaklanmıştır. Bu dinamikler, birkaç on ila birkaç 100 Å’nın uzunluk ölçekleri ve zaman içinde alt ns’den birkaç yüz ns’ye kadar olan ölçekler üzerinde meydana gelir (bkz. Şekil 1),burada mezoskopik ölçekler olarak adlandırılır. Gerçekten de bu ölçeklerde anahtar biyolojik aktivite membran seviyesinde gerçekleşir17. Bu viral tomurcuklanmaiçerir 18, kanal gating19, ve membran-protein etkileşimleri20. Ayrıca, membran proteinleri 21,22’ninenerji manzarasının, proteinlerdeki konformasyonel değişikliklerin – düzenleyici rolleri için gerekli – kolektif membran dalgalanmalarının 23’lüğünde ns zaman ölçeklerinde gerçekleştiğini gösterdiğini belirtmek önemlidir, ayrıca mezoskopik dinamiklerin hücre zarlarının biyolojik işlevindeki önemini ve biyoinspired analoglarını20. Bu makale lipid zarlarındaki iki birincil mezoskopik dinamik moda, yani bükülme dalgalanmalarına ve kalınlık dalgalanmalarına odaklanmaktadır.
Bu dalgalanma modlarını doğrudan yoklamadaki temel zorluk, standart spektroskopi yöntemlerini kullanarak mekansal ve zamansal ölçeklerine aynı anda erişmenin zorluğudur. Diğer zorluk, doğrudan temas tekniklerinin16’yıölçmek için amaçladıkları dalgalanmaları etkileyebileceğidir. Bu, biyolojik membranların bileşimsel ve yapısal karmaşıklığı ile daha da şiddetlenir24,25, lipid etki alanı oluşumu26 , 27,28,29,30ve membran asimetrisi31 ,32,33dahil olmak üzere homojen olmayan membran özellikleri ile sonuçlanır– farklı membran özelliklerinin dinamiklerini anlamak için seçici problar talep eder. Neyse ki, bu zorluklar, doğal olarak gerekli uzunluk ve zaman ölçeklerine erişen nötron spin ekosu (NSE) gibi invaziv olmayan nötron spektroskopisi yöntemleriyle aşılabilir ve fizikokimyasal ortamlarını değiştirmeden seçici membran özelliklerinin çalışmalarını daha da mümkün kılar34. Gerçekten de, son birkaç yılda NSE spektroskopisi kolektif membran dinamiklerinin benzersiz ve güçlü bir probuna dönüştü35. Lipid membranları üzerinde yapılan NSE çalışmalarından elde edilensonuçlar,lipid membranlarının mekanik36,37 ve viskoelastik38,39 özellikleri hakkında yeni içgörüler üretmiş ve biyolojik işlevdeki potansiyel rollerine yeni bir ışık tutmuş40,41.
NSE spektroskopi tekniği, ilk olarak Mezei42tarafından önerilen bir interferometrik enstrüman tasarımına dayanmaktadır Nötronlar enstrümandan geçerken nötron dönüşünün presesyonunu kontrol etmek için bir dizi spin-flipper ve manyetik bobin kullanarak. Tasarım, manyetik alan elemanlarının örnek konumuna göre manyetik olarak yansıtılmasına dayanır (Şekil 1A). Bu, nötron ve numune arasındaki enerji değişiminin yokluğunda, nötronun cihazın ilk ve ikinci yarısında ters yönlerde aynı sayıda spin precession gerçekleştirdiğini ima eder (iki pressyon bobini arasındaki π-palete dikkat edin). Sonuç olarak, nötronun son spin durumu ilk duruma göre değişmeden kalır – spin-echo olarak adlandırılan bir fenomen (Şekil 1A’dakişeffaf nötron’a bakın). Bununla birlikte, nötron numuneyle enerjik bir şekilde etkileşime girdiğinde, enerji değişimi cihazın ikinci yarısındaki spin presesyonlarının sayısını değiştirerek farklı bir son spin durumuna yol sağlar (bkz. Şekil 1A). Bu deneysel olarak kutuplaşmada bir kayıp olarak tespit edilir, bu makalede daha sonra gösterileceği gibi. NSE tekniği hakkında daha fazla bilgi için, okuyucu özel teknik makalelere atıfta bulunulmaktadır42,43,44,45.
Burada, NSE ile erişilebilen uzunluk ve zaman ölçeklerinin kabaca bir tahminini sağlamak için basitleştirilmiş bir açıklama sunulmaktadır. Uzunluk ölçekleri ulaşılabilir dalgacı transferleri aralığına göre belirlenir, Q = 4π sin φ/φ, burada 2φ saçılma açısıdır ve φ nötron dalga boyudur. Q’nun dalga boyu aralığı ve spektrometrenin ikinci kolunun dönüş derecesine göre ayarlandıklarından biri görülebilir (bkz. Şekil 1A). NSE spektrometrelerinde tipik bir Qaralığı ~0.02-2 ş-146,47ve 0.01-4 ş-1’e kadardır ve son yükseltmelerle 48,49, uzaysal ölçeklere karşılık gelen ~1-600 Å’dır. Öte yandan, erişilebilir zaman ölçeği, nötron tarafından manyetik pressyon bobinleri içinde elde edilen toplam presezyon açısından (veya fazdan) hesaplanır ve50: 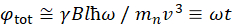 . Bu ifadede, t olarak tanımlanan Fourier
. Bu ifadede, t olarak tanımlanan Fourier 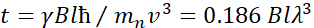
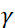 zamanıdır nötron nötron jinekagnetik oranı
zamanıdır nötron nötron jinekagnetik oranı 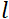 nerededir, bobin uzunluğudur ve
nerededir, bobin uzunluğudur ve 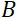 bobinin manyetik alanının gücüdür. Fourier zamanının kesinlikle enstrüman geometrisine, manyetik alan gücüne ve nötron dalga boyuna bağlı bir miktar olduğunu belirtmek gerekir. Örneğin, dalga boyu nötronları =
bobinin manyetik alanının gücüdür. Fourier zamanının kesinlikle enstrüman geometrisine, manyetik alan gücüne ve nötron dalga boyuna bağlı bir miktar olduğunu belirtmek gerekir. Örneğin, dalga boyu nötronları = 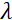 8 şve
8 şve 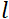 = 1,2 m ve
= 1,2 m ve 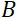 = 0,4 T’lik enstrüman ayarları kullanılarak, Fourier süresi t ~ 50 ns olarak hesaplanır. Deneysel olarak, Fourier zamanı, presesyon bobinlerindeki akımı değiştirerek (yani manyetik alan gücü) veya farklı nötron dalga boyları kullanılarak ayarlanır ve bu da ~ 1 ps ila 100 ns tipik NSE zaman ölçekleriyle sonuçlanır. Bununla birlikte, NSE spektrometrelerindeki son yükseltmeler daha uzun Fourier zamanlarına erişim sağladı, Heinz Maier-Leibnitz Zentrum51’deki J-NSE-Phoenix spektrometresinde ~400 ns’ye ve Oak Ridge National Lab 48’deki SNS-NSE spektrometresinde~400ns’ye kadar ve Institut Laue-Langevin (ILL) 49’daki IN15 NSE spektrometresinde~1.000ns’ye kadar.
= 0,4 T’lik enstrüman ayarları kullanılarak, Fourier süresi t ~ 50 ns olarak hesaplanır. Deneysel olarak, Fourier zamanı, presesyon bobinlerindeki akımı değiştirerek (yani manyetik alan gücü) veya farklı nötron dalga boyları kullanılarak ayarlanır ve bu da ~ 1 ps ila 100 ns tipik NSE zaman ölçekleriyle sonuçlanır. Bununla birlikte, NSE spektrometrelerindeki son yükseltmeler daha uzun Fourier zamanlarına erişim sağladı, Heinz Maier-Leibnitz Zentrum51’deki J-NSE-Phoenix spektrometresinde ~400 ns’ye ve Oak Ridge National Lab 48’deki SNS-NSE spektrometresinde~400ns’ye kadar ve Institut Laue-Langevin (ILL) 49’daki IN15 NSE spektrometresinde~1.000ns’ye kadar.
Membran dinamiklerinin uzunluk ve zaman ölçeğine doğrudan erişimin yanı sıra, NSE nötron izotop duyarlılığının doğal yeteneklerine sahiptir52. Özellikle, nötronların biyolojik sistemlerdeki en bol element olan hidrojenin izotoplarıyla farklı etkileşime girebilmeleri, protium döteryum ile değiştirildiğinde farklı bir nötron saçılma uzunluğu yoğunluğu,34 veya NSLD (kırılma50optik indeksine eşdeğer) ile sonuçlanır. Bu, genellikle belirli membran özelliklerini vurgulamak veya başkalarını gizlemek için kullanılan kontrast varyasyonu olarak bilinen bir yaklaşım sağlar — ikinci senaryoya kontrast eşleştirmesi denir. Kontrast değişiminin/eşleşmesinin sık uygulanması, suyun (NSLD = -0,56 × 10-6 ş-2)ağır su veya D 2 O (NSLD = 6) iledeğiştirilmesidir.4 × 10-6 ş-2) nötron sinyalini protiated lipid membranlarından yükseltmek için (NSLD ~ 0 × 10-6 ş-2). Bu yaklaşım membran yapısının çalışmalarında oldukça etkilidir, çünkü D2O’nun membranın kafa grubu bölgesine nüfuz etmesi, membran kalınlıklarının doğru bir şekilde belirlenmesine izin verir (bkz. Şekil 2A, sol panel) ve daha sofistike modeller uygulandığında farklı lipit alt gruplarının konumu53,54. Bu makalede, biyomimetik membranlarda kolektif dinamiklerin ve seçilmiş membran özelliklerinin çalışmaları için kontrast varyasyonunun kullanımına ilişkin bazı örnekler vurgulanmıştır.
Burada, NSE’nin dinamik ve fonksiyonel membran özelliklerine benzersiz içgörüler sağlamadaki etkinliği, NSE’nin model ve biyolojik olarak ilgili lipid membran sistemleri üzerinde yaptığı çalışmaların somut örnekleriyle, serbest duran membranlarda mezoscale dinamiklerine vurgu yaparak, lipozomal süspansiyonlar şeklinde gösterilmiştir. Düzlem içi membran dinamiklerinin NSE ölçümleri için okuyucu, otlatma-insidans nötron spin-echo spektroskopisi (GINSES)55,56 ve hizalanmış çok mller membran yığınlarının57 , 58,59,60ile ilgili diğer çalışmaları hakkında özel yayınlara atıfta bulunulmaktadır.
Basitlik için, bu makale iyi çalışılmış bir alan oluşturmada gösterilen üç farklı membran deuterasyon şemasını vurgulamaktadır, veya faz ayırma, lipid bilayer sistemi 1,2-dimyristoyl-sn-glisero-3-fosfokolin (DMPC) ve 1,2-distearoyl-sn-glisero-3-fosfokolin (DSPC) karışımları61,62. İki lipit, hidrokarbon zincir uzunluklarında (DMPC’de 14 karbon/kuyruk ve DSPC’de 18 karbon/kuyruk) ve jel-sıvı geçiş sıcaklıklarında (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C) bir uyumsuzluk ile karakterize edilir. Bu, DMPC:DSPC membranlarında karışımın üst ve alt geçiş sıcaklıkları arasındaki sıcaklıklarda yanal faz ayrımı ile sonuçlanır63. Burada göz önünde bulundurulan döterasyon şemaları, lipozomal membranlarda NSE ölçümlerinde erişilebilen farklı dinamik modları, yani bükme dalgalanmalarını, kalınlık dalgalanmalarını ve yanal etki alanlarının seçici bükme/kalınlık dalgalanmalarını göstermek için seçilir. Tüm lipid bileşimleri, DMPC ve DSPC’nin ticari olarak mevcut protiated ve perdeuterated varyantları kullanılarak 70:30 köstebek fraksiyonu ile hazırlanan DMPC:DSPC bilayerleri için rapor edilir. Tüm numune hazırlama adımları, D2O’da, 50 mg / mL lipit konsantrasyonuna sahip 4 mL lipozomal süspansiyona dayanmaktadır, toplam lipid kütlesi için Mtot = örnek başına 200 mg.
Protocol
Representative Results
Discussion
NSE, lipid zarlarının mezoskopik dinamiklerinin çeşitli koşullar altında ölçülmesinde güçlü ve benzersiz bir tekniktir. NSE’nin etkin kullanımı numune kalitesine, nötron kontrastı ve belirli bir örnek için araştırılabilen erişilebilir dinamiklerin aralığına bağlıdır. Bu nedenle, başarılı NSE denemeleri gerçekleştirmek ve yüksek kaliteli veri toplamak için birkaç kritik adım gereklidir. Bir NSE deneyi sırasında nötron ışın süresinin etkili kullanımını sağlamanın önemli bir…
Disclosures
The authors have nothing to disclose.
Acknowledgements
R. Ashkar, M.Nagao’ya teşekkür ediyor, L.-R. Stingaciu ve P. Zolnierczuk birçok yararlı tartışma ve kendi kiriş hatlarında NSE deneyleri ile sık sık yardım için. Yazarlar NIST ve ORNL’de nötron spin eko spektrometrelerinin kullanıldığını kabul eder. NIST’deki NSE spektrometresi, Ulusal Standartlar ve Teknoloji Enstitüsü ile Ulusal Bilim Vakfı arasında anlaşma no. DMR-1508249. ORNL’nin Spallation Nötron Kaynağı’ndaki NSE spektrometresi, ABD Enerji Bakanlığı Temel Enerji Bilimleri Ofisi Bilimsel Kullanıcı Tesisleri Bölümü tarafından desteklenmektedir. Oak Ridge Ulusal Laboratuvarı, ABD DOE Sözleşme No. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

