Espectroscopia de Eco de Rotação de Nêutrons como uma sonda única para dinâmicas de membrana lipídica e interações membrana-proteína
Summary
Este artigo descreve os protocolos de preparação de amostras, redução de dados e análise de dados em estudos de eco de giro de nêutrons (NSE) de membranas lipídicas. A rotulagem criteriosa de deutério de lipídios permite o acesso a diferentes dinâmicas de membrana em comprimento mesoscópico e escalas de tempo, sobre as quais ocorrem processos biológicos vitais.
Abstract
Bicamadas lipídicas formam a principal matriz das membranas celulares e são a principal plataforma de troca de nutrientes, interações proteína-membrana e brotamento viral, entre outros processos celulares vitais. Para uma atividade biológica eficiente, as membranas celulares devem ser rígidas o suficiente para manter a integridade da célula e seus compartimentos, mas fluidas o suficiente para permitir que componentes de membrana, como proteínas e domínios funcionais, difundam e interajam. Este delicado equilíbrio de propriedades de membrana elástica e fluida, e seu impacto na função biológica, necessitam de uma melhor compreensão da dinâmica da membrana coletiva sobre o comprimento mesoscópico e escalas de tempo dos principais processos biológicos, por exemplo, deformações de membrana e eventos de ligação de proteínas. Entre as técnicas que podem efetivamente sondar esse alcance dinâmico está a espectroscopia de nêutrons spin echo (NSE). Combinado com a rotulagem de deutério, o NSE pode ser usado para acessar diretamente flutuações de dobra e espessura, bem como dinâmica mesoscópica de características de membrana selecionadas. Este artigo fornece uma breve descrição da técnica NSE e descreve os procedimentos para a realização de experimentos NSE em membranas liposômicas, incluindo detalhes de esquemas de preparação e deuteração da amostra, juntamente com instruções para coleta e redução de dados. O artigo também introduz métodos de análise de dados usados para extrair parâmetros de membrana chave, como o módulo de rigidez de dobra, o módulo de compressão da área e a viscosidade no plano. Para ilustrar a importância biológica dos estudos da NSE, são discutidos exemplos selecionados de fenômenos de membrana sondados pela NSE, ou seja, o efeito dos aditivos sobre a rigidez de dobra de membrana, o impacto da formação de domínios sobre as flutuações da membrana e a assinatura dinâmica das interações membrana-proteína.
Introduction
A compreensão das membranas celulares e sua função evoluiu notavelmente nas últimas décadas. A visão anterior das membranas celulares como bicamadas lipídicas passivas que definem limites celulares e proteínas de membranadoméstica 1 gradualmente se transformou em um modelo dinâmico no qual bicamadas lipídicas desempenham um papel importante na regulação de processos biológicos vitais, incluindo sinalização celular, troca molecular e função proteica — para citar alguns2,3,4,5,6. Essa constatação de que as membranas celulares são altamente dinâmicas, constantemente submetidas à remodelação e redistribuição molecular, tem incitado explorações científicas além das estruturas de equilíbrio das membranas7,8,9. Assim, várias abordagens foram desenvolvidas para estudar os diversos modos dinâmicos em membranas lipídicas biológicas e bioinspiradas. Até o momento, a maioria desses estudos se concentrou principalmente nos movimentos moleculares difusivos10,11,12,13 e flutuações de forma macroscópica14,15,16, deixando uma lacuna significativa na compreensão da dinâmica da membrana intermediária, ou seja, flutuações coletivas de conjuntos lipídicos consistindo de poucos 10-100s de moléculas lipídicas. Essas dinâmicas ocorrem ao longo de escalas de comprimento de poucas dezenas a poucos 100 Å e ao longo do tempo escalas de sub-ns a algumas centenas de ns (ver Figura 1), referidas aqui como escalas mesoscópicas. É nestas escalas que a atividade biológica chave ocorre no nível de membrana17. Isso inclui o brotamento viral18, gating canal19e interações membrana-proteína20. Também é importante ressaltar que a paisagem energética das proteínas da membrana21,22 mostra que as alterações conformais nas proteínas — necessárias para seu papel regulatório — acontecem ao longo das escalas de tempons 23 de flutuações coletivas de membrana, enfatizando ainda mais a importância da dinâmica mesoscópica na função biológica das membranas celulares e seus analógicos bioinspirados20. Este artigo se concentra nos dois modos dinâmicos mesoscópicos primários em membranas lipídicas, ou seja, flutuações de dobra e flutuações de espessura.
O principal desafio em sondar diretamente esses modos de flutuação é a dificuldade em acessar simultaneamente suas escalas espaciais e temporais usando métodos de espectroscopia padrão. O outro desafio é que as técnicas de contato direto podem impactar as mesmas flutuações que se destinam a medir16. Isso é ainda mais exacerbado pela complexidade composicional e estrutural das membranas biológicas24,25, o que resulta em características de membrana não homogênea, incluindo a formação de domínio lipídudo26,27,28,29,30 e assimetria de membrana 31,32,33– exigindo sondas seletivas para entender a dinâmica de diferentes características da membrana. Felizmente, esses desafios podem ser superados com métodos não invasivos de espectroscopia de nêutrons, como o echo de giro de nêutrons (NSE), que acessam inerentemente o comprimento e as escalas de tempo necessárias, e permitem ainda estudos de características de membrana seletiva sem alterar seu ambiente físico-químico34. De fato, nos últimos anos a espectroscopia NSE evoluiu para uma sonda única e poderosa da dinâmica da membrana coletiva35. Os resultados de estudos da NSE sobre membranas lipídicas produziram novos insights sobre as propriedades mecânica36,37 e viscoelástica38,39 de membranas lipídicas e lançaram uma nova luz sobre seu potencial papel na função biológica40,41.
A técnica de espectroscopia NSE é baseada em um design de instrumento interferométrico, proposto pela primeira vez por Mezei42, usando uma série de giradoras e bobinas magnéticas para controlar a precessão do giro de nêutrons à medida que os nêutrons atravessam o instrumento. O desenho repousa no espelhamento magnético dos elementos do campo magnético em relação à posição da amostra(Figura 1A). Isso implica que, na ausência de troca de energia entre o nêutron e a amostra, o nêutron realiza o mesmo número de precessões de spin, em direções opostas, na primeira e segunda metade do instrumento (observe o π-flipper entre as duas bobinas de pré-cessão). Como resultado, o estado final de rotação do nêutron permanece inalterado em relação ao estado inicial – um fenômeno referido como spin-echo (ver nêutron transparente na Figura 1A). No entanto, quando o nêutron interage energeticamente com a amostra, a troca de energia modifica o número de precessões de spin na segunda metade do instrumento, levando a um estado de rotação final diferente (ver Figura 1A). Isso é detectado experimentalmente como uma perda de polarização, como será mostrado mais tarde neste artigo. Para mais detalhes sobre a técnica NSE, o leitor é encaminhado para documentos técnicos dedicados42,43,44,45.
Aqui, uma descrição simplificada é apresentada para fornecer uma estimativa aproximada do comprimento e escalas de tempo acessíveis com a NSE. As escalas de comprimento são determinadas pela faixa de transferências de vetores de onda alcançáveis, Q = 4π sin φ/λ, onde 2φ é o ângulo de dispersão e λ é o comprimento de onda de nêutrons. Pode-se ver que Q é definido pela faixa de comprimento de onda e a extensão da rotação do segundo braço do espectrômetro (ver Figura 1A). Uma faixa típica de Qnos espectrômetros NSE é ~0,02-2 Å-146,47, e até 0,01-4 Å-1 com atualizações recentes48,49, correspondentes a escalas espaciais de ~1-600 Å. Por outro lado, a escala de tempo acessível é calculada a partir do ângulo total de precessão (ou fase) adquirido pelo nêutron dentro das bobinas de precessão magnética, e é encontrado como50: 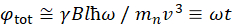 . Nesta expressão, t é o tempo de Fourier definido como
. Nesta expressão, t é o tempo de Fourier definido como 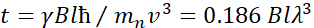 , onde está a razão
, onde está a razão 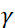 gyrommagnética de nêutrons,
gyrommagnética de nêutrons, 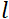 é o comprimento da bobina, e
é o comprimento da bobina, e 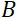 é a força do campo magnético da bobina. Vale ressaltar que o tempo de Fourier é uma quantidade estritamente dependente da geometria do instrumento, força do campo magnético e comprimento de onda de nêutrons. Por exemplo, usando nêutrons de comprimento de onda
é a força do campo magnético da bobina. Vale ressaltar que o tempo de Fourier é uma quantidade estritamente dependente da geometria do instrumento, força do campo magnético e comprimento de onda de nêutrons. Por exemplo, usando nêutrons de comprimento de onda 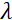 = 8 Å e configurações de instrumentos de
= 8 Å e configurações de instrumentos de 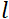 = 1,2 m e
= 1,2 m e 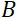 = 0,4 T, o tempo de Fourier é calculado para ser t ~ 50 ns. Experimentalmente, o tempo de Fourier é sintonizado alterando a corrente nas bobinas de precessão (ou seja, força do campo magnético) ou usando diferentes comprimentos de onda de nêutrons, resultando em escalas típicas de tempo NSE de ~ 1 ps a 100 ns. No entanto, os upgrades recentes nos espectrômetros NSE permitiram o acesso a tempos mais longos do Fourier, até ~400 ns no espectrômetro J-NSE-Phoenix no espectrômetro Heinz Maier-Leibnitz Zentrum51 e no espectrômetro SNS-NSE no Oak Ridge National Lab48, e até ~1.000 ns no espectrômetro IN15 NSE no Institut Laue-Langevin (III)49.
= 0,4 T, o tempo de Fourier é calculado para ser t ~ 50 ns. Experimentalmente, o tempo de Fourier é sintonizado alterando a corrente nas bobinas de precessão (ou seja, força do campo magnético) ou usando diferentes comprimentos de onda de nêutrons, resultando em escalas típicas de tempo NSE de ~ 1 ps a 100 ns. No entanto, os upgrades recentes nos espectrômetros NSE permitiram o acesso a tempos mais longos do Fourier, até ~400 ns no espectrômetro J-NSE-Phoenix no espectrômetro Heinz Maier-Leibnitz Zentrum51 e no espectrômetro SNS-NSE no Oak Ridge National Lab48, e até ~1.000 ns no espectrômetro IN15 NSE no Institut Laue-Langevin (III)49.
Além do acesso direto ao comprimento e escala de tempo da dinâmica da membrana, a NSE possui as capacidades inerentes à sensibilidade ao isótopo de nêutrons52. Especificamente, a capacidade dos nêutrons de interagir de forma diferente com os isótopos do hidrogênio, o elemento mais abundante em sistemas biológicos, resulta em uma densidade de comprimento de dispersão de nêutrons diferente,34 ou NSLD (o equivalente ao índice óptico de refração50), quando o prótium é substituído por deutério. Isso permite uma abordagem conhecida como variação de contraste, que é comumente usada para destacar características específicas da membrana ou ocultar outras — este último cenário é referido como correspondência de contraste. Uma aplicação frequente de variação/correspondência de contraste é a substituição de água (NSLD = -0,56 × 10-6 Å-2) por água pesada ou D2O (NSLD = 6,6 4 × 10-6 Å-2) para amplificar o sinal de nêutrons de membranas lipídicas protiadas (NSLD ~ 0 × 10-6 Å-2). Essa abordagem é altamente eficaz em estudos de estrutura de membrana porque a penetração de D2O na região do headgroup da membrana permite a determinação precisa das espessuras da membrana (ver Figura 2A, painel esquerdo) e da localização de diferentes subgrupos lipídes quando modelos mais sofisticados são aplicados53,54. Este artigo destaca alguns exemplos sobre o uso de variação de contraste para estudos de dinâmica coletiva em membranas biomiméticas e características de membrana selecionadas.
Aqui, a eficácia da NSE em fornecer insights únicos sobre propriedades de membrana dinâmica e funcional é ilustrada através de exemplos tangíveis de estudos NSE sobre sistemas de membrana lipídica modelo e biologicamente relevantes com ênfase na dinâmica da mesoescala em membranas autônomas, na forma de suspensões liposômicas. Para as medições NSE da dinâmica da membrana in-plane, o leitor é encaminhado para publicações dedicadas sobre espectroscopia de spin-echo de nêutrons de incidência de pastagem (GINSES)55,56 e outros estudos de pilhas de membrana multilamelaralinhadas 57,58,59,60.
Para simplificar, este artigo destaca três diferentes esquemas de deuteração de membrana ilustrados em uma formação de domínio bem estudada, ou separação de fase, sistema de bicamadas lipídicas de misturas 1,2-dimyristoyl-sn-glycero-3-fosphocholina (DMPC) e 1,2-distearoyl-sn-glycero-3-phosphocholine (DSPC) misturas61,62. Os dois lipídios são caracterizados por uma incompatibilidade em seu comprimento da cadeia de hidrocarbonetos (14 carbonos/cauda em DMPC vs 18 carbonos/cauda em DSPC) e sua temperatura de transição gel-fluido (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Isso resulta na separação lateral de fases nas membranas DMPC:DSPC a temperaturas entre as temperaturas de transição superior e inferior da mistura63. Os esquemas de deuteração aqui considerados são escolhidos para demonstrar os diferentes modos dinâmicos acessíveis nas medidas NSE em membranas lipoesômicas, ou seja, flutuações de dobra, flutuações de espessura e flutuações seletivas de dobra/espessura dos domínios laterais. Todas as composições lipídicas são relatadas para bicamadas DMPC:DSPC preparadas a uma fração toupeira de 70:30, utilizando variantes protiadas e semeadas comercialmente disponíveis de DMPC e DSPC. Todas as etapas de preparação da amostra são baseadas em 4 mL de suspensão liposômica, em D2O, com concentração lipídica de 50 mg/mL, para uma massa lipídica total de Mtot = 200 mgs por amostra.
Protocol
Representative Results
Discussion
NSE é uma técnica poderosa e única na medição da dinâmica mesoscópica das membranas lipídicas sob várias condições. A utilização efetiva da NSE depende da qualidade da amostra, do contraste de nêutrons e da gama de dinâmicas acessíveis que podem ser sondadas para uma determinada amostra. Assim, várias etapas críticas são necessárias para a realização de experimentos NSE bem-sucedidos e a coleta de dados de alta qualidade. Um passo fundamental para garantir o uso efetivo do tempo de feixe de nêutron…
Disclosures
The authors have nothing to disclose.
Acknowledgements
R. Ashkar agradece M. Nagao, L.-R. Stingaciu e P. Zolnierczuk para muitas discussões úteis e por sua assistência frequente com experimentos NSE em suas respectivas linhas de trave. Os autores reconhecem o uso de espectrômetros de eco de spin de nêutrons no NIST e ORNL. O espectrômetro NSE no NIST é apoiado pelo Centro de Dispersão de Nêutrons de Alta Resolução, uma parceria entre o Instituto Nacional de Padrões e Tecnologia e a Fundação Nacional de Ciência sob o acordo nº. DMR-1508249. O espectrômetro NSE na Fonte de Nêutrons spallation da ORNL é apoiado pela Divisão de Instalações de Usuários Científicos, Escritório de Ciências Básicas de Energia, Departamento de Energia dos EUA. Oak Ridge National Laboratory é gerenciado pela UT-Battelle, LLC sob o contrato us doe no. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

