Spettroscopia eco-spin neutronica come sonda unica per la dinamica della membrana lipidica e le interazioni membrana-proteina
Summary
Questo documento descrive i protocolli per la preparazione dei campioni, la riduzione dei dati e l’analisi dei dati negli studi NSE (Neutron Spin Echo) delle membrane lipidiche. L’etichettatura giudiziosa dei lipidi al deuterio consente l’accesso a diverse dinamiche di membrana su scale di lunghezza e tempo mesoscopiche, su cui si verificano processi biologici vitali.
Abstract
I doppi strati lipidici formano la matrice principale delle membrane cellulari e sono la piattaforma primaria per lo scambio di nutrienti, le interazioni proteina-membrana e il germogliamento virale, tra gli altri processi cellulari vitali. Per un’attività biologica efficiente, le membrane cellulari dovrebbero essere abbastanza rigide da mantenere l’integrità della cellula e dei suoi compartimenti ma abbastanza fluide da consentire ai componenti della membrana, come proteine e domini funzionali, di diffondersi e interagire. Questo delicato equilibrio delle proprietà elastiche e fluide della membrana, e il loro impatto sulla funzione biologica, richiedono una migliore comprensione delle dinamiche collettive della membrana su scale mesoscopiche di lunghezza e tempo dei principali processi biologici, ad esempio deformazioni della membrana ed eventi di legame proteico. Tra le tecniche che possono sondare efficacemente questa gamma dinamica c’è la spettroscopia neutron spin echo (NSE). In combinazione con l’etichettatura al deuterio, nSE può essere utilizzato per accedere direttamente alle fluttuazioni di flessione e spessore, nonché alla dinamica mesoscopica di caratteristiche selezionate della membrana. Questo documento fornisce una breve descrizione della tecnica NSE e delinea le procedure per l’esecuzione di esperimenti NSE su membrane liposomiali, compresi i dettagli della preparazione del campione e degli schemi di deuterazione, insieme alle istruzioni per la raccolta e la riduzione dei dati. Il documento introduce anche metodi di analisi dei dati utilizzati per estrarre i parametri chiave della membrana, come il modulo di rigidità di flessione, il modulo di comprimibilità dell’area e la viscosità nel piano. Per illustrare l’importanza biologica degli studi NSE, vengono discussi esempi selezionati di fenomeni di membrana sondati da NSE, vale a dire, l’effetto degli additivi sulla rigidità di flessione della membrana, l’impatto della formazione del dominio sulle fluttuazioni della membrana e la firma dinamica delle interazioni membrana-proteina.
Introduction
La comprensione delle membrane cellulari e della loro funzione si è notevolmente evoluta negli ultimi decenni. La precedente visione delle membrane cellulari come doppi strati lipidici passivi che definiscono i confini cellulari e le proteine di membrana1 si è gradualmente trasformata in un modello dinamico in cui i doppi strati lipidici svolgono un ruolo importante nella regolazione dei processi biologici vitali, tra cui la segnalazione cellulare, lo scambio molecolare e la funzione proteica – per citarne alcuni2,3,4,5,6. Questa consapevolezza che le membrane cellulari sono altamente dinamiche, costantemente in fase di rimodellamento e ridistribuzione molecolare, ha sollecitato esplorazioni scientifiche oltre le strutture di equilibrio delle membrane7,8,9. Di conseguenza, sono stati sviluppati diversi approcci per studiare le varie modalità dinamiche nelle membrane lipidiche biologiche e bioispira. Ad oggi, la maggior parte di questi studi si è concentrata principalmente sui moti molecolari diffusivi10,11, 12,13 e sulle fluttuazioni macroscopiche della forma14,15,16,lasciando un vuoto significativo nella comprensione della dinamica intermedia della membrana, cioè fluttuazioni collettive di assemblaggi lipidici costituiti da pochi 10-100 di molecole lipidiche. Queste dinamiche si verificano su scale di lunghezza da poche decine a poche 100 Å e su scale temporali di sub-ns a poche centinaia di ns (vedi Figura 1), qui indicate come scale mesoscopiche. È infatti su queste scale che l’attività biologica chiave si svolge a livello di membrana17. Ciò include il germogliamento virale18,il gating del canale19e le interazioni membrana-proteina20. È anche importante sottolineare che il panorama energetico delle proteine di membrana21,22 mostra che i cambiamenti conformazionali nelle proteine – necessari per il loro ruolo regolatore – avvengono sulle scale temporali ns23 delle fluttuazioni collettive della membrana, sottolineando ulteriormente l’importanza della dinamica mesoscopica nella funzione biologica delle membrane cellulari e dei loro analoghi bioispirati20. Questo documento si concentra sulle due principali modalità dinamiche mesoscopiche nelle membrane lipidiche, vale a dire, fluttuazioni di flessione e fluttuazioni di spessore.
La sfida principale nel sondare direttamente queste modalità di fluttuazione è la difficoltà di accedere simultaneamente alle loro scale spaziali e temporali utilizzando metodi di spettroscopia standard. L’altra sfida è che le tecniche di contatto diretto potrebbero avere un impatto sulle stesse fluttuazioni che dovrebbero misurare16. Ciò è ulteriormente esacerbato dalla complessità composizionale e strutturale delle membrane biologiche24,25, che si traduce in caratteristiche di membrana non omogenee, tra cui la formazione del dominio lipidico26,27,28,29,30 e l’asimmetria di membrana31,32,33– che richiedono sonde selettive per comprendere la dinamica delle diverse caratteristiche della membrana. Fortunatamente, queste sfide possono essere superate con metodi di spettroscopia neutronica non invasivi, come l’eco di spin neutronico (NSE), che accedono intrinsecamente alle scale di lunghezza e tempo richieste e consentono ulteriormente studi sulle caratteristiche selettive della membrana senza modificare il loro ambiente fisico-chimico34. Infatti, negli ultimi anni la spettroscopia NSE si è evoluta in una sonda unica e potente di dinamica collettiva della membrana35. I risultati degli studi NSE sulle membrane lipidiche hanno prodotto nuove intuizioni sulle proprietà meccaniche36,37 e viscoelastiche38,39 delle membrane lipidiche e hanno gettato nuova luce sul loro potenziale ruolo nella funzione biologica40,41.
La tecnica di spettroscopia NSE si basa su un progetto di strumento interferometrico, proposto per la prima volta da Mezei42, utilizzando una serie di pinne di spin e bobine magnetiche per controllare la precessione dello spin neutronico mentre i neutroni attraversano lo strumento. Il design si basa sulla specchiatura magnetica degli elementi del campo magnetico rispetto alla posizione del campione (Figura 1A). Ciò implica che in assenza di scambio di energia tra il neutrone e il campione, il neutrone esegue lo stesso numero di precessione di spin, in direzioni opposte, nella prima e nella seconda metà dello strumento (si noti la π-flipper tra le due bobine di precessione). Di conseguenza, lo stato di spin finale del neutrone rimane invariato rispetto allo stato iniziale – un fenomeno denominato spin-eco (vedi neutrone trasparente in Figura 1A). Tuttavia, quando il neutrone interagisce energeticamente con il campione, lo scambio di energia modifica il numero di precessione di spin nella seconda metà dello strumento, portando ad un diverso stato di spin finale (vedi Figura 1A). Questo è sperimentalmente rilevato come una perdita di polarizzazione, come verrà mostrato più avanti in questo articolo. Per maggiori dettagli sulla tecnica NSE, si rimanda al lettore gli articoli tecnici dedicati42,43,44,45.
Qui viene presentata una descrizione semplificata per fornire una stima approssimativa della lunghezza e delle scale temporali accessibili con NSE. Le scale di lunghezza sono determinate dall’intervallo di trasferimenti di motovettori realizzabili, Q = 4π sin θ/λ, dove 2θ è l’angolo di diffusione e λ è la lunghezza d’onda del neutrone. Si può vedere che Q è impostato dall’intervallo di lunghezze d’onda e dall’estensione della rotazione del secondo braccio dello spettrometro (vedi Figura 1A). Un tipico intervallo Qsugli spettrometri NSE è ~ 0,02-2 Å-146,47e fino a 0,01-4 Å-1 con recenti aggiornamenti48,49, corrispondenti a scale spaziali di ~ 1-600 Å. D’altra parte, la scala temporale accessibile è calcolata dall’angolo di precessione totale (o fase) acquisito dal neutrone all’interno delle bobine di precessione magnetica, e si trova ad essere50: 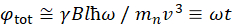 . In questa espressione, t è il tempo di Fourier definito come
. In questa espressione, t è il tempo di Fourier definito come 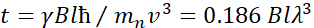 , dove è il rapporto
, dove è il rapporto 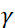 giromagnetico neutronico,
giromagnetico neutronico, 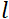 è la lunghezza della bobina e è la forza del campo
è la lunghezza della bobina e è la forza del campo 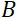 magnetico della bobina. Vale la pena sottolineare che il tempo di Fourier è una quantità che dipende strettamente dalla geometria dello strumento, dall’intensità del campo magnetico e dalla lunghezza d’onda dei neutroni. Ad esempio, usando neutroni di lunghezza d’onda
magnetico della bobina. Vale la pena sottolineare che il tempo di Fourier è una quantità che dipende strettamente dalla geometria dello strumento, dall’intensità del campo magnetico e dalla lunghezza d’onda dei neutroni. Ad esempio, usando neutroni di lunghezza d’onda 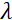 = 8 Å e impostazioni dello strumento di
= 8 Å e impostazioni dello strumento di 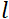 = 1,2 m e
= 1,2 m e 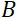 = 0,4 T, il tempo di Fourier è calcolato per essere t ~ 50 ns. Sperimentalmente, il tempo di Fourier viene regolato modificando la corrente nelle bobine di precessione (cioè l’intensità del campo magnetico) o utilizzando diverse lunghezze d’onda dei neutroni, con conseguente scala temporale tipica di NSE da ~ 1 ps a 100 ns. Tuttavia, i recenti aggiornamenti degli spettrometri NSE hanno consentito l’accesso a tempi di Fourier più lunghi, fino a ~ 400 ns sullo spettrometro J-NSE-Phoenix presso lo spettrometro Heinz Maier-Leibnitz Zentrum51 e lo spettrometro SNS-NSE presso l’Oak Ridge National Lab48e fino a ~ 1.000 ns presso lo spettrometro NSE IN15 presso l’Institut Laue-Langevin (ILL)49.
= 0,4 T, il tempo di Fourier è calcolato per essere t ~ 50 ns. Sperimentalmente, il tempo di Fourier viene regolato modificando la corrente nelle bobine di precessione (cioè l’intensità del campo magnetico) o utilizzando diverse lunghezze d’onda dei neutroni, con conseguente scala temporale tipica di NSE da ~ 1 ps a 100 ns. Tuttavia, i recenti aggiornamenti degli spettrometri NSE hanno consentito l’accesso a tempi di Fourier più lunghi, fino a ~ 400 ns sullo spettrometro J-NSE-Phoenix presso lo spettrometro Heinz Maier-Leibnitz Zentrum51 e lo spettrometro SNS-NSE presso l’Oak Ridge National Lab48e fino a ~ 1.000 ns presso lo spettrometro NSE IN15 presso l’Institut Laue-Langevin (ILL)49.
Oltre all’accesso diretto alla scala di lunghezza e tempo della dinamica di membrana, NSE ha le capacità intrinseche della sensibilità isotopica dei neutroni52. In particolare, la capacità dei neutroni di interagire in modo diverso con gli isotopi dell’idrogeno, l’elemento più abbondante nei sistemi biologici, si traduce in una diversa densità di lunghezza di scattering neutronico,34 o NSLD (l’equivalente dell’indice ottico di rifrazione50), quando il prozio è sostituito dal deuterio. Ciò consente un approccio noto come variazione di contrasto, che viene comunemente utilizzato per evidenziare specifiche caratteristiche della membrana o nascondene altre – quest’ultimo scenario è indicato come corrispondenza di contrasto. Un’applicazione frequente di variazione/corrispondenza del contrasto è la sostituzione dell’acqua (NSLD = -0,56 ×10 -6 Å -2) con acqua pesante o D2O (NSLD = 6,4 × 10-6 Å-2) per amplificare il segnale neutronico dalle membrane lipidiche protiate (NSLD ~ 0 × 10-6 Å-2). Questo approccio è altamente efficace negli studi sulla struttura della membrana perché la penetrazione di D2O nella regione del gruppo di testa della membrana consente una determinazione accurata degli spessori della membrana (vedi Figura 2A,pannello di sinistra) e della posizione di diversi sottogruppi lipidici quando vengono applicati modelli più sofisticati53,54. Questo documento evidenzia alcuni esempi sull’uso della variazione di contrasto per studi di dinamica collettiva in membrane biomimetiche e caratteristiche selezionate della membrana.
Qui, l’efficacia di NSE nel fornire approfondimenti unici sulle proprietà dinamiche e funzionali della membrana è illustrata attraverso esempi tangibili di studi NSE su sistemi di membrana lipidica modello e biologicamente rilevanti con particolare attenzione alla dinamica della mesoscala nelle membrane autoportanti, sotto forma di sospensioni liposomiali. Per le misure NSE della dinamica della membrana in-plane, il lettore è rimandato a pubblicazioni dedicate sulla spettroscopia spin-eco neutronica ad incidenza radente (GINSES)55,56 e altri studi su pile di membrane multilamellari allineate57,58,59,60.
Per semplicità, questo articolo evidenzia tre diversi schemi di deuterazione della membrana illustrati su un sistema a doppio strato lipidico ben studiato di formazione di dominio, o separazione di fase, di miscele di 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) e 1,2-distearoil-sn-glicero-3-fosfocolina (DSPC)61,62. I due lipidi sono caratterizzati da una discrepanza nella lunghezza della catena idrocarburica (14 atomi di carbonio/coda in DMPC vs 18 atomi di carbonio/coda in DSPC) e dalla loro temperatura di transizione gel-fluido (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Ciò si traduce in una separazione di fase laterale nelle membrane DMPC:DSPC a temperature comprese tra le temperature di transizione superiore e inferiore della miscela63. Gli schemi di deuterazione qui considerati sono scelti per dimostrare le diverse modalità dinamiche accessibili nelle misurazioni NSE su membrane liposomiali, vale a dire, fluttuazioni di flessione, fluttuazioni di spessore e fluttuazioni selettive di flessione / spessore di domini laterali. Tutte le composizioni lipidiche sono riportate per i doppi strati DMPC:DSPC preparati a una frazione molare di 70:30, utilizzando varianti proziate e perdeuterate disponibili in commercio di DMPC e DSPC. Tutte le fasi di preparazione del campione si basano su 4 mL di sospensione liposomiale, in D2O, con una concentrazione lipidica di 50 mg/mL, per una massa lipidica totale di Mtot = 200 mg per campione.
Protocol
Representative Results
Discussion
NSE è una tecnica potente e unica nella misurazione della dinamica mesoscopica delle membrane lipidiche in varie condizioni. L’utilizzo efficace di NSE dipende dalla qualità del campione, dal contrasto neutronico e dalla gamma di dinamiche accessibili che possono essere sonde per un determinato campione. Pertanto, sono necessari diversi passaggi critici per eseguire esperimenti NSE di successo e raccogliere dati di alta qualità. Un passo fondamentale per garantire l’uso efficace del tempo del fascio di neutroni durant…
Disclosures
The authors have nothing to disclose.
Acknowledgements
R. Ashkar ringrazia M. Nagao, L.-R. Stingaciu e P. Zolnierczuk per molte discussioni utili e per la loro frequente assistenza con esperimenti NSE sulle rispettive linee di fascio. Gli autori riconoscono l’uso di spettrometri eco-spin neutronici presso NIST e ORNL. Lo spettrometro NSE del NIST è supportato dal Center for High Resolution Neutron Scattering, una partnership tra il National Institute of Standards and Technology e la National Science Foundation sotto accordo n. DMR-1508249. Lo spettrometro NSE presso la sorgente di neutroni di spallazione dell’ORNL è supportato dalla Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy. Oak Ridge National Laboratory è gestito da UT-Battelle, LLC sotto il contratto DOE degli Stati Uniti n. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

