지질 막 역학 및 막 단백질 상호 작용을 위한 독특한 프로브로 중성자 스핀 에코 분광법
Summary
이 논문은 지질 막의 중성자 스핀 에코 (NSE) 연구에서 샘플 준비, 데이터 감소 및 데이터 분석을위한 프로토콜을 설명합니다. 지질의 현명한 중테리움 라벨링은 중요한 생물학적 과정이 발생하는 중전 길이와 시간 척도에 대한 다양한 막 역학에 접근할 수 있게 합니다.
Abstract
지질 이중층은 세포막의 주요 매트릭스를 형성하고 영양 교환, 단백질 막 상호 작용 및 바이러스 성 신진을위한 기본 플랫폼입니다, 다른 중요한 세포 과정 중. 효율적인 생물학적 활동을 위해 세포막은 세포와 구획의 무결성을 유지할 수 있을 만큼 단단해야 하지만 단백질 및 기능성 도메인과 같은 멤브레인 성분이 확산되고 상호 작용할 수 있을 만큼 충분히 유연해야 합니다. 탄성 및 유체 막 특성의 섬세한 균형, 생물학적 기능에 미치는 영향은 주요 생물학적 과정의 장편 길이 및 시간 척도,즉 막 변형 및 단백질 결합 이벤트의 집단 막 역학에 대한 더 나은 이해가 필요합니다. 이 동적 범위를 효과적으로 조사 할 수있는 기술 중 중 하나는 중성자 스핀 에코 (NSE) 분광법입니다. 중수소 라벨링과 결합된 NSE는 벤딩 및 두께 변동뿐만 아니라 선택 된 멤브레인 피처의 중전적 역학에 직접 액세스하는 데 사용할 수 있습니다. 이 백서는 NSE 기술에 대한 간략한 설명을 제공하고 샘플 준비 및 중음 체계의 세부 사항과 데이터 수집 및 감소에 대한 지침을 포함하여 리포소말 멤브레인에 대한 NSE 실험을 수행하는 절차를 간략하게 설명합니다. 또한 굴곡 강성 계수, 영역 압축성 계수 및 평면 점도와 같은 주요 멤브레인 파라미터를 추출하는 데 사용되는 데이터 분석 방법을 소개합니다. NSE 연구의 생물학적 중요성을 설명하기 위해 NSE가 조사한 멤브레인 현상의 예를 선택하여 멤브레인 굽힘 강성에 첨가제의 효과, 막 변동에 대한 도메인 형성의 영향 및 막 단백질 상호 작용의 동적 시그니처에 대해 논의됩니다.
Introduction
세포막과 그 기능에 대한 이해는 지난 수십 년 동안 현저하게 진화했습니다. 세포막의 이전 보기는 세포 경계와 집막단백질을 정의하는 수동 지질 양층으로 1은 점차 지질 이중층이 세포 신호, 분자 교환 및 단백질 기능을 포함한 중요한 생물학적 과정을 조절하는 데 중요한 역할을하는 동적 모델로 점차 변형되어 몇가지 2,3,4,5,6의이름을 지정합니다. 세포막이 매우 역동적이고 지속적으로 리모델링 및 분자 재분배를 겪고 있다는 이러한 실현은 멤브레인7,8,9의평형 구조를 넘어 과학 탐사를 촉구했습니다. 이에 따라 생물학적 및 생체 영감 지질 막에서 다양한 동적 모드를 연구하기 위해 여러 가지 접근법이 개발되었습니다. 현재까지, 이들 연구의 대부분은 주로 확산 분자 운동에 초점을 맞추고있다 10,11,12,13 및 거시적 모양 변동14,15,16,중간 막 역학이해에 상당한 격차를 떠나, 즉, 지질 분자의 몇 10-100s로 구성 지질 어셈블리의 집단 변동. 이러한 역학은 수십 ~소수의 100 Å의 길이 저울과 몇 백 ns (그림 1참조)에 하위 ns의 시간 동안 저울에 걸쳐 발생, 중시경 비늘로 여기에 언급. 그것은 실제로 주요 생물학적 활동이 막 수준17에서일어나는 이 비늘에 실제로 입니다. 여기에는 바이러스신진(18),채널 게이팅19,막 단백질 상호작용(20)이포함됩니다. 또한 멤브레인 단백질21,22의 에너지 경관은 단백질의 형성적 변화가 규제 역할에 필요한 것을 보여 주며, ns 시간척도23의 집단 막 변동에 걸쳐 발생하며, 세포막의 생물학적 기능과 생체 영감 에 대한 생물학적 기능에 있는 중각역학의 중요성을 더욱 강조하는 것이중요하다. 이 논문은 지질 막의 두 가지 주요 중전적 동적 모드, 즉 굽힘 변동 및 두께 변동에 중점을 둡니다.
이러한 변동 모드를 직접 조사하는 데 중요한 과제는 표준 분광법 방법을 사용하여 공간 및 측두형 스케일에 동시에 액세스하는 데 어려움이 있다는 것입니다. 다른 과제는 직접 접촉 기술이16을측정하기 위한 것과 동일한 변동에 영향을 줄 수 있다는 것입니다. 이는 생물학적 멤브레인(24)의 조성 및 구조적복잡성에의해 더욱 악화되어지질영역형성(26,27,28,29,30 및 막 비대칭31, 32,33)등 비균일한 멤브레인 특징을 초래하여 다른 멤브레인 특징의 역학을 이해하기 위해 선택적 프로브를 요구한다. 다행히도, 이러한 과제는 본질적으로 필요한 길이와 시간 척도에 액세스하는 중성자 스핀 에코(NSE)와 같은 비침습적 중성자 분광법 방법으로 극복될 수 있으며, 또한 그들의 물리화학적 환경을 변경하지 않고 선택적 멤브레인 특징의 연구를 가능하게 한다34. 실제로, 지난 몇 년 동안 NSE 분광기는 집단 막 역학의 독특하고 강력한 프로브로 진화했다35. 지질막에 대한 NSE 연구의 결과 기계적36,37 및 점성탄38,지질 막의39 속성에 대한 새로운 통찰력을 생산하고 생물학적 기능40,41에서자신의 잠재적 인 역할에 새로운 빛을 발산했다.
NSE 분광법은메제이(42)가처음 제안한 간섭계 설계를 기반으로 하며, 일련의 스핀 플리퍼와 자기 코일을 사용하여 중성자가 계측기를 통과할 때 중성자 스핀의 침체를 제어합니다. 설계는 시료위치(도 1A)에대하여 자기장 원소의 자기 미러링에 달려 있다. 이것은 중성자와 시료 사이의 에너지 교환이 없는 경우 중성자는 악기의 상반기와 후반에서 반대 방향으로 동일한 수의 스핀 프런치를 수행한다는 것을 의미합니다 (두 번의 패침체 코일 사이의 π 플립퍼를 알 수 있음). 그 결과, 중성자의 최종 스핀 상태는 초기 상태에 비해 변경되지 않은 상태로 유지- 스핀 에코라고 하는 현상(도 1A에서투명 중성자 참조). 그러나 중성자가 시료와 활발하게 상호 작용할 때 에너지 교환은 계측기의 후반부에서 스핀 프론치 수를 수정하여 다른 최종 스핀 상태로 이어집니다(그림 1A참조). 이것은 실험적으로 이 논문의 후반에 나타난 바와 같이 편광의 손실로 검출됩니다. NSE 기술에 대한 자세한 내용은 독자가 전용 기술논문(42,43,44,45)을참조합니다.
여기서, 단순화된 설명은 NSE로 액세스할 수 있는 길이 및 시간 척도에 대한 대략적인 추정치를 제공하기 위해 제시된다. 길이 스케일은 달성 가능한 파벡터 전달, Q = 4π 신 θ/λ의범위에 의해 결정되며, 여기서2θ는 산란 각및 λ는 중성자 파장이다. 하나는 Q가 파장 범위와 분광계의 두 번째 팔의 회전 정도에 의해 설정된 것을 볼 수 있습니다 (그림 1A참조). NSE 분광기의 전형적인 Q-범위는~0.02-2Å-146,47,및 0.01-4Å-1까지 최근 업그레이드48,49,~1-600 Å의 공간 척도에 해당한다. 한편, 접근 가능한 시간 척도는 자기 패침체 코일 내의 중성자에 의해 획득된 총 경기 침체 각도(또는 상)로부터계산되며, 50으로 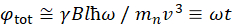 나타났다. 이 표현식에서 t는
나타났다. 이 표현식에서 t는 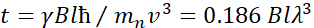
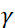 중성자자성자성비의 위치가
중성자자성자성비의 위치가 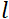 코일 길이이며 코일 자기장의 강도로 정의된 포리에
코일 길이이며 코일 자기장의 강도로 정의된 포리에 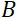 시간이다. Fourier 시간은 계측기 기하학, 자기장 강도 및 중성자 파장에 엄격하게 의존하는 양이라는 점을 지적할 가치가 있습니다. 예를 들어, 파장의 중성자를 사용
시간이다. Fourier 시간은 계측기 기하학, 자기장 강도 및 중성자 파장에 엄격하게 의존하는 양이라는 점을 지적할 가치가 있습니다. 예를 들어, 파장의 중성자를 사용 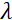 = 8 Å 및 악기 설정 =
= 8 Å 및 악기 설정 = 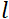 1.2 m 및 =
1.2 m 및 = 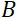 0.4 T, Fourier 시간은 t ~ 50 ns로 계산됩니다. 실험적으로, 포리에 시간은 p침체 코일(즉, 자기장 강도)에서 전류를 변경하거나 다른 중성자 파장을 사용하여 조정되어~ 1 ps ~ 100ns의 전형적인 NSE 시간 척도를 초래한다. 그러나, NSE 분광기의 최근 업그레이드는 하이네즈 마이어-라이프니츠 젠트럼51의 J-NSE-피닉스 분광기에서 최대 ~400ns까지 더 긴 포리어 타임에 접근할 수 있게 해주었으며, 오크 리지 국립 연구소48의SNS-NSE 분광기, 그리고 IN15 NSE 분광기에서 최대 ~1,000ns까지인루트 랭에프(Ill-Inute)
0.4 T, Fourier 시간은 t ~ 50 ns로 계산됩니다. 실험적으로, 포리에 시간은 p침체 코일(즉, 자기장 강도)에서 전류를 변경하거나 다른 중성자 파장을 사용하여 조정되어~ 1 ps ~ 100ns의 전형적인 NSE 시간 척도를 초래한다. 그러나, NSE 분광기의 최근 업그레이드는 하이네즈 마이어-라이프니츠 젠트럼51의 J-NSE-피닉스 분광기에서 최대 ~400ns까지 더 긴 포리어 타임에 접근할 수 있게 해주었으며, 오크 리지 국립 연구소48의SNS-NSE 분광기, 그리고 IN15 NSE 분광기에서 최대 ~1,000ns까지인루트 랭에프(Ill-Inute)
멤브레인 역학의 길이 및 시간 척도에 직접 액세스 하는 것 외에도, NSE는 중성자 동위원소 감도 의 고유 기능을 가지고52. 구체적으로, 중성자의 능력은 생물학적 시스템에서 가장 풍부한 소원인 수소의 동위원소와 다르게 상호 작용하는 능력으로, 상이한 중성자 산란 길이 밀도,34 또는 NSLD(굴절의 광학 지수에 상응하는50)를초래하며, 프로티엄은 중투란에 의해 대체된다. 이를 통해 특정 멤브레인 피쳐를 강조하거나 다른 사람을 숨기는 데 일반적으로 사용되는 콘트라스트 변형이라고 하는 접근 방식을 사용하여 후자의 시나리오를 대비 일치라고 합니다. 대조적 변화/매칭의 빈번한 적용은 중수 또는 D 2O(NSLD = 6.4 ×10 -6 å-2)에 의해 물의 대체물(NSLD= -0.56 × 10-6Å-2)으로유량지질막(NSLD ~0~0× -6Å-2)으로부터 중성자 신호를 증폭시키는 것이다. 이러한 접근법은 멤브레인의 헤드그룹 영역으로D2O의침투가 멤브레인 두께(도 2A,왼쪽 패널 참조) 및 보다 정교한 모델이적용될때 다른 지질 하위 그룹의 위치의 정확한 측정을 허용하기 때문에 멤브레인 구조의 연구에서 매우 효과적이다. 이 논문은 생물 막의 집단 역학 및 선택 막 특징의 연구를 위한 대비 변이의 사용에 대한 몇 가지 예를 강조합니다.
여기서, 동역 및 기능성 멤브레인 특성에 대한 고유한 통찰력을 제공하는 NSE의 효과는 리포소말 현탁액의 형태로 독립형 멤브레인의 중전역학에 중점을 둔 모델 및 생물학적으로 관련된 지질 막 시스템에 대한 NSE 연구의 유형 사례를 통해 설명됩니다. 평면 막 역학의 NSE 측정을 위해, 독자는 방목 발생 중성자 스핀 에코 분광법 (GINSES)55,56 및 정렬 다중 다각막 스택의 다른 연구에 전담 간행물을참조57,58,59,60.
단순성, 이 논문은 잘 연구된 도메인 형성, 또는 위상 분리, 1,2-dimyristoyl-sn-glycero-3-인포콜린(DMPC)의 지질 이중층 시스템 및 1,2-distearoyl-sn-glycero-3-phosphocholine(DSPC) 혼합물(DSPC) 혼합물(DSPC)61개에 대해 세 가지 다른 계획을 강조합니다. 두 지질은 탄화수소 사슬 길이(DMPC의 14탄/꼬리 vs DSPC의 18탄소/꼬리)와 겔유체 전이 온도(Tm, DMPC = 23°C vsTm, DSPC = 55°C)의 불일치를 특징으로 합니다. 이는 DMPC:DSPC 멤브레인에서 혼합물63의상부 및 낮은 전이 온도 사이의 온도에서 측면 상 분리를 초래한다. 여기서 고려되는 증단 방식은 리포소말 멤브레인, 즉 굽힘 변동, 두께 변동 및 측면 도메인의 선택적 굽힘/두께 변동에 대한 NSE 측정에서 액세스할 수 있는 다양한 동적 모드를 시연하기 위해 선택됩니다. 모든 지질 조성물은 DMPC및 DSPC의 상용 및 정해 변형을 사용하여 70:30의 몰트 분수로 제조된 DMPC:DSPC 이중층에 대해 보고됩니다. 모든 시료 준비 단계는 D2O에서 지질 농도가 50 mg/mL인 4mL의 지질 현탁액을 기반으로 하며, 총 지질 질량인 Mtot = 샘플당 200 mg을 기준으로 합니다.
Protocol
Representative Results
Discussion
NSE는 다양한 조건에서 지질 막의 중각역학을 측정하는 강력하고 독특한 기술입니다. NSE의 효과적인 활용은 샘플 품질, 중성자 대비 및 지정된 샘플에 대해 조사할 수 있는 접근 가능한 역학의 범위에 따라 달라집니다. 따라서 성공적인 NSE 실험을 수행하고 고품질 데이터를 수집하려면 몇 가지 중요한 단계가 필요합니다. NSE 실험 중 중성자 빔 시간을 효과적으로 사용하는 데 있어 중요한 단계는 NS…
Disclosures
The authors have nothing to disclose.
Acknowledgements
R. 아쉬카르 감사 M. 나가오, L.-R. Stingaciu, 그리고 P. Zolnierczuk 많은 유용한 토론과 그들의 각각의 빔라인에 NSE 실험에 그들의 빈번한 지원에 대 한. 저자는 NIST와 ORNL에서 중성자 스핀 에코 분광기의 사용을 인정합니다. NIST의 NSE 분광기는 국가 표준 기술 연구소와 국가 과학 재단 간의 파트너십인 고해상도 중성자 산란 센터의 지원을 받고 있습니다. DMR-1508249. ORNL의 Spallation 중성자 소스의 NSE 분광기는 미국 에너지부 기초 에너지 과학 부서인 과학 사용자 시설 부서에서 지원됩니다. 오크 리지 국립 연구소는 UT-Battelle에 의해 관리됩니다, 미국 DOE 계약 번호에 따라 LLC. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

