La spectroscopie neutronique à haute résolution pour étudier la dynamique picoseconde-nanoseconde des protéines et de l’eau d’hydratation
Summary
La spectroscopie de rétrodiffusion neutronique offre un accès non destructif et sans marquage à la dynamique ps-ns des protéines et de leur eau d’hydratation. Le flux de travail est présenté avec deux études sur les protéines amyloïdes: sur la dynamique résolue dans le temps du lysozyme pendant l’agrégation et sur la dynamique de l’eau d’hydratation de tau lors de la formation de fibres.
Abstract
La diffusion neutronique offre la possibilité de sonder la dynamique dans les échantillons pour une large gamme d’énergies d’une manière non destructive et sans marquage autre que le deutérium. En particulier, la spectroscopie de rétrodiffusion neutronique enregistre les signaux de diffusion à plusieurs angles de diffusion simultanément et est bien adaptée pour étudier la dynamique des systèmes biologiques sur l’échelle de temps ps-ns. En utilisant des composants tampons D2O et éventuellement deutérés, la méthode permet de surveiller à la fois la diffusion du centre de masse et les mouvements du squelette et de la chaîne latérale (dynamique interne) des protéines à l’état liquide.
De plus, la dynamique de l’eau d’hydratation peut être étudiée en utilisant des poudres de protéines perdeutérées hydratées avecH2O. Cet article présente le flux de travail utilisé sur l’instrument IN16B à l’Institut Laue-Langevin (ILL) pour étudier la dynamique des protéines et de l’eau d’hydratation. La préparation d’échantillons de solution et d’échantillons de poudre de protéines hydratées par échange de vapeur est expliquée. La procédure d’analyse des données pour la dynamique des protéines et de l’eau d’hydratation est décrite pour différents types d’ensembles de données (spectres quasi-élastiques ou balayages à fenêtre fixe) qui peuvent être obtenus sur un spectromètre de rétrodiffusion de neutrons.
La méthode est illustrée par deux études portant sur des protéines amyloïdes. L’agrégation du lysozyme en agrégats sphériques de taille μm – notés particules – se produit dans un processus en une étape sur l’espace et la plage temporelle sondés sur IN16B, tandis que la dynamique interne reste inchangée. De plus, la dynamique de l’hydratation de l’eau de tau a été étudiée sur des poudres hydratées de protéines perdeutérées. Il est montré que les mouvements de translation de l’eau sont activés lors de la formation de fibres amyloïdes. Enfin, les étapes critiques du protocole sont discutées quant à la position de la diffusion neutronique par rapport à l’étude de la dynamique par rapport à d’autres méthodes biophysiques expérimentales.
Introduction
Le neutron est une particule massive et sans charge qui a été utilisée avec succès au fil des ans pour sonder des échantillons dans divers domaines allant de la physique fondamentale à la biologie1. Pour les applications biologiques, la diffusion neutronique aux petits angles, la diffusion inélastique des neutrons, la cristallographie et la réflectométrie neutroniques sont largement utilisées 2,3,4. La diffusion inélastique des neutrons fournit une mesure de la dynamique en moyenne d’ensemble sans nécessiter de marquage spécifique en soi, et une qualité de signal qui ne dépend pas de la taille ou de la protéine5. La mesure peut être effectuée en utilisant un environnement très complexe pour la protéine étudiée qui imite le milieu intracellulaire, comme un lysat bactérien deutéré ou même in vivo 3,6,7. Différentes configurations expérimentales peuvent être utilisées pour étudier la dynamique, à savoir i) le temps de vol donnant accès à la dynamique sous-ps-ps, ii) l’accès à la rétrodiffusion donnant accès à la dynamique ps-ns, et iii) l’accès spin-echo-donnant accès à la dynamique de ns à des centaines de ns. La rétrodiffusion neutronique utilise la loi de Bragg 2d sinθ = nλ, où d est la distance entre les plans d’un cristal, θ l’angle de diffusion, n l’ordre de diffusion et λ la longueur d’onde. L’utilisation de cristaux pour la rétrodiffusion vers les détecteurs permet d’obtenir une haute résolution en énergie, typiquement ~0,8 μeV. Pour mesurer l’échange d’énergie, soit un entraînement Doppler transportant un cristal en rétrodiffusion est utilisé pour définir et régler la longueur d’onde neutronique entrante 8,9,10 (Figure 1), soit une configuration de temps de vol peut être utilisée au prix d’une diminution de la résolution d’énergie 11.
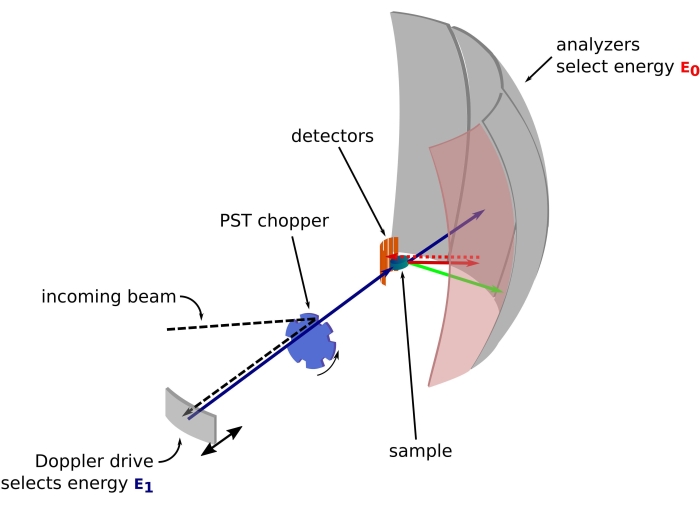
Figure 1 : Croquis d’un spectromètre de rétrodiffusion à neutrons avec un entraînement Doppler. Le faisceau entrant frappe le hacheur42 de transformation de l’espace de phase (PST), ce qui augmente le flux à la position de l’échantillon. Il est ensuite rétrodiffusé vers l’échantillon par le lecteur Doppler, qui sélectionne une énergieE1 (flèche cyan). Les neutrons sont ensuite dispersés par l’échantillon (avec différentes énergies représentées par la couleur des flèches) et les analyseurs, constitués de cristaux de Si 111, ne feront que rétrodiffuser des neutrons avec une énergie spécifique E0 (flèches de couleur rouge ici). Par conséquent, le transfert de quantité de mouvement q est obtenu à partir de la position détectée du neutron sur le réseau de détecteurs, et le transfert d’énergie est obtenu à partir de la différence E1– E0. Le temps de vol prévu pour l’impulsion de neutrons produite par le PST est utilisé pour rejeter le signal des neutrons diffusés directement vers les tubes détecteurs. Abréviation : PST = transformation de l’espace de phase. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Pour la spectroscopie de rétrodiffusion, la principale contribution au signal des échantillons riches en protons d’hydrogène, tels que les protéines, provient de la diffusion incohérente, pour laquelle l’intensité de diffusion Sinc(q, ω) est représentée par Eq (1)12
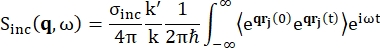 (1)
(1)
Où σinc est la section efficace incohérente de l’élément considéré, k’ est la norme du vecteur d’onde diffusé, k la norme du vecteur d’onde entrant, q (= k – k’) le transfert de quantité de mouvement, r j(t) le vecteur position de l’atome j au temps t, et ω la fréquence correspondant au transfert d’énergie entre le neutron entrant et le système. Les crochets angulaires indiquent la moyenne de l’ensemble. Par conséquent, la diffusion incohérente sonde l’auto-corrélation d’une seule particule moyenne d’ensemble des positions des atomes avec le temps et donne l’autodynamique moyenne sur tous les atomes du système et différentes origines temporelles (moyenne d’ensemble). La fonction de diffusion est la transformée de Fourier dans le temps de la fonction de diffusion intermédiaire I(q, t), qui peut être vue comme la transformée de Fourier dans l’espace de la fonction de corrélation de van Hove représentée par Eq (2):
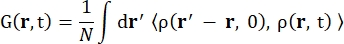 (2)
(2)
Où ρ(r,t) est la densité de probabilité de trouver un atome à la position r et au temps t 13.
Pour un processus de diffusion de Fickian, la fonction d’autodiffusion résulte (voir Eq (3)) après une double transformée de Fourier dans une fonction de diffusion consistant en un Lorentzien de largeur de droite donnée par γ = Dq2.
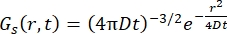 (3)
(3)
Des modèles plus sophistiqués ont été développés et jugés utiles, tels que le modèle de diffusion de saut de Singwi et Sjölander pour la dynamique des protéines internes ps-ns14 ou le modèle de rotation de Sears pour l’eau d’hydratation15,16,17.
Sur l’instrument de rétrodiffusion de neutrons (NBS) IN16B8,9 à l’ILL, Grenoble, France (Figure supplémentaire S1), une configuration couramment utilisée avec des protéines est constituée de cristaux de Si 111 pour les analyseurs avec un entraînement Doppler pour régler la longueur d’onde entrante (Figure supplémentaire S2A), donnant ainsi accès à la plage de transfert de quantité de mouvement ~0,2 Å-1 < q < ~2 Å-1 et à la plage de transfert d’énergie de –30 μeV < 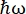 < 30 μeV-correspondant à des échelles de temps allant de quelques ps à quelques ns et des distances de quelques Å. En outre, IN16B offre la possibilité d’effectuer des balayages élastiques et inélastiques à fenêtre fixe (E/IFWS)10, qui incluent l’acquisition de données lors d’un transfert d’énergie fixe. Comme le flux est limité lorsque l’on travaille avec des neutrons, E/IFWS permet de maximiser le flux pour un transfert d’énergie, réduisant ainsi le temps d’acquisition nécessaire pour obtenir un rapport signal sur bruit satisfaisant. Une option plus récente est le spectromètre de rétrodiffusion et de temps de vol (BATS) mode11, qui permet de mesurer une large gamme de transferts d’énergie (par exemple, -150 μeV <
< 30 μeV-correspondant à des échelles de temps allant de quelques ps à quelques ns et des distances de quelques Å. En outre, IN16B offre la possibilité d’effectuer des balayages élastiques et inélastiques à fenêtre fixe (E/IFWS)10, qui incluent l’acquisition de données lors d’un transfert d’énergie fixe. Comme le flux est limité lorsque l’on travaille avec des neutrons, E/IFWS permet de maximiser le flux pour un transfert d’énergie, réduisant ainsi le temps d’acquisition nécessaire pour obtenir un rapport signal sur bruit satisfaisant. Une option plus récente est le spectromètre de rétrodiffusion et de temps de vol (BATS) mode11, qui permet de mesurer une large gamme de transferts d’énergie (par exemple, -150 μeV < 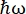 < 150 μeV), avec un flux plus élevé qu’avec le lecteur Doppler, mais au prix d’une résolution énergétique inférieure (figure supplémentaire S2B).
< 150 μeV), avec un flux plus élevé qu’avec le lecteur Doppler, mais au prix d’une résolution énergétique inférieure (figure supplémentaire S2B).
Une propriété importante de la diffusion des neutrons est que la section efficace incohérente σinc a une valeur 40 fois plus élevée pour l’hydrogène que pour le deutérium et est négligeable pour d’autres éléments couramment trouvés dans les échantillons biologiques. Par conséquent, la dynamique des protéines dans un environnement liquide peut être étudiée en utilisant un tampon deutérisé, et l’état de la poudre permet l’étude soit de la dynamique interne des protéines avec de la poudre de protéine hydrogénée hydratée avec D2O, soit de l’étude de l’eau d’hydratation pour la poudre de protéine perdeutérée hydratée avecH2O. À l’état liquide, la rétrodiffusion neutronique permet généralement d’accéder simultanément à l’autodiffusion du centre de masse des protéines (diffusion de type fickien) et à leur dynamique interne. Ces derniers sont des mouvements de colonne vertébrale et de chaîne latérale généralement décrits par le modèle dit de diffusion de saut ou d’autres 3,18. Dans les poudres de protéines hydrogénées, la diffusion des protéines est absente et seule la dynamique interne doit être modélisée. Pour l’eau d’hydratation, les contributions des mouvements translationnels et rotationnels des molécules d’eau présentent une dépendance différente du transfert de quantité de mouvement q, ce qui permet leur distinction dans le processus d’analyse des données17.
Cet article illustre la méthode de rétrodiffusion des neutrons avec l’étude des protéines qui se sont avérées capables de se déplier, de s’agréger sous une forme canonique composée d’empilements de brins β – le motif dit de βcroisée 19,20 – et de former des fibres allongées. C’est ce qu’on appelle l’agrégation amyloïde, qui est largement étudiée en raison de son rôle central dans les maladies neurodégénératives telles que la maladie d’Alzheimer ou la maladie de Parkinson21,22. L’étude des protéines amyloïdes est également motivée par le rôle fonctionnel qu’elles peuvent jouer 23,24 ou leur fort potentiel pour le développement de nouveaux biomatériaux25. Les déterminants physico-chimiques de l’agrégation amyloïde restent flous, et aucune théorie générale de l’agrégation amyloïde n’est disponible, malgré d’énormes progrès au cours des dernières années21,26.
L’agrégation amyloïde implique des changements dans la structure et la stabilité des protéines avec le temps, dont l’étude implique naturellement une dynamique, liée à la stabilité de la conformation des protéines, à la fonction protéique et au paysage énergétique des protéines27. La dynamique est directement liée à la stabilité d’un état spécifique par la contribution entropique pour les mouvements les plus rapides28, et la fonction protéique peut être maintenue par des mouvements sur différentes échelles de temps de sub-ps pour les protéines sensibles à la lumière29 à ms pour les mouvements de domaine, ce qui peut être facilité par la dynamique picoseconde-nanoseconde30.
Deux exemples d’utilisation de la spectroscopie de rétrodiffusion neutronique pour étudier les protéines amyloïdes seront présentés, l’un à l’état liquide pour étudier la dynamique des protéines et l’autre à l’état de poudre hydratée pour étudier la dynamique de l’hydratation de l’eau. Le premier exemple concerne l’agrégation du lysozyme en sphères de taille μm (appelées particules) suivies en temps réel5, et le second une comparaison de la dynamique de l’eau aux états natifs et agrégés de la protéine humaine tau31.
Le lysozyme est une enzyme impliquée dans la défense immunitaire et est composé de 129 résidus d’acides aminés. Le lysozyme peut former des particules dans un tampon deutéré à une température de 10,5 et à une température de 90 °C. Avec la diffusion neutronique, nous avons montré que l’évolution temporelle du coefficient de diffusion du centre de masse du lysozyme suit la cinétique exponentielle unique de la fluorescence T de la thioflavine (une sonde fluorescente utilisée pour surveiller la formation de motifs de β croisée amyloïde32), indiquant que les superstructures particulaires de formation et les modèles de β croisée se produisent en une seule étape avec le même taux. De plus, la dynamique interne est restée constante tout au long du processus d’agrégation, ce qui peut s’expliquer soit par un changement conformationnel rapide qui ne peut être observé sur les instruments NBS, soit par l’absence de changement significatif de l’énergie interne des protéines lors de l’agrégation.
La protéine tau humaine est une protéine intrinsèquement désordonnée (PDI) constituée de 441 acides aminés pour l’isoforme dite 2N4R, qui est notamment impliquée dans la maladie d’Alzheimer33. En utilisant la rétrodiffusion neutronique sur des poudres de protéine tau perdeutérée, nous avons montré que la dynamique de l’eau d’hydratation est augmentée à l’état fibreux, avec une population plus élevée de molécules d’eau subissant des mouvements de translation. Le résultat suggère qu’une augmentation de l’entropie de l’eau d’hydratation pourrait conduire la fibrillation amyloïde de tau.
Protocol
Representative Results
Discussion
La spectroscopie neutronique est la seule méthode qui permet de sonder la dynamique ps-ns moyenne d’ensemble des échantillons de protéines, quelle que soit la taille de la protéine ou la complexité de la solution lorsque la deutération est utilisée6. Plus précisément, en sondant l’autodiffusion d’assemblages protéiques en solution, la taille hydrodynamique de ces assemblages peut être déterminée sans ambiguïté. Néanmoins, la méthode est généralement limitée par le faible …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les auteurs remercient Michaela Zamponi du Jülich Centre for Neutron Science du Heinz Maier-Leibnitz Zentrum, Garching, Allemagne, pour une partie des expériences de diffusion neutronique menées sur l’instrument SPHERES. Ces travaux ont bénéficié des activités du consortium Deuteration Laboratory (DLAB) financé par l’Union européenne dans le cadre des contrats HPRI-2001-50065 et RII3-CT-2003-505925, et de l’activité financée par le UK Engineering and Physical Sciences Research Council (EPSRC) au sein de l’Institut Laue Langevin EMBL DLAB sous les subventions GR/R99393/01 et EP/C015452/1. Le soutien apporté par la Commission européenne au titre du 7e programme-cadre par l’intermédiaire de l’action clé « Renforcer l’Espace européen de la recherche, infrastructures de recherche » est reconnu [Contrat 226507 (NMI3)]. Kevin Pounot et Christian Beck remercient le ministère fédéral de l’Éducation et de la Recherche (BMBF, numéro de subvention 05K19VTB) pour le financement de leurs bourses postdoctorales.
Materials
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 |
|
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
References
- Jacrot, B. Des neutrons pour la science: Histoire de l’Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer’s β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry – An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).

