단백질과 수화수의 피코초-나노초 역학을 연구하기 위한 고분해능 중성자 분광법
Summary
중성자 후방 산란 분광법은 단백질과 단백질의 수화수의 ps-ns 역학에 대한 비파괴적이고 표지 없는 접근을 제공합니다. 워크플로우는 아밀로이드 단백질에 대한 두 가지 연구, 즉 응집 중 리소자임의 시간 분해 역학과 섬유 형성 시 타우의 수화 물 역학에 대한 연구로 제공됩니다.
Abstract
중성자 산란은 중수소 이외의 표지 없이 비파괴적인 방식으로 광범위한 에너지에 대한 샘플 내 역학을 조사할 수 있는 가능성을 제공합니다. 특히, 중성자 후방 산란 분광법은 여러 산란 각도에서 산란 신호를 동시에 기록하며 ps-ns 시간 척도에서 생물학적 시스템의 역학을 연구하는 데 매우 적합합니다. D2O및 중수소화 완충액 성분을 사용함으로써, 이 방법은 액체 상태에서 단백질의 질량 중심 확산 및 골격 및 측쇄 운동(내부 역학)을 모두 모니터링할 수 있습니다.
또한, 수화수 역학은 H2O로 수화된 과중수소화 단백질 분말을 사용하여 연구할 수 있습니다. 이 논문은 단백질 및 수화수 역학을 조사하기 위해 Institut Laue-Langevin(ILL)의 IN16B 기기에 사용된 워크플로를 제시합니다. 증기 교환을 이용한 용액 시료 및 수화된 단백질 분말 시료의 제조에 대해 설명합니다. 단백질 및 수화수 역학에 대한 데이터 분석 절차는 중성자 후방 산란 분광계에서 얻을 수 있는 다양한 유형의 데이터 세트(준탄성 스펙트럼 또는 고정 창 스캔)에 대해 설명됩니다.
이 방법은 아밀로이드 단백질과 관련된 두 가지 연구로 설명됩니다. 리소자임이 μm 크기의 구형 응집체(미립자로 표시됨)로 응집되는 것은 IN16B에서 조사된 공간 및 시간 범위에서 1단계 공정으로 발생하는 것으로 나타났지만 내부 역학은 변하지 않았습니다. 또한, 타우의 수화수의 역학은 과중수소화 단백질의 수화된 분말에 대해 연구되었다. 물의 병진 운동은 아밀로이드 섬유의 형성시 활성화되는 것으로 나타났습니다. 마지막으로, 프로토콜의 중요한 단계는 다른 실험적 생물물리학적 방법과 관련하여 역학 연구와 관련하여 중성자 산란이 어떻게 위치하는지에 대해 논의됩니다.
Introduction
중성자는 기초 물리학에서 생물학에 이르기까지 다양한 분야의 샘플을 탐사하는 데 수년에 걸쳐 성공적으로 사용되어 온 전하가 없는 거대한 입자입니다1. 생물학적 응용을 위해, 소각 중성자 산란, 비탄성 중성자 산란, 중성자 결정학 및 반사 측정법이 광범위하게 사용된다 2,3,4. 비탄성 중성자 산란은 특정 표지 그 자체를 필요로 하지 않고 역학의 앙상블 평균 측정을 제공하며, 크기 또는 단백질에 의존하지 않는 신호 품질을 제공한다5. 측정은 중수소화 박테리아 용해물 또는 생체 내 3,6,7과 같은 세포내 배지를 모방하는 연구 중인 단백질에 대해 매우 복잡한 환경을 사용하여 수행할 수 있습니다. 역학을 연구하기 위해 다양한 실험 설정, 즉 i) sub-ps-ps 역학에 대한 비행 시간 액세스 제공, ii) ps-ns 역학에 대한 후방 산란 제공 액세스 및 iii) ns에서 수백 ns까지의 역학에 대한 스핀 에코 제공 액세스. 중성자 후방 산란은 브래그의 법칙 2d sinθ = nλ를 사용하며, 여기서 d는 결정에서 평면 사이의 거리, θ는 산란 각도, n은 산란 차수, λ는 파장입니다. 검출기를 향한 후방 산란을 위해 크리스털을 사용하면 일반적으로 ~0.8μeV의 높은 에너지 분해능을 달성할 수 있습니다. 에너지 교환을 측정하기 위해, 후방 산란에서 크리스털을 운반하는 도플러 드라이브를 사용하여 들어오는 중성자 파장8,9,10을 정의하고 조정하거나(그림 1), 에너지 분해능을 감소시키는 대가로 ToF(Time-of-Flight) 설정을 사용할 수 있다(11).
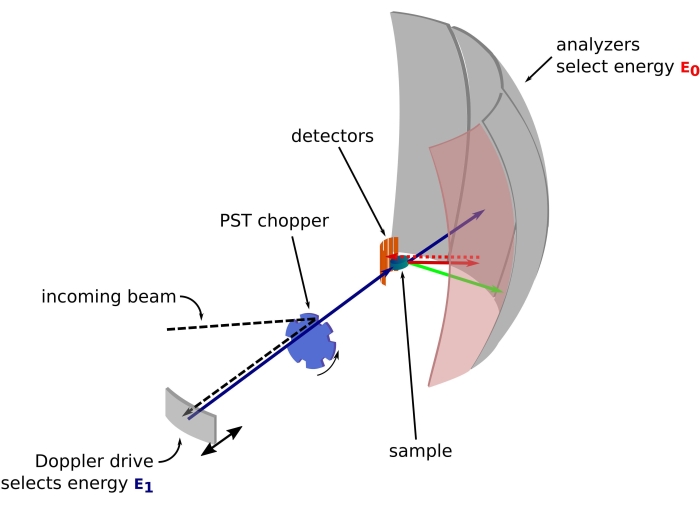
그림 1: 도플러 드라이브가 있는 중성자 후방 산란 분광계의 스케치. 인입 빔은 위상 공간 변환(PST) 초퍼(42)에 부딪히고, 이는 샘플 위치에서의 플럭스를 증가시킨다. 그런 다음 에너지 E1(청록색 화살표)을 선택하는 도플러 드라이브에 의해 샘플을 향해 후방 산란됩니다. 그런 다음 중성자는 샘플에 의해 산란되고(화살표 색상으로 표시되는 다른 에너지로) Si 111 결정으로 만들어진 분석기는 특정 에너지 E0(여기서는 빨간색 화살표)를 가진 중성자만 후방 산란합니다. 따라서, 운동량 전달 q는 검출기 어레이 상의 중성자의 검출 위치로부터 얻어지고, 에너지 전달은 차이E1-E0로부터 얻어진다. PST에 의해 생성된 중성자 펄스에 대해 예상되는 비행 시간은 검출기 튜브를 향해 직접 산란된 중성자의 신호를 버리는 데 사용됩니다. 약어: PST = 위상 공간 변환. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
후방 산란 분광법의 경우, 단백질과 같은 수소 양성자가 풍부한 샘플의 신호에 대한 주요 기여는 비간섭성 산란에서 비롯되며, 이에 대해 산란 강도 Sinc(q, ω)는 Eq (1)12로 표시됩니다
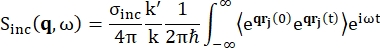 (1개)
(1개)
여기서σ inc는 고려된 요소의 비간섭성 단면이고, k’는 산란된 파동 벡터의 노름이고, k는 들어오는 파동 벡터의 노름이고, q(= k – k’)는 운동량 전달, r j(t)는 시간 t에서 원자 j의 위치 벡터이고, ω는 들어오는 중성자와 시스템 사이의 에너지 전달에 해당하는 주파수입니다. 꺾은 괄호는 앙상블 평균을 나타냅니다. 따라서 비간섭성 산란은 시간에 따른 원자 위치의 앙상블 평균 단일 입자 자기 상관을 조사하고 시스템의 모든 원자와 다른 시간 기원(앙상블 평균)에 대한 자체 역학 평균을 제공합니다. 산란 함수는 중간 산란 함수 I(q, t)의 시간에서의 푸리에 변환이며, 이는 Eq (2)에 의해 표시된 van Hove 상관 함수의 공간에서의 푸리에 변환으로 볼 수 있습니다.
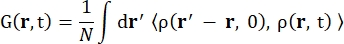 (2개)
(2개)
여기서 ρ(r,t)는 위치 r과 시간 t 13에서 원자를 찾을 확률 밀도입니다.
Fickian 확산 과정의 경우, γ = Dq2로 주어진 선폭의 로렌치안으로 구성된 산란 함수에서 이중 푸리에 변환 후 자기 확산 함수 결과(Eq (3) 참조)가 발생합니다.
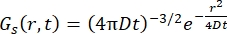 (3)
(3)
ps-ns 내부 단백질 역학14에 대한 Singwi 및 Sjölander의 점프 확산 모델 또는 수화수15,16,17에 대한 Sears의 회전 모델과 같이 보다 정교한 모델이 개발되어 유용한 것으로 나타났습니다.
프랑스 그르노블의 ILL에 있는 중성자 후방 산란(NBS) 기기 IN16B 8,9(보충 그림 S1)에서 단백질과 함께 일반적으로 사용되는 설정은 들어오는 파장을 조정하기 위한 도플러 드라이브가 있는 분석기용 Si 111 결정으로 구성되며(보충 그림 S2A), 이에 따라 ~0.2 Å-1 <q < ~2 Å-1의 운동량 전달 범위 및 –30 μeV의 에너지 전달 범위에 대한 액세스를 제공합니다<  < 30 μeV – 몇 ps에서 몇 ns 범위의 시간 척도 및 몇 Å의 거리에 해당합니다. 또한 IN16B는 고정 에너지 전송에서 데이터 수집을 포함하는 탄성 및 비탄성 고정 창 스캔(E/IFWS)10을 수행할 수 있는 가능성을 제공합니다. 중성자로 작업할 때 플럭스가 제한되기 때문에 E/IFWS를 사용하면 한 번의 에너지 전달에 대해 플럭스를 최대화할 수 있으므로 만족스러운 신호 대 잡음비를 얻는 데 필요한 획득 시간을 줄일 수 있습니다. 보다 최근의 옵션은 후방 산란 및 비행 시간 분광계(BATS) 모드(11)로, 도플러 드라이브보다 더 높은 플럭스로 광범위한 에너지 전달(예: -150μeV < <
< 30 μeV – 몇 ps에서 몇 ns 범위의 시간 척도 및 몇 Å의 거리에 해당합니다. 또한 IN16B는 고정 에너지 전송에서 데이터 수집을 포함하는 탄성 및 비탄성 고정 창 스캔(E/IFWS)10을 수행할 수 있는 가능성을 제공합니다. 중성자로 작업할 때 플럭스가 제한되기 때문에 E/IFWS를 사용하면 한 번의 에너지 전달에 대해 플럭스를 최대화할 수 있으므로 만족스러운 신호 대 잡음비를 얻는 데 필요한 획득 시간을 줄일 수 있습니다. 보다 최근의 옵션은 후방 산란 및 비행 시간 분광계(BATS) 모드(11)로, 도플러 드라이브보다 더 높은 플럭스로 광범위한 에너지 전달(예: -150μeV < <  150μeV)을 측정할 수 있지만 에너지 분해능은 더 낮습니다(보충 그림 S2B).
150μeV)을 측정할 수 있지만 에너지 분해능은 더 낮습니다(보충 그림 S2B).
중성자 산란의 중요한 특성은 incoherent 단면σ inc가 중수소보다 수소에 대해 40배 더 높은 값을 가지며 생물학적 샘플에서 일반적으로 발견되는 다른 원소에 대해서는 무시할 수 있다는 것입니다. 따라서, 액체 환경에서 단백질의 역학은 중수소화 완충액을 사용하여 연구 할 수 있으며, 분말 상태는 D 2 O로 수화 된 수소화 단백질 분말을 사용한 단백질 내부 역학 연구 또는 H2O로 수화 된 과중수소화 단백질 분말에 대한 수화수 연구를 허용합니다. 액체 상태에서, 중성자 후방 산란은 전형적으로 단백질의 질량 중심 자기 확산 (Fickian-type diffusion)과 그 내부 역학에 동시에 접근 할 수있게한다. 후자는 일반적으로 소위 점프 확산 모델 또는 기타 3,18에 의해 설명되는 백본 및 사이드 체인 운동입니다. 수소화 단백질 분말에서는 단백질 확산이 없으며 내부 역학만 모델링하면 됩니다. 수화수의 경우, 물 분자의 병진 및 회전 운동의 기여도는 운동량 전달 q에 대한 다른 의존성을 나타내며, 이는 데이터 분석 프로세스17에서 구별할 수 있습니다.
이 논문은 전개될 수 있는 것으로 밝혀진 단백질에 대한 연구와 함께 중성자 후방 산란 방법을 설명하고, 소위 교차 β 패턴19,20이라고 하는 β가닥 스택으로 구성된 표준 형태로 응집되고 길쭉한 섬유를 형성합니다. 이것은 소위 아밀로이드 응집체이며, 이는 알츠하이머 병이나 파킨슨 병과 같은 신경 퇴행성 질환에서 중심적인 역할을하기 때문에 광범위하게 연구되고 있습니다21,22. 아밀로이드 단백질에 대한 연구는 또한 아밀로이드 단백질이 할 수 있는 기능적 역할에 의해 동기가 부여된다23,24 또는 새로운 생체 재료 개발에 대한 높은 잠재력 25. 아밀로이드 응집의 물리 화학적 결정 인자는 불분명하며, 지난 몇 년 동안 엄청난 진전이 있었음에도 불구하고 아밀로이드 응집에 대한 일반적인 이론은 없다21,26.
아밀로이드 응집은 시간에 따른 단백질 구조와 안정성의 변화를 의미하며, 이에 대한 연구는 자연적으로 단백질 형태 안정성, 단백질 기능 및 단백질 에너지 환경과 관련된 역학을 의미한다27. 동역학은 가장 빠른 운동(28)에 대한 엔트로피 기여를 통해 특정 상태의 안정성과 직접적으로 연결되며, 단백질 기능은 빛에 민감한 단백질(29 )에 대한 sub-ps에서 도메인 운동에 대한 ms에 이르기까지 다양한 시간 척도의 운동에 의해 유지될 수 있으며, 이는 피코초-나노초 역학(30)에 의해 촉진될 수 있다.
아밀로이드 단백질을 연구하기 위해 중성자 후방 산란 분광법을 사용하는 두 가지 예가 제시될 것인데, 하나는 단백질 역학을 연구하기 위해 액체 상태에서, 다른 하나는 수화 물 역학을 연구하기 위해 수화된 분말 상태입니다. 첫 번째 예는 리소자임을 μm 크기의 구체(미립자라고 함)로 응집하는 것과 관련이있으며, 두 번째 예는 인간 단백질 타우(tau)의 천연 상태와 응집 상태에서의 물 역학 비교에 관한 것이다31.
리소자임은 면역 방어에 관여하는 효소로 129개의 아미노산 잔기로 구성되어 있습니다. 리소자임은 10.5의 pD 및 90°C의 온도에서 중수소화 완충액에서 미립자를 형성할 수 있습니다. 중성자 산란을 통해 우리는 리소자임의 질량 중심 확산 계수의 시간 진화가 티오플라빈 T 형광(아밀로이드 교차 β 패턴32의 형성을 모니터링하는 데 사용되는 형광 프로브)의 단일 지수 동역학을 따른다는 것을 보여주었으며, 이는 형성 미립자 상부 구조와 교차 β 패턴이 동일한 속도로 단일 단계에서 발생함을 나타냅니다. 또한, 내부 역학은 응집 과정 전반에 걸쳐 일정하게 유지되었으며, 이는 NBS 기기에서 관찰할 수 없는 빠른 구조적 변화 또는 응집 시 단백질 내부 에너지의 유의미한 변화가 없는 것으로 설명할 수 있습니다.
인간 단백질 타우(tau)는 소위 2N4R 이소형(isoform)을 위한 441개의 아미노산으로 구성된 본질적으로 무질서한 단백질(IDP)로, 특히 알츠하이머병에 관여한다33. 과중수소화된 단백질 타우의 분말에 중성자 후방 산란을 사용하여, 우리는 수화 물 역학이 섬유 상태에서 증가하고, 더 많은 물 분자 집단이 병진 운동을 겪는다는 것을 보여주었습니다. 결과는 수화수 엔트로피의 증가가 타우의 아밀로이드 세동을 유발할 수 있음을 시사합니다.
Protocol
Representative Results
Discussion
중성자 분광법(neutron spectroscopy)은 중수소를 사용할 때 단백질의 크기나 용액의 복잡성에 관계없이 단백질 샘플의 앙상블 평균 ps-ns 역학을 조사할 수 있는 유일한 방법이다6. 구체적으로, 용액에서 단백질 어셈블리의 자체 확산을 조사함으로써, 이러한 어셈블리의 유체역학적 크기를 명확하게 결정할 수 있습니다. 그럼에도 불구하고, 이 방법은 일반적으로 낮은 중성자 플럭스에…
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자들은 SPHERES 장비에서 수행된 중성자 산란 실험의 일부에 대해 독일 가르힝의 Heinz Maier-Leibnitz Zentrum에 있는 Jülich 중성자 과학 센터의 Michaela Zamponi에게 감사를 표합니다. 이 작업은 HPRI-2001-50065 및 RII3-CT-2003-505925 계약에 따라 유럽 연합이 자금을 지원하는 DLAB(Deuteration Laboratory) 컨소시엄의 활동과 보조금 GR/R99393/01 및 EP/C015452/1에 따라 Institut Laue Langevin EMBL DLAB 내에서 영국 공학 및 물리 과학 연구 위원회(EPSRC)가 자금을 지원하는 활동의 혜택을 받았습니다. 제7차 프레임워크 프로그램에 따른 유럽연합 집행위원회(European Commission)의 주요 조치: 유럽 연구 영역 강화, 연구 인프라 강화(Key Action: Strengthening the European Research Area, Research Infrastructures)를 통해 [계약 226507(NMI3)]을 인정합니다. 케빈 푸노 (Kevin Pounot)와 크리스티안 벡 (Christian Beck)은 박사후 연구원 기금에 대해 연방 교육 연구부 (BMBF, 보조금 번호 05K19VTB)에 감사드립니다.
Materials
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 |
|
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
References
- Jacrot, B. Des neutrons pour la science: Histoire de l’Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer’s β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry – An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).

