Högupplöst neutronspektroskopi för att studera pikosekund-nanosekunddynamik hos proteiner och hydratiseringsvatten
Summary
Neutronbackspridningsspektroskopi erbjuder en icke-destruktiv och etikettfri tillgång till ps-ns-dynamiken hos proteiner och deras hydratiseringsvatten. Arbetsflödet presenteras med två studier på amyloidproteiner: på den tidsupplösta dynamiken av lysozym under aggregering och på hydratiseringsvattendynamiken hos tau vid fiberbildning.
Abstract
Neutronspridning erbjuder möjligheten att undersöka dynamiken i prover för ett brett spektrum av energier på ett icke-destruktivt sätt och utan annan märkning än deuterium. I synnerhet registrerar neutronspridningsspektroskopi spridningssignalerna vid flera spridningsvinklar samtidigt och är väl lämpad för att studera dynamiken i biologiska system på ps-ns tidsskala. Genom att använda D2O-och eventuellt deutererade buffertkomponenter möjliggör metoden övervakning av både masscentrumdiffusion och ryggrads- och sidokedjerörelser (inre dynamik) hos proteiner i flytande tillstånd.
Dessutom kan hydratiseringsvattendynamik studeras genom att använda pulver av perdeutererade proteiner hydratiserade medH2O. Denna uppsats presenterar arbetsflödet som används på instrumentet IN16B vid Institut Laue-Langevin (ILL) för att undersöka protein- och hydratiseringsvattendynamik. Beredningen av lösningsprover och hydratiserade proteinpulverprover med ångutbyte förklaras. Dataanalysproceduren för både protein- och hydratiseringsvattendynamik beskrivs för olika typer av dataset (kvasielastiska spektra eller fasta fönsterskanningar) som kan erhållas på en neutronspridningsspektrometer.
Metoden illustreras med två studier med amyloidproteiner. Aggregeringen av lysozym i sfäriska aggregat av μm-storlek – betecknade partiklar – visas ske i en enstegsprocess på rymd- och tidsintervallet som sonderas på IN16B, medan den interna dynamiken förblir oförändrad. Vidare studerades dynamiken hos hydratiseringsvatten av tau på hydratiserade pulver av perdeuterat protein. Det visas att translationella rörelser av vatten aktiveras vid bildandet av amyloidfibrer. Slutligen diskuteras kritiska steg i protokollet för hur neutronspridning positioneras när det gäller studier av dynamik med avseende på andra experimentella biofysiska metoder.
Introduction
Neutronen är en laddningsfri och massiv partikel som framgångsrikt har använts genom åren för att undersöka prover inom olika områden från grundläggande fysik till biologi1. För biologiska tillämpningar används neutronspridning med liten vinkel, inelastisk neutronspridning och neutronkristallografi och reflektometri i stor utsträckning 2,3,4. Inelastisk neutronspridning ger en ensemble-medelvärdesmätning av dynamiken utan att kräva specifik märkning i sig, och en signalkvalitet som inte beror på storleken eller proteinet5. Mätningen kan göras med hjälp av en mycket komplex miljö för proteinet som studeras som efterliknar det intracellulära mediet, såsom ett deutererat bakteriellt lysat eller till och med in vivo 3,6,7. Olika experimentella uppställningar kan användas för att studera dynamiken, nämligen i) time-of-flight-giving access to sub-ps-ps dynamics, ii) backscattering-giving access to ps-ns dynamics, och iii) spin-echo-giving access to dynamics from ns to hundreds ns. Neutronåterspridning använder Braggs lag 2d sinθ = nλ, där d är avståndet mellan plan i en kristall, θ spridningsvinkeln, n spridningsordningen och λ våglängden. Användningen av kristaller för bakåtspridning mot detektorerna möjliggör en hög upplösning i energi, typiskt ~ 0,8 μeV. För att mäta energiutbytet används antingen en dopplerdrivenhet som bär en kristall i backscattering för att definiera och ställa in den inkommande neutronvåglängden 8,9,10 (figur 1), eller en flygtidsinställning kan användas på bekostnad av en minskning av energiupplösningen 11.
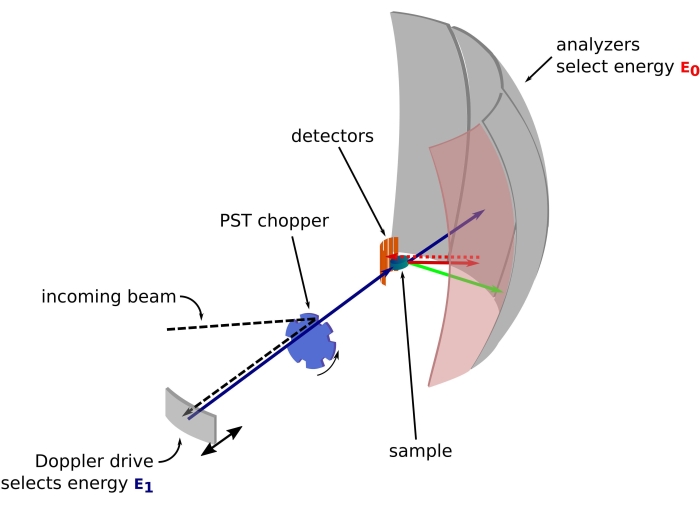
Figur 1: Skiss av en neutronspridningsspektrometer med dopplerdrift. Den inkommande strålen träffar fasrumstransformationen (PST) chopper42, vilket ökar flödet vid provpositionen. Den sprids sedan bakåt mot provet av dopplerdrivningen, som väljer en energi E1 (cyanpil). Neutronerna sprids sedan av provet (med olika energier representerade av pilarnas färg) och analysatorerna, gjorda av Si 111-kristaller, kommer bara att sprida neutroner med en specifik energi E0 (rödfärgade pilar här). Därför erhålls momentöverföringen q från neutronens detekterade position på detektormatrisen och energiöverföringen erhålls från skillnaden E1– E0. Den flygtid som förväntas för neutronpulsen som produceras av PST används för att kassera signalen från neutronerna spridda direkt mot detektorrören. Förkortning: PST = fasrymdtransformation. Klicka här för att se en större version av denna figur.
För backscatteringspektroskopi kommer det huvudsakliga bidraget till signalen från väteprotonrika prover, såsom proteiner, från inkoherent spridning, för vilken spridningsintensiteten Sinc (q, ω) visas av Eq (1) 12
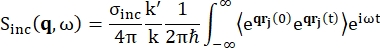 (1)
(1)
Därσ inc är det osammanhängande tvärsnittet av det element som beaktas, är k’ normen för den spridda vågvektorn, k normen för den inkommande vågvektorn, q (= k – k’) momentöverföringen, r j (t) positionsvektorn för atom j vid tiden t och ω frekvensen som motsvarar energiöverföringen mellan den inkommande neutronen och systemet. Vinkelparenteserna anger ensemblemedelvärdet. Därför undersöker inkoherent spridning ensemblemedelvärdet av enpartikels självkorrelation av atompositioner med tiden och ger självdynamiken i genomsnitt över alla atomer i systemet och olika tidsursprung (ensemblemedelvärde). Spridningsfunktionen är Fouriertransformen i tiden av den mellanliggande spridningsfunktionen I(q, t), som kan ses som Fouriertransformen i rummet av van Hove-korrelationsfunktionen som visas av Eq (2):
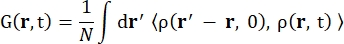 (2)
(2)
Där ρ(r,t) är sannolikhetstätheten för att hitta en atom vid position r och tiden t 13.
För en fickiansk diffusionsprocess resulterar självdiffusionsfunktionen (se Eq (3)) efter en dubbel Fouriertransform i en spridningsfunktion bestående av en Lorentzian med linjebredd given av γ = Dq2.
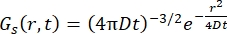 (3)
(3)
Mer sofistikerade modeller utvecklades och befanns användbara såsom hoppdiffusionsmodellen av Singwi och Sjölander för ps-ns interna proteindynamik14 eller rotationsmodellen av Sears för hydratiseringsvatten15,16,17.
På neutronspridningsinstrumentet IN16B 8,9 vid ILL, Grenoble, Frankrike (kompletterande figur S1) består en inställning som vanligtvis används med proteiner av Si 111-kristaller för analysatorerna med en dopplerdrivning för att ställa in den inkommande våglängden (kompletterande figur S2A), vilket ger tillgång till momentöverföringsområdet ~ 0,2 Å-1 < q < ~ 2 Å-1 och energiöverföringsområdet –30 μeV < 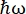 < 30 μeV motsvarande tidsskalor från några ps till några ns och avstånd på några Å. Dessutom erbjuder IN16B möjligheten att utföra elastiska och oelastiska skanningar med fasta fönster (E/IFWS)10, vilket inkluderar datainsamling vid en fast energiöverföring. Eftersom flödet är begränsat vid arbete med neutroner möjliggör E/IFWS maximering av flödet för en energiöverföring, vilket minskar förvärvstiden som behövs för att uppnå ett tillfredsställande signal-brusförhållande. Ett nyare alternativ är backscattering och time-of-flight spectrometer (BATS) mode11, som möjliggör mätning av ett brett spektrum av energiöverföringar, (t.ex. -150 μeV < <
< 30 μeV motsvarande tidsskalor från några ps till några ns och avstånd på några Å. Dessutom erbjuder IN16B möjligheten att utföra elastiska och oelastiska skanningar med fasta fönster (E/IFWS)10, vilket inkluderar datainsamling vid en fast energiöverföring. Eftersom flödet är begränsat vid arbete med neutroner möjliggör E/IFWS maximering av flödet för en energiöverföring, vilket minskar förvärvstiden som behövs för att uppnå ett tillfredsställande signal-brusförhållande. Ett nyare alternativ är backscattering och time-of-flight spectrometer (BATS) mode11, som möjliggör mätning av ett brett spektrum av energiöverföringar, (t.ex. -150 μeV < < 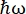 150 μeV), med ett högre flöde än med dopplerdriften, men på bekostnad av en lägre energiupplösning (kompletterande figur S2B).
150 μeV), med ett högre flöde än med dopplerdriften, men på bekostnad av en lägre energiupplösning (kompletterande figur S2B).
En viktig egenskap hos neutronspridning är att det inkoherenta tvärsnittet σinc har ett 40 gånger högre värde för väte än för deuterium och är försumbar för andra element som vanligtvis finns i biologiska prover. Därför kan dynamiken hos proteiner i en flytande miljö studeras med hjälp av en deutererad buffert, och pulvertillståndet möjliggör studier av antingen proteinets inre dynamik med hydrerat proteinpulver hydratiserat med D2O, eller studien av hydratiseringsvatten för perdeutererat proteinpulver hydratiserat medH2O. I flytande tillstånd tillåter neutronåterspridning typiskt samtidig åtkomst till masscentrets självdiffusion av proteiner (diffusion av fickian-typ) och deras interna dynamik. De senare är ryggrads- och sidokedjerörelser som vanligtvis beskrivs av den så kallade hoppdiffusionsmodellen eller andra 3,18. I hydrerade proteinpulver saknas proteindiffusionen och endast intern dynamik behöver modelleras. För hydratiseringsvatten uppvisar bidragen från translationella och roterande rörelser av vattenmolekyler ett annat beroende av momentöverföringen q, vilket möjliggör deras distinktion i dataanalysprocessen17.
Denna artikel illustrerar neutronspridningsmetoden med studier av proteiner som visade sig kunna utvecklas, aggregera till en kanonisk form bestående av staplar av β-strängar – det så kallade kors-β mönstret19,20 – och bilda långsträckta fibrer. Detta är den så kallade amyloidaggregeringen, som studeras omfattande på grund av dess centrala roll i neurodegenerativa sjukdomar som Alzheimers eller Parkinsons sjukdomar21,22. Studien av amyloidproteinerna motiveras också av den funktionella roll de kan spela 23,24 eller deras höga potential för utveckling av nya biomaterial25. De fysikalisk-kemiska determinanterna för amyloidaggregeringen är fortfarande oklara, och ingen allmän teori om amyloidaggregering finns tillgänglig, trots enorma framsteg under de senaste åren21,26.
Amyloidaggregering innebär förändringar i proteinstruktur och stabilitet med tiden, vars studie naturligtvis innebär dynamik, kopplad till proteinkonformationsstabilitet, proteinfunktion och proteinenergilandskap27. Dynamik är direkt kopplad till stabiliteten hos ett specifikt tillstånd genom det entropiska bidraget för de snabbaste rörelserna28, och proteinfunktionen kan upprätthållas av rörelser på olika tidsskalor från sub-ps för ljuskänsliga proteiner29 till ms för domänrörelser, vilket kan underlättas av picosecond-nanosecond dynamics30.
Två exempel på att använda neutronbackspridningsspektroskopi för att studera amyloidproteiner kommer att presenteras, ett i flytande tillstånd för att studera proteindynamik och ett i hydratiserat pulvertillstånd för att studera hydratiseringsvattendynamik. Det första exemplet gäller aggregering av lysozym i μm-sfärer (kallade partiklar) följt i realtid5, och det andra en jämförelse av vattendynamik i naturliga och aggregerade tillstånd av det humana proteinet tau31.
Lysozym är ett enzym som är involverat i immunförsvaret och består av 129 aminosyrarester. Lysozym kan bilda partiklar i deutererad buffert vid pD 10,5 och vid en temperatur på 90 °C. Med neutronspridning visade vi att tidsutvecklingen av massdiffusionskoefficienten för lysozym följer den enda exponentiella kinetiken för tioflavin T-fluorescens (en fluorescerande sond som används för att övervaka bildandet av amyloidkors-β mönster32), vilket indikerar att bildningspartikelformiga överbyggnader och kors-β mönster förekommer i ett enda steg med samma hastighet. Dessutom förblev den interna dynamiken konstant under hela aggregeringsprocessen, vilket kan förklaras antingen av en snabb konformationsförändring som inte kan observeras på NBS-instrument eller av frånvaron av signifikant förändring i proteinets interna energi vid aggregering.
Det humana proteinet tau är ett egenstört protein (IDP) som består av 441 aminosyror för den så kallade 2N4R-isoformen, som är särskilt involverad i Alzheimers sjukdom33. Med hjälp av neutronåterspridning på pulver av perdeutererat protein tau visade vi att hydratiseringsvattendynamiken ökar i fibertillståndet, med en högre population av vattenmolekyler som genomgår translationella rörelser. Resultatet tyder på att en ökning av hydreringsvattenentropi kan driva amyloidflimmer av tau.
Protocol
Representative Results
Discussion
Neutronspektroskopi är den enda metoden som gör det möjligt att undersöka den ensemble-genomsnittliga ps-ns-dynamiken hos proteinprover oavsett proteinets storlek eller lösningens komplexitet när deuteration används6. Specifikt, genom att undersöka självdiffusion av proteinaggregat i lösning, kan den hydrodynamiska storleken hos sådana sammansättningar entydigt bestämmas. Icke desto mindre begränsas metoden vanligen av det låga neutronflödet, vilket innebär långa förvärvstider …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna är tacksamma mot Michaela Zamponi vid Jülich Centre for Neutron Science vid Heinz Maier-Leibnitz Zentrum, Garching, Tyskland, för en del av neutronspridningsexperimenten som utförts på instrumentet SPHERES. Detta arbete har gynnats av verksamheten i konsortiet Deuteration Laboratory (DLAB) som finansieras av Europeiska unionen enligt kontrakten HPRI-2001-50065 och RII3-CT-2003-505925, och från UK Engineering and Physical Sciences Research Council (EPSRC)-finansierad verksamhet inom Institut Laue Langevin EMBL DLAB under bidrag GR/R99393/01 och EP/C015452/1. Stöd från Europeiska kommissionen inom sjunde ramprogrammet genom nyckelåtgärden: förstärkning av det europeiska området för forskningsverksamhet, forskningsinfrastruktur erkänns [kontrakt 226507 (NMI3)]. Kevin Pounot och Christian Beck tackar det federala ministeriet för utbildning och forskning (BMBF, bidragsnummer 05K19VTB) för finansiering av sina postdoktorala stipendier.
Materials
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 |
|
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
References
- Jacrot, B. Des neutrons pour la science: Histoire de l’Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer’s β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry – An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).

