Beredning och morfologisk analys av kycklingkraniala neurala vapencellkulturer
Summary
Detta mångsidiga protokoll beskriver isoleringen av premigrativa neurala vapenceller (NCC) genom excision av kraniala neurala veck från kycklingembryon. Vid plätering och inkubation uppstår migrerande NCC från neurala vikplantor, vilket möjliggör bedömning av cellmorfologi och migration i en förenklad 2D-miljö.
Abstract
Under ryggradsdjurens utveckling migrerar neurala vapenceller (NCC) i stor utsträckning och differentieras till olika celltyper som bidrar till strukturer som kraniofacialt skelett och det perifera nervsystemet. Även om det är viktigt att förstå NCC-migration i samband med ett 3D-embryo, underlättar isolering av migrerande celler i 2D-odling visualisering och funktionell karakterisering, vilket kompletterar embryonala studier. Detta protokoll demonstrerar en metod för att isolera kycklingkraniala neurala veck för att generera primära NCC-kulturer. Migrerande NCC kommer ut från neurala vikplantor pläterade på ett fibronektinbelagt substrat. Detta resulterar i spridda, vidhäftande NCC-populationer som kan bedömas genom färgning och kvantitativa morfologiska analyser. Denna förenklade kulturmetod är mycket anpassningsbar och kan kombineras med andra tekniker. Till exempel kan NCC-emigrations- och migrationsbeteenden utvärderas genom time-lapse-avbildning eller funktionellt frågas genom att inkludera hämmare eller experimentella manipuleringar av genuttryck (t.ex. DNA, morfolino eller CRISPR-elektroporering). På grund av sin mångsidighet ger denna metod ett kraftfullt system för att undersöka kranial NCC-utveckling.
Introduction
Neurala vapenceller (NCC) är en övergående cellpopulation i embryon från ryggradsdjur. NCC specificeras vid neuralplattans gränser och genomgår en epitel-till-mesenkymal övergång (EMT) för att migrera från dorsala neuralröret1. Efter EMT sprids NCC i stor utsträckning genom embryot, vilket i slutändan differentierar och bidrar till olika strukturer, inklusive kraniofacialt skelett, utflödeskanal i hjärtat och majoriteten av det perifera nervsystemet2. Förändringar i cellpolaritet, cytoskelett och vidhäftningsegenskaper ligger till grund för detta skifte från en premigrerande till en migrerande cellpopulation3. Att studera NCC EMT och migration ger insikter i grundläggande mekanismer för cellmotilitet och informerar om insatser för att förebygga och behandla fosterskador och cancermetastaser.
Medan in vivo-analys är avgörande för att förstå NCC-utvecklingsprocesser i ett embryonalt sammanhang, erbjuder in vitro-metoder visuell och fysisk tillgänglighet som underlättar ytterligare experimentella vägar. I en förenklad 2D-miljö kan NCC-morfologi, cytoskelettstrukturer och migrerat avstånd utvärderas. Dessutom kan effekterna av genetisk eller löslig faktorstörning på migrerande beteenden hos rörliga NCC analyseras 4,5,6,7,8,9,10. Dessutom kan isolerade premigrerande eller migrerande NCC samlas in, poolas och användas för metoder med hög genomströmning för att studera utvecklingsreglering av NCC genom proteomisk, transkriptomisk och epigenomisk profilering 7,11. Medan metoder finns tillgängliga för att förbereda kranial NCC från olika utvecklingsmodellorganismer12,13,14, visar den här artikeln mekaniken i tillvägagångssättet för dem som först lär sig att odla kranial NCC från kycklingembryon.
Det nuvarande protokollet beskriver en mångsidig teknik för beredning av NCC-kulturer av kycklingkranier (figur 1). Eftersom NCC migrerar lätt från explanterade neurala veck till ett odlingssubstrat, separerar kyckling-NCC naturligt från embryonal vävnad och primära kulturer genereras lätt. Eftersom NCC: er i mitten av hjärnan migrerar massor från kranialneurala veck (i motsats till den långvariga, cell-för-cell-delamineringen i stammen15), består dessa kulturer huvudsakligen av migrerande kraniala neurala vapenceller, med initial neural fold excision som ger en insamlingsmetod för premigrativa NCC. En grundläggande metod för dissekering och odling av kycklingkraniala neurala veck är detaljerad, och förslag på olika applikationer och variationer på denna metod erbjuds.
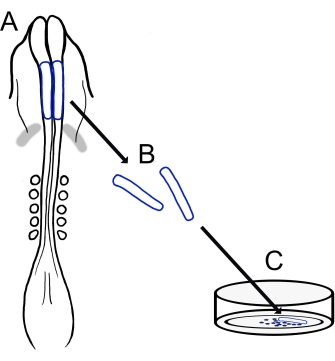
Figur 1: Schematisk översikt över chick cranial neural fold culture protocol. (A,B) Kraniala neurala veck (skisserade i blått) skärs ut från ett kycklingembryo med fem somiter (visas i dorsalvy i A). Grå band, hjärtmåne. (C) När de pläteras på fibronektin kommer migrerande neurala vapenceller ut från neurala veck och sprids på substratet. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
Tekniken som beskrivs här ger en anpassningsbar metod för att isolera kycklingneurala veck och plätera dem för att skapa kulturer av migrerande kraniala NCC. Dessa kulturer ger förenklade 2D-förhållanden för enkel analys av kyckling NCC-migration och morfologi som kan komplettera mer tekniskt utmanande i ovo-avbildningsmetoder24,25,26. Även om denna in vitro-metod är relativt enkel, beror konsekventa …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi tackar Corinne A. Fairchild och Katie L. Vermillion, som deltog i utvecklingen av vår version av chick cranial neural fold culture protocol.
Materials
| AxioObserver equipped with an LSM710 confocal scan head controlled by ZEN 3.0 SR software | Zeiss | Used alpha Plan-Apochromat 100x/1.46 Oil DIC M27 objective | |
| CaCl2 | Sigma-Aldrich | C3306 | |
| Chamber dishes (glass bottom, single or divided) | MatTek; Cell Vis | P35G-1.5-14-C (MatTek) X000NOJQGX (Cellvis) X000NOK1OJ (Cellvis) |
Single chamber 35 mm or 4 chamber 35 mm |
| Cover glass | Carolina Biological Supply Company | 633029, 633031, 633033, 633035, 633037 | circles, 0.13–0.17 mm thickness, available in 12-25 mm diameter |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | Alternative for L15 media |
| Egg incubator | Sportsman | 1502 | |
| FBS | Life Technologies | 10437-028 | |
| Fibronectin | Fisher Scientific | CB-40008A | |
| Filter paper | Whatman | grade 3MM chromatography | |
| Forceps (blunt) | Fisher Scientific; Thomas Scientific | 08-890 (Fisher);1141W97 (Thomas) | |
| Forceps (fine) | Fine Science Tools | 11252-20 | Dumont #5 |
| Image J | https://fiji.sc/ | Free image analysis software | |
| KCl | Sigma-Aldrich | P3911 | |
| KH2PO4 | Sigma-Aldrich | P0662 | |
| L15 media | Invitrogen | 11415064 | |
| L-glutamine | Invitrogen | 25030 | |
| Mounting Media (Vectashield or ProLong Gold) | Vector Laboratories; Thermofisher Scientific | H-1700 (Vectashield); P36930 (ProLong Gold) | |
| Na2HPO4 | Sigma-Aldrich | S9638 | |
| NaCl | Sigma-Aldrich | S9888 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Penicillin/streptomycin | Life Technologies | 15140-148 | 10,000 Units/mL Penicillin; 10,000 mg/mL Streptomycin |
| Petri Dishes | VWR (or similar) | 60 mm, 100 mm | |
| Phalloidin | Sigma-Aldrich | P1951 | multiple flurophores available |
| Pin holder | Fine Science Tools | 26016-12 | For tungsten needle (alternative for spring scissors) |
| Scissors (dissection) | Fine Science Tools | 14061-10 | |
| Spring Scissors | Fine Science Tools | 15000-08 | 2.5 mm cutting edge (alternative for tungsten needle) |
| Sylgard | Krayden | Sylgard 184 | |
| Syringe Filters | Sigma-Aldrich | SLGVM33RS | Millex-GV Syringe Filter Unit, 0.22 µm, PVDF, 33 mm, gamma sterilized |
| Tissue culture dishes | Sarstedt | 83-3900 | 35 mm culture dishes for bulk neural fold cultures |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Tungsten wire | Variety of sources | 0.01" diameter for tungsten needle (alternative for spring scissors) |
References
- Pla, P., Monsoro-Burq, A. H. The neural border: Induction, specification and maturation of the territory that generates neural crest cells. Developmental Biology. 444, 36-46 (2018).
- Tang, W., Bronner, M. E. Neural crest lineage analysis: From past to future trajectory. Development. 147 (20), (2021).
- Piacentino, M. L., Li, Y., Bronner, M. E. Epithelial-to-mesenchymal transition and different migration strategies as viewed from the neural crest. Current Opinion in Cell Biology. 66, 43-50 (2020).
- McLennan, R., et al. Neural crest cells bulldoze through the microenvironment using Aquaporin 1 to stabilize filopodia. Development. 147 (1), 185231 (2020).
- Carmona-Fontaine, C., et al. Complement fragment C3a controls mutual cell attraction during collective cell migration. Developmental Cell. 21 (6), 1026-1037 (2011).
- Giovannone, D., et al. Slits affect the timely migration of neural crest cells via robo receptor. Developmental Dynamics. 241 (8), 1274-1288 (2012).
- Vermillion, K. L., Lidberg, K. A., Gammill, L. S. Cytoplasmic protein methylation is essential for neural crest migration. Journal of Cell Biology. 204 (1), 95-109 (2014).
- Yang, X., Li, J., Zeng, W., Li, C., Mao, B. Elongator Protein 3 (Elp3) stabilizes Snail1 and regulates neural crest migration in Xenopus. Scientific Reports. 6 (1), 1-9 (2016).
- Gonzalez Malagon, S. G., et al. Glycogen synthase kinase 3 controls migration of the neural crest lineage in mouse and Xenopus. Nature Communications. 9 (1), 1-15 (2018).
- Bhattacharya, D., Azambuja, A. P., Simoes-Costa, M. Metabolic reprogramming promotes neural crest migration via yap/tead signaling. Developmental Cell. 53 (2), 199-211 (2020).
- Jacques-Fricke, B. T., et al. Profiling NSD3-dependent neural crest gene expression reveals known and novel candidate regulatory factors. Developmental Biology. 475, 118-130 (2021).
- Bronner-Fraser, M., García-Castro, M. Chapter 4 manipulations of neural crest cells or their migratory pathways. Methods in Cell Biology. 87, 75-96 (2008).
- Milet, C., Monsoro-Burq, A. H. Dissection of xenopus laevis neural crest for in vitro explant culture or in vivo transplantation. Journal of Visualized Experiments. (85), e51118 (2014).
- Malagon, S. G. G., et al. Dissection, culture and analysis of primary cranial neural crest cells from mouse for the study of neural crest cell delamination and migration. Journal of Visualized Experiments. (152), e60051 (2019).
- Theveneau, E., Mayor, R. Neural crest delamination and migration: From epithelium-to-mesenchyme transition to collective cell migration. Developmental Biology. 366 (1), 34-54 (2012).
- Conrad, G. W., Bee, J. A., Roche, S. M., Teillet, M. A. Fabrication of microscalpels by electrolysis of tungsten wire in a meniscus. Journal of Neuroscience Methods. 50 (1), 123-127 (1993).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88 (1), 49-92 (1951).
- Gammill, L. S., Jacques-Fricke, B., Roffers-Agarwal, J. Embryological and genetic manipulation of chick development. Methods in Molecular Biology. 1920, 75-97 (2019).
- Vandekerckhove, J., Deboben, A., Nassal, M., Wieland, T. The phalloidin binding site of F-actin. The EMBO Journal. 4 (11), 2815-2818 (1985).
- Bronner-Fraser, M. Analysis of the early stages of trunk neural crest migration in avian embryos using monoclonal antibody HNK-1. Developmental Biology. 115 (1), 44-55 (1986).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Soille, P., Vincent, L. Determining watersheds in digital pictures via flooding simulations. Visual Communications and Image Processing ’90: Fifth in a Series. (1360), 240-250 (1990).
- Haupt, A., Minc, N. How cells sense their own shape – mechanisms to probe cell geometry and their implications in cellular organization and function. Journal of Cell Science. 131 (6), (2018).
- Ezin, M., Fraser, S. Chapter 11 time-lapse imaging of the early avian embryo. Methods in Cell Biology. 87, 211-236 (2008).
- Kulesa, P. M., Bailey, C. M., Cooper, C., Fraser, S. E. In ovo live imaging of avian embryos. Cold Spring Harbor Protocols. 5 (6), (2010).
- McKinney, M. C., Kulesa, P. M. Live imaging of the neural crest cell epithelial-to-mesenchymal transition in the chick embryo. Methods in Molecular Biology. 2179, 107-114 (2021).
- Gustafson, C. M., Roffers-Agarwal, J., Gammill, L. S. Chick cranial neural crest cells release extracellular vesicles that are critical for their migration. Journal of Cell Science. , (2022).
- Williams, R., Sauka-Spengler, T. Ex ovo electroporation of early chicken embryos. STAR Protocols. 2 (2), 100424 (2021).
- Moulton, J. D. Using morpholinos to control gene expression. Current Protocols in Nucleic Acid Chemistry. 68 (1), 4-30 (2017).
- Gandhi, S., et al. A single-plasmid approach for genome editing coupled with long-term lineage analysis in chick embryos. Development. 148 (7), (2021).
- Williams, R. M., et al. Reconstruction of the Global Neural Crest Gene Regulatory Network In Vivo. Developmental Cell. 51 (2), 255-267 (2019).

