הכנה וניתוח מורפולוגי של תרביות תאי צמרת עצביים גולגולתיים של אפרוחים
Summary
פרוטוקול רב-תכליתי זה מתאר את הבידוד של תאי פסגה עצבית קדם-מיגרטיבית (NCCs) באמצעות כריתה של קפלים עצביים גולגולתיים מעוברי אפרוחים. עם הציפוי והדגירה, NCCs נודדים מגיחים מתוך קצוות עצביים, מה שמאפשר הערכה של מורפולוגיה של תאים ונדידה בסביבה דו-ממדית פשוטה.
Abstract
במהלך התפתחות בעלי חוליות, תאי פסגה עצביים (NCCs) נודדים באופן נרחב ומתמיינים לסוגי תאים שונים התורמים למבנים כמו השלד הקרניופציאלי ומערכת העצבים ההיקפית. בעוד שזה קריטי להבין את נדידת NCC בהקשר של עובר תלת-ממדי, בידוד תאים נודדים בתרבית דו-ממדית מקל על הדמיה ואפיון פונקציונלי, ומשלים מחקרים עובריים. הפרוטוקול הנוכחי מדגים שיטה לבידוד קפלים עצביים גולגולתיים של אפרוחים כדי ליצור תרביות NCC ראשוניות. NCCs נודדים מגיחים מתוך צמחי קפלים עצביים המצופים על מצע מצופה פיברונקטין. התוצאה היא אוכלוסיות NCC מפוזרות ודבקות שניתן להעריך על ידי כתמים וניתוחים מורפולוגיים כמותיים. גישה תרבותית פשוטה זו ניתנת להתאמה רבה וניתן לשלב אותה עם טכניקות אחרות. לדוגמה, הגירה של NCC והתנהגויות נדידה יכולות להיות מוערכות על ידי הדמיה בהילוך מהיר או על ידי שאילתה תפקודית על ידי הכללת מעכבים או מניפולציות ניסיוניות של ביטוי גנים (למשל, דנ”א, מורפולינו או אלקטרופורציה של קריספר). בגלל הרבגוניות שלה, שיטה זו מספקת מערכת רבת עוצמה לחקר התפתחות NCC גולגולתי.
Introduction
תאי פסגה עצביים (NCCs) הם אוכלוסיית תאים חולפת בעוברים בעלי חוליות. NCCs מוגדרים בגבולות הלוח העצבי ועוברים מעבר אפיתליאלי למזנכימלי (EMT) כדי לנדוד מהצינור העצבי הגבי1. לאחר EMT, NCCs מתפזרים באופן נרחב ברחבי העובר, ובסופו של דבר מבדילים ותורמים למבנים שונים, כולל השלד הקרניופציאלי, דרכי היציאה של הלב, ורוב מערכת העצבים ההיקפית2. שינויים בקוטביות התא, בשלד הציטוסקולרי ובתכונות ההידבקות עומדים בבסיס המעבר הזה מאוכלוסיית תאים נודדיםלאוכלוסיית תאים נודדים 3. חקר NCC EMT והגירה מספק תובנות על מנגנונים בסיסיים של תנועתיות תאים ומודיע על המאמצים למנוע ולטפל במומים מולדים וגרורות סרטניות.
בעוד שניתוח in vivo חיוני להבנת תהליכים התפתחותיים של NCC בהקשר עוברי, שיטות in vitro מציעות נגישות חזותית ופיזית המאפשרת אפיקים ניסיוניים נוספים. בסביבה דו-ממדית פשוטה, ניתן להעריך מורפולוגיה של NCC, מבנים ציטוסקטליים ומרחק שהועבר. יתר על כן, ניתן לנתח את ההשפעות של הפרעה גנטית או גורם מסיס על התנהגויות נודדות של NCCs תנועתיים 4,5,6,7,8,9,10. בנוסף, ניתן לאסוף, לאגד ולהשתמש ב-NCCs טרום-מבודדים או נודדים עבור מתודולוגיות בעלות תפוקה גבוהה כדי לחקור את הוויסות ההתפתחותי של NCCs באמצעות פרופילים פרוטאומיים, תעתיקיים ואפיגנומיים 7,11. בעוד שקיימות שיטות להכנת NCCs גולגולתיים מאורגניזמי מודל התפתחותיים שונים12,13,14, מאמר זה מדגים את המכניקה של הגישה עבור אלה שלומדים לראשונה לתרבית NCC גולגולתי מעוברי אפרוחים.
הפרוטוקול הנוכחי מתאר טכניקה רב-תכליתית להכנת תרביות NCC גולגולתיות של אפרוחים (איור 1). מאחר ש-NCCs נודדים בקלות מקפלים עצביים מושתלים אל מצע תרבית, NCCs של אפרוחים נפרדים באופן טבעי מרקמה עוברית, ותרביות ראשוניות נוצרות בקלות. כאשר NCCs של המוח האמצעי נודדים בהמוניהם מהקפלים העצביים הגולגולתיים (בניגוד לדלמינציה הממושכת, תא אחר תא, בתא המטען15), תרביות אלה מורכבות בעיקר מתאי צמרת עצבית גולגולתיים נודדים, כאשר כריתה ראשונית של קפל עצבי מספקת שיטת איסוף עבור NCCs קדם-מגרטוריים. מפורטת שיטה בסיסית לניתוח וגידול קפלים עצביים גולגולתיים של אפרוחים, ומוצעות הצעות ליישומים שונים ווריאציות על שיטה זו.
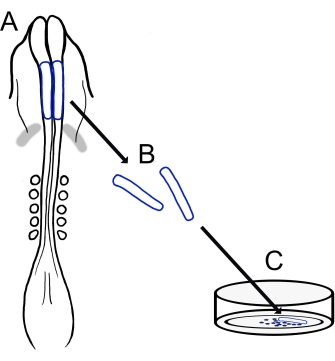
איור 1: סקירה סכמטית של פרוטוקול תרבית הקיפול העצבי הגולגולתי של אפרוחים. (A,B) קפלים עצביים גולגולתיים (מסומנים בכחול) נכרתים מעובר אפרוח עם חמישה סומיטים (מוצג במבט הגבי ב-A). פסים אפורים, סהר לבבי. (C) כאשר הם מצופים על פיברונקטין, תאי פסגה עצביים נודדים מגיחים מהקפלים העצביים ומתפזרים על המצע. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Protocol
Representative Results
Discussion
הטכניקה המתוארת כאן מספקת שיטה ניתנת להתאמה של בידוד קפלים עצביים של אפרוחים וציפוי שלהם כדי ליצור תרבויות של NCCs גולגולתיים נודדים. תרביות אלה מספקות תנאים דו-ממדיים פשוטים לניתוח קל של נדידת NCC של אפרוחים ומורפולוגיה שיכולים להשלים מאתגרים יותר מבחינה טכנית בשיטות הדמיה של ovo <sup class=…
Disclosures
The authors have nothing to disclose.
Acknowledgements
אנו מודים לקורין א. פיירצ’יילד וקייטי ל. ורמיליון, שהשתתפו בפיתוח הגרסה שלנו לפרוטוקול תרבית הקפל העצבי הגולגולתי של אפרוחים.
Materials
| AxioObserver equipped with an LSM710 confocal scan head controlled by ZEN 3.0 SR software | Zeiss | Used alpha Plan-Apochromat 100x/1.46 Oil DIC M27 objective | |
| CaCl2 | Sigma-Aldrich | C3306 | |
| Chamber dishes (glass bottom, single or divided) | MatTek; Cell Vis | P35G-1.5-14-C (MatTek) X000NOJQGX (Cellvis) X000NOK1OJ (Cellvis) |
Single chamber 35 mm or 4 chamber 35 mm |
| Cover glass | Carolina Biological Supply Company | 633029, 633031, 633033, 633035, 633037 | circles, 0.13–0.17 mm thickness, available in 12-25 mm diameter |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | Alternative for L15 media |
| Egg incubator | Sportsman | 1502 | |
| FBS | Life Technologies | 10437-028 | |
| Fibronectin | Fisher Scientific | CB-40008A | |
| Filter paper | Whatman | grade 3MM chromatography | |
| Forceps (blunt) | Fisher Scientific; Thomas Scientific | 08-890 (Fisher);1141W97 (Thomas) | |
| Forceps (fine) | Fine Science Tools | 11252-20 | Dumont #5 |
| Image J | https://fiji.sc/ | Free image analysis software | |
| KCl | Sigma-Aldrich | P3911 | |
| KH2PO4 | Sigma-Aldrich | P0662 | |
| L15 media | Invitrogen | 11415064 | |
| L-glutamine | Invitrogen | 25030 | |
| Mounting Media (Vectashield or ProLong Gold) | Vector Laboratories; Thermofisher Scientific | H-1700 (Vectashield); P36930 (ProLong Gold) | |
| Na2HPO4 | Sigma-Aldrich | S9638 | |
| NaCl | Sigma-Aldrich | S9888 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Penicillin/streptomycin | Life Technologies | 15140-148 | 10,000 Units/mL Penicillin; 10,000 mg/mL Streptomycin |
| Petri Dishes | VWR (or similar) | 60 mm, 100 mm | |
| Phalloidin | Sigma-Aldrich | P1951 | multiple flurophores available |
| Pin holder | Fine Science Tools | 26016-12 | For tungsten needle (alternative for spring scissors) |
| Scissors (dissection) | Fine Science Tools | 14061-10 | |
| Spring Scissors | Fine Science Tools | 15000-08 | 2.5 mm cutting edge (alternative for tungsten needle) |
| Sylgard | Krayden | Sylgard 184 | |
| Syringe Filters | Sigma-Aldrich | SLGVM33RS | Millex-GV Syringe Filter Unit, 0.22 µm, PVDF, 33 mm, gamma sterilized |
| Tissue culture dishes | Sarstedt | 83-3900 | 35 mm culture dishes for bulk neural fold cultures |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Tungsten wire | Variety of sources | 0.01" diameter for tungsten needle (alternative for spring scissors) |
References
- Pla, P., Monsoro-Burq, A. H. The neural border: Induction, specification and maturation of the territory that generates neural crest cells. Developmental Biology. 444, 36-46 (2018).
- Tang, W., Bronner, M. E. Neural crest lineage analysis: From past to future trajectory. Development. 147 (20), (2021).
- Piacentino, M. L., Li, Y., Bronner, M. E. Epithelial-to-mesenchymal transition and different migration strategies as viewed from the neural crest. Current Opinion in Cell Biology. 66, 43-50 (2020).
- McLennan, R., et al. Neural crest cells bulldoze through the microenvironment using Aquaporin 1 to stabilize filopodia. Development. 147 (1), 185231 (2020).
- Carmona-Fontaine, C., et al. Complement fragment C3a controls mutual cell attraction during collective cell migration. Developmental Cell. 21 (6), 1026-1037 (2011).
- Giovannone, D., et al. Slits affect the timely migration of neural crest cells via robo receptor. Developmental Dynamics. 241 (8), 1274-1288 (2012).
- Vermillion, K. L., Lidberg, K. A., Gammill, L. S. Cytoplasmic protein methylation is essential for neural crest migration. Journal of Cell Biology. 204 (1), 95-109 (2014).
- Yang, X., Li, J., Zeng, W., Li, C., Mao, B. Elongator Protein 3 (Elp3) stabilizes Snail1 and regulates neural crest migration in Xenopus. Scientific Reports. 6 (1), 1-9 (2016).
- Gonzalez Malagon, S. G., et al. Glycogen synthase kinase 3 controls migration of the neural crest lineage in mouse and Xenopus. Nature Communications. 9 (1), 1-15 (2018).
- Bhattacharya, D., Azambuja, A. P., Simoes-Costa, M. Metabolic reprogramming promotes neural crest migration via yap/tead signaling. Developmental Cell. 53 (2), 199-211 (2020).
- Jacques-Fricke, B. T., et al. Profiling NSD3-dependent neural crest gene expression reveals known and novel candidate regulatory factors. Developmental Biology. 475, 118-130 (2021).
- Bronner-Fraser, M., García-Castro, M. Chapter 4 manipulations of neural crest cells or their migratory pathways. Methods in Cell Biology. 87, 75-96 (2008).
- Milet, C., Monsoro-Burq, A. H. Dissection of xenopus laevis neural crest for in vitro explant culture or in vivo transplantation. Journal of Visualized Experiments. (85), e51118 (2014).
- Malagon, S. G. G., et al. Dissection, culture and analysis of primary cranial neural crest cells from mouse for the study of neural crest cell delamination and migration. Journal of Visualized Experiments. (152), e60051 (2019).
- Theveneau, E., Mayor, R. Neural crest delamination and migration: From epithelium-to-mesenchyme transition to collective cell migration. Developmental Biology. 366 (1), 34-54 (2012).
- Conrad, G. W., Bee, J. A., Roche, S. M., Teillet, M. A. Fabrication of microscalpels by electrolysis of tungsten wire in a meniscus. Journal of Neuroscience Methods. 50 (1), 123-127 (1993).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88 (1), 49-92 (1951).
- Gammill, L. S., Jacques-Fricke, B., Roffers-Agarwal, J. Embryological and genetic manipulation of chick development. Methods in Molecular Biology. 1920, 75-97 (2019).
- Vandekerckhove, J., Deboben, A., Nassal, M., Wieland, T. The phalloidin binding site of F-actin. The EMBO Journal. 4 (11), 2815-2818 (1985).
- Bronner-Fraser, M. Analysis of the early stages of trunk neural crest migration in avian embryos using monoclonal antibody HNK-1. Developmental Biology. 115 (1), 44-55 (1986).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Soille, P., Vincent, L. Determining watersheds in digital pictures via flooding simulations. Visual Communications and Image Processing ’90: Fifth in a Series. (1360), 240-250 (1990).
- Haupt, A., Minc, N. How cells sense their own shape – mechanisms to probe cell geometry and their implications in cellular organization and function. Journal of Cell Science. 131 (6), (2018).
- Ezin, M., Fraser, S. Chapter 11 time-lapse imaging of the early avian embryo. Methods in Cell Biology. 87, 211-236 (2008).
- Kulesa, P. M., Bailey, C. M., Cooper, C., Fraser, S. E. In ovo live imaging of avian embryos. Cold Spring Harbor Protocols. 5 (6), (2010).
- McKinney, M. C., Kulesa, P. M. Live imaging of the neural crest cell epithelial-to-mesenchymal transition in the chick embryo. Methods in Molecular Biology. 2179, 107-114 (2021).
- Gustafson, C. M., Roffers-Agarwal, J., Gammill, L. S. Chick cranial neural crest cells release extracellular vesicles that are critical for their migration. Journal of Cell Science. , (2022).
- Williams, R., Sauka-Spengler, T. Ex ovo electroporation of early chicken embryos. STAR Protocols. 2 (2), 100424 (2021).
- Moulton, J. D. Using morpholinos to control gene expression. Current Protocols in Nucleic Acid Chemistry. 68 (1), 4-30 (2017).
- Gandhi, S., et al. A single-plasmid approach for genome editing coupled with long-term lineage analysis in chick embryos. Development. 148 (7), (2021).
- Williams, R. M., et al. Reconstruction of the Global Neural Crest Gene Regulatory Network In Vivo. Developmental Cell. 51 (2), 255-267 (2019).

