Forberedelse og morfologisk analyse af chick kraniale neurale crestcellekulturer
Summary
Denne alsidige protokol beskriver isoleringen af præmigratoriske neurale kamceller (NCC’er) gennem udskæring af kraniale neurale folder fra kyllingembryoner. Ved plettering og inkubation kommer migrerende NCC’er ud af neurale foldeksplanter, hvilket giver mulighed for vurdering af cellemorfologi og migration i et forenklet 2D-miljø.
Abstract
Under hvirveldyrudvikling migrerer neurale kamceller (NCC’er) i vid udstrækning og differentierer sig til forskellige celletyper, der bidrager til strukturer som det kraniofaciale skelet og det perifere nervesystem. Selvom det er afgørende at forstå NCC-migration i forbindelse med et 3D-embryo, letter isolering af migrerende celler i 2D-kultur visualisering og funktionel karakterisering, der supplerer embryonale undersøgelser. Den nuværende protokol demonstrerer en metode til isolering af chick kraniale neurale folder for at generere primære NCC-kulturer. Migrerende NCC’er kommer ud af neurale fold explants belagt på et fibronectinbelagt substrat. Dette resulterer i spredte, vedhængende NCC-populationer, der kan vurderes ved farvning og kvantitative morfologiske analyser. Denne forenklede kulturtilgang er meget tilpasningsdygtig og kan kombineres med andre teknikker. For eksempel kan NCC-emigration og migrerende adfærd evalueres ved time-lapse-billeddannelse eller funktionelt forespørges ved at inkludere hæmmere eller eksperimentelle manipulationer af genekspression (f.eks. DNA, morpholino eller CRISPR-elektroporation). På grund af sin alsidighed giver denne metode et kraftfuldt system til undersøgelse af kranial NCC-udvikling.
Introduction
Neurale kamceller (NCC’er) er en forbigående cellepopulation i hvirveldyrembryoner. NCC’er specificeres ved grænserne af den neurale plade og gennemgår en epitel-til-mesenkymal overgang (EMT) for at migrere fra dorsalneuralrøret1. Efter EMT spredes NCC’er i vid udstrækning gennem embryoet og differentierer og bidrager i sidste ende til forskellige strukturer, herunder kraniofacial skelet, hjertets udstrømningskanal og størstedelen af det perifere nervesystem2. Ændringer i cellepolaritet, cytoskelettet og vedhæftningsegenskaber ligger til grund for dette skift fra en præmigrativ til en migrerende cellepopulation3. At studere NCC EMT og migration giver indsigt i grundlæggende mekanismer for cellemotilitet og informerer indsatsen for at forebygge og behandle fødselsdefekter og kræftmetastase.
Mens in vivo-analyse er afgørende for at forstå NCC-udviklingsprocesser i en embryonal sammenhæng, tilbyder in vitro-metoder visuel og fysisk tilgængelighed, der letter yderligere eksperimentelle veje. I et forenklet 2D-miljø kan NCC-morfologi, cytoskeletale strukturer og migreret afstand evalueres. Desuden kan virkningerne af genetisk eller opløselig faktorforstyrrelse på migrerende adfærd hos bevægelige NCC’er analyseres 4,5,6,7,8,9,10. Derudover kan isolerede præmigratoriske eller migrerende NCC’er indsamles, samles og bruges til metoder med høj kapacitet til at studere udviklingsreguleringen af NCC’er gennem proteomisk, transkriptomisk og epigenomisk profilering 7,11. Mens der findes metoder til fremstilling af kraniale NCC’er fra forskellige udviklingsmodelorganismer12,13,14, demonstrerer denne artikel mekanikken i tilgangen for dem, der først lærer at dyrke kranial NCC fra kyllingembryoner.
Den nuværende protokol beskriver en alsidig teknik til fremstilling af chick kraniale NCC-kulturer (figur 1). Fordi NCC’er migrerer let fra explantede neurale folder på et kultursubstrat, adskilles chick NCC’er naturligt fra embryonalt væv, og primære kulturer genereres let. Da NCC’er i mellemhjernen migrerer en masse fra kraniale neurale folder (i modsætning til den langvarige cell-for-celle-delaminering i bagagerummet15), består disse kulturer hovedsageligt af migrerende kraniale neurale kamceller, med indledende neurale foldudskæringer, der giver en indsamlingsmetode til præmigratoriske NCC’er. En grundlæggende metode til dissekering og dyrkning af chick kraniale neurale folder er detaljeret, og der tilbydes forslag til forskellige applikationer og variationer af denne metode.
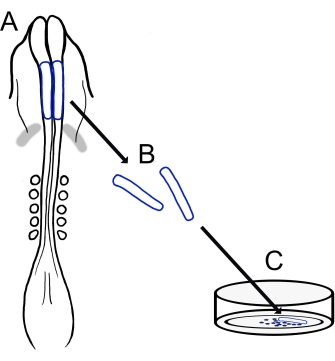
Figur 1: Skematisk oversigt over chick cranial neural fold kultur protokol. (A,B) Kraniale neurale folder (skitseret med blåt) udskæres fra et kyllingembryo med fem somitter (vist i dorsal visning i A). Grå bånd, hjertehalvmåne. (C) Når de er belagt med fibronectin, kommer migrerende neurale kamceller ud af de neurale folder og spredes på substratet. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
Teknikken beskrevet her giver en tilpasningsdygtig metode til at isolere chick neurale folder og plating dem for at skabe kulturer af migrerende kraniale NCC’er. Disse kulturer giver forenklede 2D-betingelser for nem analyse af chick NCC-migration og morfologi, der kan supplere mere teknisk udfordrende i ovo-billeddannelsesmetoder 24,25,26. Selv om denne in vitro-metode er forholdsvis enkel, afhænger de konsis…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi takker Corinne A. Fairchild og Katie L. Vermillion, som deltog i udviklingen af vores version af chick cranial neural fold culture protocol.
Materials
| AxioObserver equipped with an LSM710 confocal scan head controlled by ZEN 3.0 SR software | Zeiss | Used alpha Plan-Apochromat 100x/1.46 Oil DIC M27 objective | |
| CaCl2 | Sigma-Aldrich | C3306 | |
| Chamber dishes (glass bottom, single or divided) | MatTek; Cell Vis | P35G-1.5-14-C (MatTek) X000NOJQGX (Cellvis) X000NOK1OJ (Cellvis) |
Single chamber 35 mm or 4 chamber 35 mm |
| Cover glass | Carolina Biological Supply Company | 633029, 633031, 633033, 633035, 633037 | circles, 0.13–0.17 mm thickness, available in 12-25 mm diameter |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | Alternative for L15 media |
| Egg incubator | Sportsman | 1502 | |
| FBS | Life Technologies | 10437-028 | |
| Fibronectin | Fisher Scientific | CB-40008A | |
| Filter paper | Whatman | grade 3MM chromatography | |
| Forceps (blunt) | Fisher Scientific; Thomas Scientific | 08-890 (Fisher);1141W97 (Thomas) | |
| Forceps (fine) | Fine Science Tools | 11252-20 | Dumont #5 |
| Image J | https://fiji.sc/ | Free image analysis software | |
| KCl | Sigma-Aldrich | P3911 | |
| KH2PO4 | Sigma-Aldrich | P0662 | |
| L15 media | Invitrogen | 11415064 | |
| L-glutamine | Invitrogen | 25030 | |
| Mounting Media (Vectashield or ProLong Gold) | Vector Laboratories; Thermofisher Scientific | H-1700 (Vectashield); P36930 (ProLong Gold) | |
| Na2HPO4 | Sigma-Aldrich | S9638 | |
| NaCl | Sigma-Aldrich | S9888 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Penicillin/streptomycin | Life Technologies | 15140-148 | 10,000 Units/mL Penicillin; 10,000 mg/mL Streptomycin |
| Petri Dishes | VWR (or similar) | 60 mm, 100 mm | |
| Phalloidin | Sigma-Aldrich | P1951 | multiple flurophores available |
| Pin holder | Fine Science Tools | 26016-12 | For tungsten needle (alternative for spring scissors) |
| Scissors (dissection) | Fine Science Tools | 14061-10 | |
| Spring Scissors | Fine Science Tools | 15000-08 | 2.5 mm cutting edge (alternative for tungsten needle) |
| Sylgard | Krayden | Sylgard 184 | |
| Syringe Filters | Sigma-Aldrich | SLGVM33RS | Millex-GV Syringe Filter Unit, 0.22 µm, PVDF, 33 mm, gamma sterilized |
| Tissue culture dishes | Sarstedt | 83-3900 | 35 mm culture dishes for bulk neural fold cultures |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Tungsten wire | Variety of sources | 0.01" diameter for tungsten needle (alternative for spring scissors) |
References
- Pla, P., Monsoro-Burq, A. H. The neural border: Induction, specification and maturation of the territory that generates neural crest cells. Developmental Biology. 444, 36-46 (2018).
- Tang, W., Bronner, M. E. Neural crest lineage analysis: From past to future trajectory. Development. 147 (20), (2021).
- Piacentino, M. L., Li, Y., Bronner, M. E. Epithelial-to-mesenchymal transition and different migration strategies as viewed from the neural crest. Current Opinion in Cell Biology. 66, 43-50 (2020).
- McLennan, R., et al. Neural crest cells bulldoze through the microenvironment using Aquaporin 1 to stabilize filopodia. Development. 147 (1), 185231 (2020).
- Carmona-Fontaine, C., et al. Complement fragment C3a controls mutual cell attraction during collective cell migration. Developmental Cell. 21 (6), 1026-1037 (2011).
- Giovannone, D., et al. Slits affect the timely migration of neural crest cells via robo receptor. Developmental Dynamics. 241 (8), 1274-1288 (2012).
- Vermillion, K. L., Lidberg, K. A., Gammill, L. S. Cytoplasmic protein methylation is essential for neural crest migration. Journal of Cell Biology. 204 (1), 95-109 (2014).
- Yang, X., Li, J., Zeng, W., Li, C., Mao, B. Elongator Protein 3 (Elp3) stabilizes Snail1 and regulates neural crest migration in Xenopus. Scientific Reports. 6 (1), 1-9 (2016).
- Gonzalez Malagon, S. G., et al. Glycogen synthase kinase 3 controls migration of the neural crest lineage in mouse and Xenopus. Nature Communications. 9 (1), 1-15 (2018).
- Bhattacharya, D., Azambuja, A. P., Simoes-Costa, M. Metabolic reprogramming promotes neural crest migration via yap/tead signaling. Developmental Cell. 53 (2), 199-211 (2020).
- Jacques-Fricke, B. T., et al. Profiling NSD3-dependent neural crest gene expression reveals known and novel candidate regulatory factors. Developmental Biology. 475, 118-130 (2021).
- Bronner-Fraser, M., García-Castro, M. Chapter 4 manipulations of neural crest cells or their migratory pathways. Methods in Cell Biology. 87, 75-96 (2008).
- Milet, C., Monsoro-Burq, A. H. Dissection of xenopus laevis neural crest for in vitro explant culture or in vivo transplantation. Journal of Visualized Experiments. (85), e51118 (2014).
- Malagon, S. G. G., et al. Dissection, culture and analysis of primary cranial neural crest cells from mouse for the study of neural crest cell delamination and migration. Journal of Visualized Experiments. (152), e60051 (2019).
- Theveneau, E., Mayor, R. Neural crest delamination and migration: From epithelium-to-mesenchyme transition to collective cell migration. Developmental Biology. 366 (1), 34-54 (2012).
- Conrad, G. W., Bee, J. A., Roche, S. M., Teillet, M. A. Fabrication of microscalpels by electrolysis of tungsten wire in a meniscus. Journal of Neuroscience Methods. 50 (1), 123-127 (1993).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88 (1), 49-92 (1951).
- Gammill, L. S., Jacques-Fricke, B., Roffers-Agarwal, J. Embryological and genetic manipulation of chick development. Methods in Molecular Biology. 1920, 75-97 (2019).
- Vandekerckhove, J., Deboben, A., Nassal, M., Wieland, T. The phalloidin binding site of F-actin. The EMBO Journal. 4 (11), 2815-2818 (1985).
- Bronner-Fraser, M. Analysis of the early stages of trunk neural crest migration in avian embryos using monoclonal antibody HNK-1. Developmental Biology. 115 (1), 44-55 (1986).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Soille, P., Vincent, L. Determining watersheds in digital pictures via flooding simulations. Visual Communications and Image Processing ’90: Fifth in a Series. (1360), 240-250 (1990).
- Haupt, A., Minc, N. How cells sense their own shape – mechanisms to probe cell geometry and their implications in cellular organization and function. Journal of Cell Science. 131 (6), (2018).
- Ezin, M., Fraser, S. Chapter 11 time-lapse imaging of the early avian embryo. Methods in Cell Biology. 87, 211-236 (2008).
- Kulesa, P. M., Bailey, C. M., Cooper, C., Fraser, S. E. In ovo live imaging of avian embryos. Cold Spring Harbor Protocols. 5 (6), (2010).
- McKinney, M. C., Kulesa, P. M. Live imaging of the neural crest cell epithelial-to-mesenchymal transition in the chick embryo. Methods in Molecular Biology. 2179, 107-114 (2021).
- Gustafson, C. M., Roffers-Agarwal, J., Gammill, L. S. Chick cranial neural crest cells release extracellular vesicles that are critical for their migration. Journal of Cell Science. , (2022).
- Williams, R., Sauka-Spengler, T. Ex ovo electroporation of early chicken embryos. STAR Protocols. 2 (2), 100424 (2021).
- Moulton, J. D. Using morpholinos to control gene expression. Current Protocols in Nucleic Acid Chemistry. 68 (1), 4-30 (2017).
- Gandhi, S., et al. A single-plasmid approach for genome editing coupled with long-term lineage analysis in chick embryos. Development. 148 (7), (2021).
- Williams, R. M., et al. Reconstruction of the Global Neural Crest Gene Regulatory Network In Vivo. Developmental Cell. 51 (2), 255-267 (2019).

