माइकोहेटरोट्रॉफ़िक पौधों में एंडोफाइटिक कवक की पहचान करने के लिए अलगाव, लक्षण वर्णन और कुल डीएनए निष्कर्षण
Summary
वर्तमान लेख का उद्देश्य पौधे से जुड़े एंडोफाइटिक कवक के अलगाव के लिए विस्तृत और पर्याप्त प्रोटोकॉल प्रदान करना है, आइसोलेट्स का दीर्घकालिक संरक्षण, रूपात्मक लक्षण वर्णन, और बाद में आणविक पहचान और मेटागेनोमिक विश्लेषण के लिए कुल डीएनए निष्कर्षण।
Abstract
माइकोहेटरोट्रॉफ़िक पौधे माइकोरिज़ल निर्भरता के सबसे चरम रूपों में से एक प्रस्तुत करते हैं, जो पूरी तरह से अपनी ऑटोट्रॉफ़िक क्षमता खो चुके हैं। किसी भी अन्य महत्वपूर्ण संसाधन के रूप में आवश्यक, कवक जिसके साथ ये पौधे घनिष्ठ रूप से जुड़ते हैं, उनके लिए आवश्यक हैं। इसलिए, मायकोहेटरोट्रॉफ़िक प्रजातियों का अध्ययन करने में कुछ सबसे प्रासंगिक तकनीकें हैं जो संबंधित कवक की जांच को सक्षम करती हैं, विशेष रूप से जड़ों और भूमिगत अंगों में रहने वाले। इस संदर्भ में, संस्कृति-निर्भर और संस्कृति-स्वतंत्र एंडोफाइटिक कवक की पहचान करने की तकनीक आमतौर पर लागू की जाती है। फंगल एंडोफाइट्स को अलग करना उन्हें रूपात्मक रूप से पहचानने, उनकी विविधता का विश्लेषण करने और आर्किड बीजों के सहजीवी अंकुरण में अनुप्रयोगों के लिए इनोकुला बनाए रखने का एक साधन प्रदान करता है। हालांकि, यह ज्ञात है कि पौधों के ऊतकों में रहने वाले गैर-खेती योग्य कवक की एक विशाल विविधता है। इस प्रकार, संस्कृति-स्वतंत्र आणविक पहचान तकनीक प्रजातियों की विविधता और बहुतायत का एक व्यापक आवरण प्रदान करती है। इस लेख का उद्देश्य दो जांच प्रक्रियाओं को शुरू करने के लिए आवश्यक पद्धतिगत समर्थन प्रदान करना है: एक संस्कृति-निर्भर और एक स्वतंत्र। संस्कृति-निर्भर प्रोटोकॉल के बारे में, संग्रह स्थलों से प्रयोगशाला सुविधाओं तक पौधों के नमूनों को इकट्ठा करने और बनाए रखने की प्रक्रियाएं विस्तृत हैं, साथ ही माइकोहेटरोट्रॉफ़िक पौधों के भूमिगत और हवाई अंगों से फिलामेंटस कवक को अलग करने के साथ-साथ, आइसोलेट्स का संग्रह रखते हुए, स्लाइड कल्चर पद्धति द्वारा रूपात्मक रूप से हाइपहे की विशेषता रखते हुए, और कुल डीएनए निष्कर्षण द्वारा कवक की आणविक पहचान। संस्कृति-स्वतंत्र पद्धतियों को शामिल करते हुए, विस्तृत प्रक्रियाओं में एक वाणिज्यिक किट का उपयोग करके मेटागेनोमिक विश्लेषण और एक्लोरोफिलस पौधों के अंगों से कुल डीएनए निष्कर्षण के लिए पौधों के नमूने एकत्र करना शामिल है। अंत में, निरंतरता प्रोटोकॉल (जैसे, पोलीमरेज़ चेन रिएक्शन [पीसीआर], अनुक्रमण) भी विश्लेषण के लिए सुझाव दिए जाते हैं, और तकनीक यहां प्रस्तुत की जाती हैं।
Introduction
एंडोफाइटिक कवक, परिभाषा के अनुसार, वे हैं जो अगोचर संक्रमणों में पौधे के अंगों और ऊतकों के आंतरिक भाग में रहते हैं (यानी, उनके मेजबान को नुकसान पहुंचाए बिना)1,2. ये कवक तटस्थ या लाभकारी मेजबान पौधों के साथ बातचीत कर सकते हैं, रोगजनकों और प्रतिकूल पर्यावरणीय परिस्थितियों के प्रतिरोध प्रदान कर सकते हैं, और पौधे के लिए लाभकारी यौगिकों के संश्लेषण में योगदान कर सकते हैं (जैसे, विकास कारक और अन्य फाइटोहोर्मोन)1,3. माइकोरिज़ल एंडोफाइट्स कवक हैं जो पौधे के साथ माइकोरिज़ल संघों को स्थापित करते हैं, पोषक तत्व हस्तांतरण में भाग लेते हैं4. ऑर्किडेसी में, माइकोरिज़ल एंडोफाइट्स के साथ बातचीत प्रजातियों के विशाल बहुमत में बीज अंकुरण के लिए मौलिक है, और परिवार में सभी पौधों में अंकुर स्थापना5. ऐसे संदर्भों में, मायकोहेटेरोट्रॉफ़िक ऑर्किड अपने माइकोरिज़ल भागीदारों के बारे में कुल निर्भरता के मामले का प्रतिनिधित्व करते हैं, क्योंकि वे अपने पूरे जीवन चक्र के दौरान इन कवक द्वारा खनिज पोषक तत्वों और कार्बन यौगिकोंके हस्तांतरण पर निर्भर करते हैं। इसलिए, माइकोहेटरोट्रॉफ़िक जीवन रणनीतियों की जांच करते समय कवक को जोड़ने का अलगाव और पहचान एक मौलिक आधार है। इसके अलावा, बहुत कम mycoheterotrophic पौधों या यहां तक कि इन कवक 7,8 की वास्तविक विविधता में कवक endophytes की भूमिकाओं के बारे में जाना जाता है.
एंडोफाइटिक कवक की जांच विभिन्न तकनीकों के माध्यम से आयोजित की जा सकती है, जिसे पारंपरिक रूप से संस्कृति-स्वतंत्र या निर्भर के रूप में वर्णित किया गया है, उदाहरण के लिए: (ए) प्रत्यक्ष अवलोकन, (बी) फंगल अलगाव और रूपात्मक और / या आणविक पहचान, और (सी) पौधे के ऊतकों और आणविक पहचान9 का कुल डीएनए निष्कर्षण। प्रत्यक्ष अवलोकन (ए) में, एंडोफाइटिक कवक की जांच की जा सकती है, जबकि अभी भी प्रकाश या इलेक्ट्रॉन माइक्रोस्कोपी9 द्वारा पौधों की कोशिकाओं और ऊतकों के इंटीरियर में है, क्योंकि विभिन्न माइक्रोस्कोपी प्रोटोकॉल पेना-पासोस एट अल.10द्वारा विस्तृत हैं। अलगाव विधियों (बी) द्वारा, फंगल एंडोफाइट्स को उनकी कॉलोनियों, हाइपहे और प्रजनन या प्रतिरोध संरचना आकृति विज्ञान के अनुसार चित्रित किया जा सकता है। इसके अलावा, अलगाव तकनीकों के माध्यम से, डीएनए निष्कर्षण, आणविक पहचान अनुक्रमों (बारकोड या उंगलियों के निशान), औरअनुक्रमण 11 के प्रवर्धन के माध्यम से आइसोलेट्स की आणविक पहचान का संचालन करना संभव है। उत्तरार्द्ध तकनीक (सी) डीएनए निष्कर्षण प्रति एंडोफाइटिक कवक की आणविक पहचान को सक्षम बनाता है, जबकि पौधे के ऊतकों (मेटाबारकोडिंग) के इंटीरियर में, पुस्तकालय की तैयारी औरअनुक्रमण 12 के बाद।
इसके अलावा, फंगल आइसोलेट्स को सहजीवी अंकुरण परीक्षणों में लागू किया जा सकता है, ऑटोट्रॉफिक या मायकोहेटेरोट्रॉफ़िक ऑर्किड से बीज का उपयोग करके। इस तरह के एक आवेदन का एक उदाहरण सिस्टी एट अल.13 द्वारा की गई जांच है, जो पोगोनियोप्सिस शेंकी, एक मायकोहेटेरोट्रॉफ़िक ऑर्किड में प्रोटोकॉर्म विकास के अंकुरण और प्रारंभिक चरणों का वर्णन करता है, इसके कुछ आइसोलेट्स के सहयोग से, जिसमें गैर-माइकोरिज़ल एंडोफाइटिक कवक शामिल हैं। लागू सहजीवी अंकुरण प्रोटोकॉल विस्तृत है और पेना-पासोस एट अल.10 द्वारा एक वीडियो में प्रस्तुत किया गया है। विभिन्न पौधों के अंगों के साथ मिलकर कवक को अलग करने से पौधे-कवक इंटरैक्शन की प्रकृति के बारे में विविध जांच केंद्रित होती है (उदाहरण के लिए, एसोसिएशन के पारिस्थितिक या शारीरिक पहलुओं को समझने के लिए, साथ ही कवक से पौधे तक पोषक तत्व हस्तांतरण में पूछताछ)9।
धारा 1 में प्रस्तुत पद्धतियां भूमिगत अंग के नमूनों के संग्रह पर आधारित हैं, क्योंकि ये अंग संग्रह में सबसे अधिक कठिनाइयां पेश करते हैं, और वे प्रमुख रुचि के हैं क्योंकि माइकोरिज़ल एंडोफाइट्स उन्हें उपनिवेश बनाते हैं। हालांकि, दोनों शामिल प्रोटोकॉल (चरण 1.1 और 1.2) अन्य मायकोहेट्रोट्रॉफ़िक पौधों के अंगों (जैसे, प्रकंद, पुष्प उपजी और फल) पर लागू किए जा सकते हैं। चरण 1.1 में वर्णित संग्रह पद्धति को रूपात्मक लक्षण वर्णन (धारा 4 और 5) और/या अलग पहचान (धारा 6) के लिए कुल डीएनए निष्कर्षण के लिए एंडोफाइटिक कवक (धारा 2) को अलग करने के लिए नामित किया गया है। दूसरी ओर, चरण 1.2 में वर्णित संग्रह पद्धति विशेष रूप से मेटाबारकोडिंग तकनीकों (धारा 7) के लिए पौधे के ऊतकों के कुल डीएनए निष्कर्षण को सौंपी गई है। धारा 3 में, फिलामेंटस कवक भंडारण और संरक्षण के लिए चार तरीके प्रस्तुत किए गए हैं, दो अल्पकालिक भंडारण (3-6 महीने) के लिए और अन्य दो दीर्घकालिक भंडारण (>1 वर्ष) के लिए पर्याप्त हैं। रूपात्मक लक्षण वर्णन (खंड 4 और 5) इसे सुदृढ़ करने और फंगल मैक्रो- और माइक्रोमोर्फोलॉजी पर महत्वपूर्ण जानकारी प्रदान करने के लिए आणविक पहचान से जुड़ा हो सकता है। चित्रा 1 इसके बाद वर्णित सामूहिक पद्धतियों को सारांशित करता है।
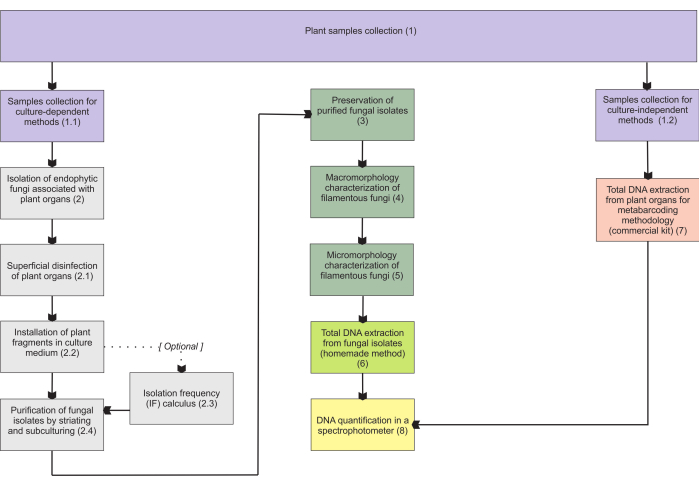
चित्रा 1: प्रस्तुत विधियों का योजनाबद्ध सारांश। “संस्कृति-निर्भर और -स्वतंत्र पद्धतियों द्वारा पौधे का संग्रह और फंगल अलगाव, संरक्षण और आणविक पहचान”। कृपया इस चित्र का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
Protocol
Representative Results
Discussion
पौधों के नमूनों की सतही विघटन प्रस्तुत प्रोटोकॉल में सबसे महत्वपूर्ण चरणों में से एक है। अंतिम धोने से बूंदों के साथ पीडीए व्यंजनों में कोई संदूषण अत्यधिक वांछनीय नहीं है। बैक्टीरिया अक्सर अलगाव व्य…
Disclosures
The authors have nothing to disclose.
Acknowledgements
हम FAPESP (2015/26479-6) और CNPq (447453/2014-9) से फंडिंग का धन्यवाद करते हैं। JLSM उत्पादकता अनुदान के लिए CNPq को धन्यवाद देता है (303664/2020-7)। MPP धन्यवाद Capes (मास्टर डिग्री छात्रवृत्ति, प्रक्रिया 88887.600591/2021-00) और CNPq।
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
References
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

