Isolamento, caracterização e extração de DNA total para identificação de fungos endofíticos em plantas micoheterotróficas
Summary
O presente artigo tem como objetivo fornecer protocolos detalhados e adequados para o isolamento de fungos endofíticos associados a plantas, preservação a longo prazo de isolados, caracterização morfológica e extração de DNA total para posterior identificação molecular e análises metagenômicas.
Abstract
As plantas micoheterotróficas apresentam uma das formas mais extremas de dependência micorrízica, tendo perdido totalmente sua capacidade autotrófica. Tão essenciais quanto qualquer outro recurso vital, os fungos com os quais essas plantas se associam intimamente são essenciais para elas. Assim, algumas das técnicas mais relevantes no estudo de espécies micoheterotróficas são as que possibilitam a investigação de fungos associados, especialmente aqueles que habitam raízes e órgãos subterrâneos. Nesse contexto, técnicas de identificação de fungos endofíticos dependentes e independentes de cultura são comumente aplicadas. O isolamento de endófitos fúngicos fornece um meio para identificá-los morfologicamente, analisar sua diversidade e manter inóculos para aplicações na germinação simbiótica de sementes de orquídeas. No entanto, sabe-se que existe uma grande variedade de fungos não cultiváveis habitando tecidos vegetais. Assim, técnicas de identificação molecular independentes de cultura oferecem uma cobertura mais ampla da diversidade e abundância de espécies. Este artigo tem como objetivo fornecer o suporte metodológico necessário para o início de dois procedimentos de investigação: um dependente de cultura e outro independente. Em relação ao protocolo cultura-dependente, são detalhados os processos de coleta e manutenção de amostras de plantas desde os locais de coleta até as instalações laboratoriais, além do isolamento de fungos filamentosos de órgãos subterrâneos e aéreos de plantas micoheterotróficas, manutenção de uma coleção de isolados, caracterização morfológica de hifas pela metodologia de cultura em lâminas e identificação molecular de fungos por extração de DNA total. Englobando metodologias independentes de cultura, os procedimentos detalhados incluem a coleta de amostras de plantas para análises metagenômicas e extração de DNA total de órgãos de plantas aclorofilas usando um kit comercial. Finalmente, protocolos de continuidade (por exemplo, reação em cadeia da polimerase [PCR], sequenciamento) também são sugeridos para análises, e técnicas são apresentadas aqui.
Introduction
Os fungos endofíticos são, por definição, aqueles que habitam o interior dos órgãos e tecidos vegetais em infecções imperceptíveis (isto é, sem causar danos ao seu hospedeiro)1,2. Esses fungos podem interagir de forma neutra ou benéfica com as plantas hospedeiras, podem conferir resistência a patógenos e condições ambientais desfavoráveis e podem contribuir para a síntese de compostos benéficos para a planta (por exemplo, fatores de crescimento e outros fitohormônios)1,3. Os endofíticos micorrízicos são fungos que estabelecem associações micorrízicas com a planta, participando da transferência denutrientes4. Em Orchidaceae, a interação com endófitos micorrízicos é fundamental para a germinação de sementes na grande maioria das espécies e estabelecimento de plântulas em todas as plantas da família5. Nesses contextos, as orquídeas micoheterotróficas representam um caso de total dependência em relação aos seus parceiros micorrízicos, uma vez que dependem da transferência de nutrientes minerais e compostos de carbono por esses fungos durante todo o seu ciclo devida6. Portanto, o isolamento e a identificação de fungos associados são uma base fundamental na investigação de estratégias de vida micoheterotrófica. Além disso, pouco se sabe sobre o papel dos fungos endófitos em plantas micoheterotróficas ou mesmo sobre a real diversidade desses fungos 7,8.
A investigação de fungos endofíticos pode ser realizada por meio de diferentes técnicas, tradicionalmente descritas como independentes ou dependentes de cultura, como, por exemplo: (a) observação direta, (b) isolamento fúngico e identificação morfológica e/ou molecular, e (c) extração total de DNA de tecidos vegetais e identificaçãomolecular9. Na observação direta (a), fungos endofíticos podem ser investigados ainda no interior de células e tecidos vegetais por microscopia de luz oueletrônica9, uma vez que diferentes protocolos de microscopia são detalhados por Pena-Passos et al.10. Pelos métodos de isolamento (b), os endófitos fúngicos podem ser caracterizados de acordo com suas colônias, hifas e morfologia da estrutura reprodutiva ou de resistência. Além disso, por meio de técnicas de isolamento, é possível realizar a identificação molecular de isolados por meio de extração de DNA, amplificação de sequências de identificação molecular (códigos de barras ou impressões digitais) e sequenciamento11. Esta última técnica (c) permite a identificação molecular de fungos endofíticos por extração de DNA no interior dos tecidos vegetais (metacódigo de barras), seguida de preparo e sequenciamento de bibliotecas12.
Além disso, isolados fúngicos podem ser aplicados em ensaios de germinação simbiótica, utilizando sementes de orquídeas autotróficas ou micoheterotróficas. Um exemplo dessa aplicação é a investigação conduzida por Sisti et al.13, descrevendo a germinação e os estágios iniciais de desenvolvimento do protocormo em Pogoniopsis schenckii, uma orquídea micoheterotrófica, em associação com alguns de seus isolados, compostos por fungos endofíticos não micorrízicos. O protocolo de germinação simbiótico aplicado é detalhado e apresentado em vídeo por Pena-Passos et al.10. O isolamento de fungos em associação com diferentes órgãos vegetais permite diversos enfoques de investigação sobre a natureza das interações planta-fungos (por exemplo, compreender aspectos ecológicos ou fisiológicos da associação, bem como investigações sobre a transferência de nutrientes dos fungos para a planta)9.
As metodologias apresentadas na seção 1 baseiam-se em uma coleção de amostras de órgãos subterrâneos, uma vez que esses órgãos apresentam maiores dificuldades de coleta, e são de grande interesse, uma vez que os endófitos micorrízicos os colonizam. No entanto, ambos os protocolos incluídos (etapas 1.1 e 1.2) podem ser aplicados a outros órgãos de plantas micoheterotróficas (por exemplo, rizomas, caules florais e frutos). A metodologia de coleta descrita na etapa 1.1 é designada para o isolamento de fungos endofíticos (seção 2) para caracterização morfológica (seções 4 e 5) e/ou extração total de DNA para identificação de isolados (seção 6). Por outro lado, a metodologia de coleta descrita na etapa 1.2 é atribuída exclusivamente à extração total de DNA de tecidos vegetais para técnicas de metabarcoding (seção 7). Na seção 3, são apresentados quatro métodos para armazenamento e preservação de fungos filamentosos, dois para armazenamento de curta duração (3-6 meses) e os outros dois adequados para armazenamento de longo prazo (>1 ano). A caracterização morfológica (seções 4 e 5) pode ser associada à identificação molecular para reforçá-la e fornecer informações importantes sobre macro e micromorfologia fúngica. A Figura 1 resume as metodologias coletivas descritas a seguir.
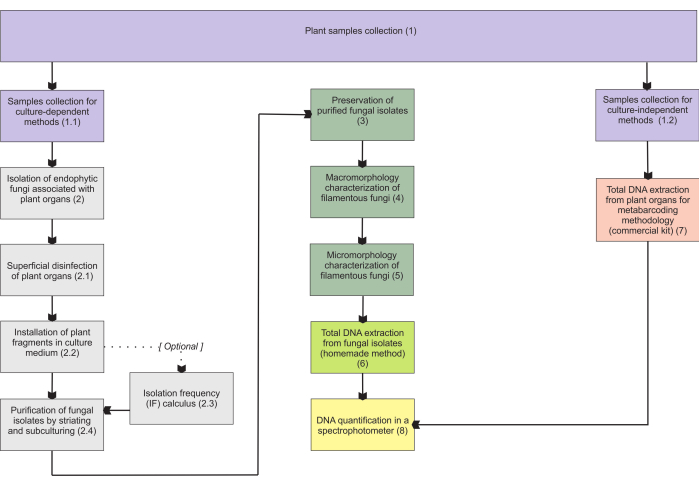
Figura 1: Sumarização esquemática dos métodos apresentados. Coleta de plantas e isolamento, preservação e identificação molecular de fungos por metodologias dependentes e independentes de cultura. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
A desinfestação superficial de amostras de plantas é uma das etapas mais críticas do protocolo apresentado. Nenhuma contaminação nas placas de PDA com gotas da última lavagem são altamente desejáveis. Bactérias são frequentemente observadas como contaminantes nas placas de isolamento, geralmente mais do que fungos esporulantes transportados pelo ar, considerando que bactérias endofíticas também são comuns nos tecidos vegetais 3,11. Assim, a adiçã…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos o financiamento da FAPESP (2015/26479-6) e do CNPq (447453/2014-9). JLSM agradece ao CNPq pelas bolsas de produtividade (303664/2020-7). O MPP agradece à Capes (bolsa de mestrado, processo 88887.600591/2021-00) e ao CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
References
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

