菌従属栄養植物における内生菌を同定するための単離、特性評価、および全DNA抽出
Summary
本稿では、植物関連内生菌の単離、分離株の長期保存、形態学的特性評価、およびその後の分子同定およびメタゲノム解析のための総DNA抽出のための詳細かつ適切なプロトコルを提供することを目的としています。
Abstract
菌従属栄養植物は、菌根依存の最も極端な形態の1つであり、独立栄養能力を完全に失っています。他の重要な資源と同様に、これらの植物が密接に関連している菌類は、彼らにとって不可欠です。したがって、菌従属栄養種の研究に最も関連性の高い技術のいくつかは、関連する真菌、特に根や地下器官に生息する真菌の調査を可能にするものです。これに関連して、文化依存性および培養非依存性の内生菌を同定する技術が一般的に適用される。真菌エンドファイトを単離することは、それらを形態学的に識別し、それらの多様性を分析し、ランの種子の共生発芽に応用するための接種を維持するための手段を提供します。しかし、植物組織に生息する非培養性真菌は多種多様であることが知られている。このように、培養に依存しない分子同定技術は、種の多様性と存在量をより広くカバーします。本稿は、文化依存型と独立型の2つの調査手順を開始するために必要な方法論的サポートを提供することを目的としています。培養依存性プロトコールに関しては、菌従属栄養植物の地下および空中器官からの糸状菌の単離、分離株の収集、スライド培養法による菌糸の形態学的特徴付け、および全DNA抽出による真菌の分子同定とともに、収集場所から実験室施設までの植物サンプルの収集と維持のプロセスが詳細に説明されています。培養に依存しない方法論を網羅する詳細な手順には、メタゲノム解析のための植物サンプルの収集や、市販のキットを使用したクロロフィル植物器官からの総DNA抽出が含まれます。最後に、連続性プロトコル(ポリメラーゼ連鎖反応[PCR]、シーケンシングなど)も分析に推奨されており、ここではその手法を紹介します。
Introduction
内生菌は、定義上、目立たない感染症で植物の器官や組織の内部に生息する菌類です(つまり、宿主に害を及ぼすことなく)1,2。これらの真菌は、宿主植物と中立的または有益に相互作用し、病原体や不利な環境条件に対する耐性を与え、植物にとって有益な化合物(成長因子やその他の植物ホルモンなど)の合成に寄与する可能性があります1,3。菌根内生菌は、植物と菌根の関連を確立し、栄養伝達に関与する真菌です4。ラン科では、菌根内生菌との相互作用は、大多数の種で種子の発芽、および科のすべての植物で苗の確立の基本です5。このような文脈では、菌従属栄養性のランは、菌根のパートナーに関する完全な依存の事例を表しており、ライフサイクル全体にわたってこれらの真菌によるミネラル栄養素と炭素化合物の移行に依存しています6。したがって、関連菌類の単離と同定は、菌従属栄養生物の生命戦略を研究する際の基本的な基盤となります。さらに、菌従属栄養植物における真菌エンドファイトの役割や、これらの真菌の実際の多様性についてはほとんど知られていません7,8。
内生菌類の調査は、伝統的に培養非依存性または依存性として説明されているさまざまな手法 を介して 実施できます:たとえば、(a)直接観察、(b)真菌の単離および形態学的および/または分子的同定、および(c)植物組織の全DNA抽出および分子同定9。直接観察(a)では、内生菌は、植物細胞や組織の内部にいる間に、光学顕微鏡または電子顕微鏡法9によって調査される可能性があり、さまざまな顕微鏡プロトコルがPena-Passosらによって詳述されています10。単離法(b)により、真菌エンドファイトは、そのコロニー、菌糸、および生殖または抵抗構造の形態に従って特徴付けることができます。また、単離技術 により 、DNA抽出、分子同定配列(バーコードまたは指紋)の増幅、およびシーケンシングによる単離株の分子同定を行うことが可能です11。後者の技術(c)は、植物組織の内部でDNA抽出(メタバーコーディング)を行い、その後にライブラリの調製とシーケンシングを行う12。
さらに、真菌分離株は、独立栄養または菌従属栄養性のランからの種子を使用して、共生発芽試験に適用できます。このような応用の一例は、Sistiらによって実施された調査であり13、菌従属栄養性のランである Pogoniopsis schenckiiの発芽と原生皮発生の初期段階を、非菌根性内生菌を含むその分離株のいくつかに関連して説明しています。適用された共生発芽プロトコルは、Pena-Passosらによるビデオで詳細に説明されています10。異なる植物器官と関連して真菌を単離することで、植物と真菌の相互作用の性質に関する多様な研究の焦点が可能になります(例えば、関連性の生態学的または生理学的側面のいずれかを理解するため、および真菌から植物への栄養素の移動に関する調査)9。
セクション1で提示された方法論は、地下臓器サンプルの収集に基づいており、これらの臓器は収集が最も困難であり、菌根内生菌がコロニーを形成するため、非常に興味深いものです。ただし、含まれている両方のプロトコル(ステップ1.1および1.2)は、他の菌従属栄養植物器官(例、根茎、花茎、および果実)に適用できます。ステップ1.1で説明した収集方法は、形態学的特性評価(セクション4および5)のための内生菌(セクション2)および/または分離株同定のための全DNA抽出(セクション6)に指定されています。一方、ステップ1.2で説明した収集方法は、メタバーコーディング技術(セクション7)のための植物組織の全DNA抽出にのみ割り当てられます。セクション3では、糸状菌の保存と保存のための4つの方法が提示されており、2つは短期保存(3〜6か月)に、他の2つは長期保存(>1年)に適しています。形態学的特徴付け(セクション4および5)は、それを強化し、真菌のマクロおよびミクロモルフォロジーに関する重要な情報を提供するために、分子同定と関連している可能性があります。 図 1 は、以降に説明する集合的な方法論をまとめたものです。
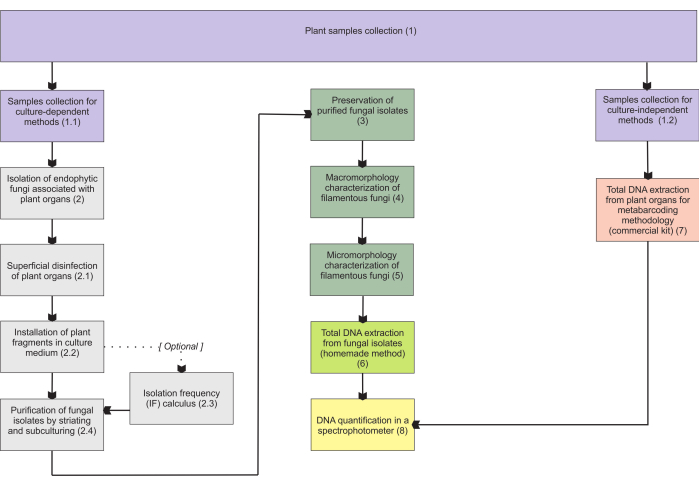
図1:提示された方法の概略図。植物の収集と真菌の分離、保存、および培養依存および非依存の方法論による分子同定。この図の拡大版をご覧になるには、ここをクリックしてください。
Protocol
Representative Results
Discussion
植物サンプルの表面的な駆除は、提示されたプロトコルの最も重要な段階の1つです。最後の洗浄からの滴でPDA皿に汚染しないことが強く望まれます。内生菌が植物組織内でも一般的であることを考慮すると、細菌は隔離皿の汚染物質として頻繁に観察され、通常は空気中の胞子形成菌よりも多くなります3,11。したがって、臓器片を設置する際の培…
Disclosures
The authors have nothing to disclose.
Acknowledgements
FAPESP(2015/26479-6)及びCNPq(447453/2014-9)からの資金提供に感謝します。JLSMは、生産性助成金(303664/2020-7)を提供してくれたCNPqに感謝します。MPPはCapes(修士号奨学金、プロセス88887.600591 / 2021-00)とCNPqに感謝します。
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
References
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

