Een Protocol voor de productie van Integrase-deficiënte lentivirale vectoren voor CRISPR/Cas9-gemedieerde Gene knock-out in cellen verdelen
Summary
We beschrijven de strategie van de productie van integrase-deficiënte lentivirale vectoren (IDLVs) als voertuigen voor het leveren van CRISPR/Cas9 naar de cellen. Met een vermogen om te bemiddelen snelle en robuuste gene bewerken in cellen, IDLVs presenteren een veiliger en even effectieve vector platform voor de levering van het gen in vergelijking met integrase-bevoegde vectoren.
Abstract
Lentivirale vectoren zijn een ideale keuze voor het bewerken van gene onderdelen leveren naar de cellen als gevolg van hun capaciteit voor stabiel transducing een brede waaier van cellen en bemiddelen van hoge niveaus van de genexpressie. Echter, hun vermogen om te integreren in het genoom van de cel host verhoogt het risico dat mutageniteit en dus verhoogt bezorgdheid over de veiligheid en beperkt hun gebruik in klinische instellingen. Verder, de aanhoudende uitdrukking van gen-editing-onderdelen geleverd door deze verhogingen van de integratie-bevoegde lentivirale vectoren (ICLVs) de kans op promiscue gentargeting. Als alternatief, heeft een nieuwe generatie van integrase-deficiënte lentivirale vectoren (IDLVs) ontwikkeld, waarin veel van deze zorgen. Hier het protocol van de productie van een nieuwe en verbeterde IDLV platform voor CRISPR-gemedieerde gene bewerken en lijst de stappen betrokken bij het zuiveren en concentratie van dergelijke vectoren wordt beschreven en hun transductie en bewerken van gen efficiëntie met behulp van HEK-293T cellen werd aangetoond. Dit protocol is eenvoudig schaalbaar en kan worden gebruikt voor het genereren van hoge titer IDLVs die kunnen transducing cellen in vitro en in vivo. Dit protocol kunnen bovendien gemakkelijk aan te passen voor de productie van ICLVs.
Introduction
Precieze gene bewerken vormt de hoeksteen van grote biomedische ontwikkelingen die betrekking hebben op de ontwikkeling van nieuwe strategieën om genetische ziekten te bestrijden. Op de voorhoede van gen-bewerken technologieën is de methode afhankelijk van het gebruik van de clustered regularly –Iknterspaced short palindromic repeats (CRISPR) / Cas9-systeem dat aanvankelijk werd geïdentificeerd Als een onderdeel van bacteriële immuniteit tegen de invasie van virale genetische materiaal (herzien in verwijzingen1,2). Een groot voordeel van het systeem van CRISPR/Cas9 over andere gen-bewerkingsgereedschap, zoals zink-vinger nucleasen (ZFNs) en transcriptie activator-achtige effector nucleasen (TALENs) (herzien in verwijzing3), is de relatieve eenvoud van plasmide ontwerp en bouw van de CRISPR componenten — een functie die de uitbreiding van het gen-bewerkingen van enkele gespecialiseerde laboratoria tot een veel ruimere onderzoeksgemeenschap heeft aangedreven. Bovendien hebben de eenvoud van CRISPR/Cas9 programmering en haar capaciteit voor multiplexed doel erkenning voor het verder aangewakkerd zijn populariteit als een rendabele en easy-to-use technologie. Onder de verschillende methoden beschikbaar voor onderzoekers te leveren van dergelijke componenten bewerken van gen naar cellen, blijven virale vectoren verreweg het populairste en meest efficiënte systeem.
Lentivirale vectoren (LVs) opgedoken als het voertuig van keuze te leveren van de onderdelen van CRISPR/Cas9 systeem in vivo voor uiteenlopende toepassingen4,5,6,7. Aantal belangrijke functies maken LVs een populaire keuze voor dit proces met inbegrip van hun vermogen om te verdelen en niet-delende cellen, lage immunogeniciteit zowel minimale cellulaire toxiciteit (herzien in verwijzing8) besmetten. Dientengevolge, is LV-gemedieerde gentherapie werkzaam in de behandeling van infectieziekten, zoals HIV-1, HBV en HSV-1, alsook in de correctie van fouten ten grondslag liggen aan erfelijke ziekten bij de mens, zoals mucoviscidose en neo-vasculaire macula degeneratie 4 , 5 , 7 , 9 , 10 , 11. Bovendien, LVs effectief zijn gewijzigd om een multiplex gene bewerkingsfuncties op verschillende genomic loci met behulp van een enkele vector systeem12.
Echter de inherente eigenschap van LVs te integreren in het genoom van de gastheer kan mutagene en handicaps vaak hun nut als transgenic leverende voertuigen, met name in klinische instellingen. Bovendien, aangezien stabiel geïntegreerde LVs hun transgenen op een duurzaam hoog niveau uiten, dit systeem is niet geschikt voor de levering van gen-bewerken onderdelen zoals CRISPR/Cas9; overexpressie van Cas9-gids RNA (gRNA), en soortgelijke eiwitten zoals ZFNs, worden geassocieerd met verhoogde niveaus van off-target effecten, waaronder ongewenste mutaties13,14,15,16 , 17 en het potentieel van de cytotoxiciteit18kan verbeteren. Daarom, om precieze gen-bewerken met minimale uit-target effecten, is het noodzakelijk om ontwerpsystemen waarmee voor de voorbijgaande expressie van gen editing-onderdelen.
In de afgelopen jaren een scala aan levering platforms zijn ontwikkeld om te Transient express CRISPR/Cas9 in cellen16,19,20,21 (herzien in verwijzing22). Het gaat hierbij om methoden die afhankelijk zijn van direct invoering van gezuiverde Cas9 samen met de juiste gids RNAs in cellen, die werd getoond om meer effectief bij gerichte gen-bewerken in vergelijking met de plasmide gemedieerde transfectie16. Studies hebben aangetoond dat ribonucleoprotein (RNP) complexen bestaande uit begeleiden RNA/Cas9 deeltjes zijn snel overgedragen na bemiddeling van DNA decollete op hun doelen, suggereert dat op korte termijn uitdrukking van deze componenten is voldoende om te bereiken robuuste gene16bewerken. Denkbaar, niet-integratie van virale vector platforms zoals adeno-geassocieerde virale vectoren (AAVs) bieden een levensvatbaar alternatief voor het bewerken van gen machines leveren aan cellen. Helaas, AAV capsids aanzienlijk lager verpakking mogelijkheid dan LVs bezitten (< 5kb), die ernstig belemmert hun vermogen voor het inpakken van de multi-component CRISPR toolkit binnen een enkele vector (herzien in verwijzing8). Het is vermeldenswaard dat toevoeging van verbindingen die remmen Histon deacetylases (b.v., natrium butyraat23) of belemmeren van de celcyclus (b.v., cafeïne24) is aangetoond dat het verhogen van lentivirale titers. Ondanks de recente vooruitgang, zijn de Transiënte expressiesystemen ontwikkeld tot nu toe nog steeds belemmerd door verschillende tekortkomingen, zoals lagere productie-efficiëntie, die tot verminderde virale titers, en lage transductie efficiëntie van de virussen die zijn gegenereerd leiden door dergelijke benaderingen25.
Integrase-deficiënte lentivirale vectoren (IDLVs) vormen een grote vooruitgang in de ontwikkeling van gen-leverende voertuigen, zoals zij het vermogen van de verpakking van LVs met het extra voordeel van AAV-achtige episomal onderhoud in cellen combineren. Deze voorzieningen helpen IDLVs grotendeels het omzeilen van de belangrijke kwesties in verband met het integreren van vectoren, vis-à-vis continu overexpressie van potentieel genotoxisch elementen en integratie-gemedieerde mutageniteit. Het werd eerder aangetoond dat IDLVs met succes kan worden aangepast voor het verbeteren van de episomal gen expressie26,27. Met betrekking tot de CRISPR/Cas9 IDLV-gemedieerde levering beperkt lage productie titers en lagere expressie van isomer overgedragen genomen ten opzichte van integrase-bedreven lentivirale systemen hun nut als bona fide tools voor het leveren van genoom-bewerken transgene constructies. We onlangs aangetoond dat zowel transgenic expressie en virale titers IDLV productie gekoppeld aanzienlijk worden verhoogd door de opneming van bindende sites voor de transcriptiefactor Sp1 binnen de virale expressie cassette28. De gewijzigde IDLVs ondersteund krachtig CRISPR-gemedieerde gene zowel in vitro (HEK-293T cellen) en in-vivo (in post mitotische hersenen neuronen), terwijl het minimale uit-target mutaties ten opzichte van de bijbehorende ICLV-gemedieerde inducerende bewerken systemen28. Globaal, ontwikkelden we een roman, compact, alles-in-één CRISPR toolkit uitgevoerd op een platform van IDLV en geschetst van de verschillende voordelen van het gebruik van dergelijke een leveringsvoertuig voor het bewerken van de verbeterde gen.
Hier, is het protocol van de productie van de IDLV-CRISPR/Cas9-systeem beschreven, met inbegrip van de verschillende stappen in de vergadering, zuivering, concentratie en titratie van IDLVs, evenals strategieën voor het valideren van het bewerken van gene werkzaamheid van deze vectoren. Dit protocol is eenvoudig schaalbaar naar de behoeften van verschillende onderzoekers en is ontworpen om met succes tot LV vectoren met titers in het bereik van 1 x 1010 eenheden (TU) transducing / mL. De vectoren gegenereerd door dit protocol kunnen worden gebruikt om verscheidene verschillende soorten cellen, met inbegrip van moeilijk-aan-transduce embryonale stamcellen, hematopoietische cellen (T-cellen en macrofagen), en gekweekt en in vivo– efficiënt te besmetten ingespoten neuronen. Bovendien is het protocol is even geschikt voor de productie van integrase-bevoegde lentivirale vectoren in vergelijkbare hoeveelheden.
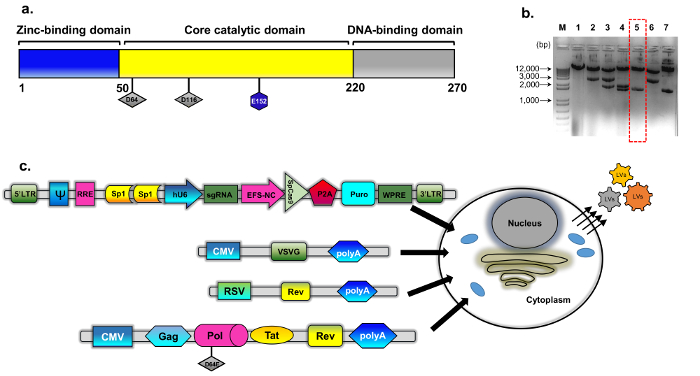
Figuur 1: IDLV verpakking. (a) schematische voorstelling van de wild type integrase eiwit (b) de gemodificeerde plasmide is afgeleid van psPAX2 (Zie methoden, plasmide constructie voor details). Vertegenwoordiger agarose gel afbeelding van klonen gescreend voor gemuteerde integrase klonen. Bereid met behulp van een standaard plasmide DNA isolatie Mini-Kit DNA-monsters werden geanalyseerd door de spijsvertering met EcoRV en SphI. De kloon van goed verteerd (nummer 5, onderbroken rood kader) werd verder gecontroleerd door directe (Sanger) volgorde van de vervanging van de D64E in INT. De integrase-deficiënte verpakking cassette heette pBK43. (c) schematische voorstelling van het protocol van de voorbijgaande transfectie werkzaam voor het genereren van IDLV-CRISPR/Cas9 vectoren, toont 293T cellen transfected met VSV-G, verpakking en transgenic cassettes (plasmide van de alles-in-één van de Sp1-CRISPR/Cas9). Virale deeltjes die bud uit van de celmembraan bevatten de full-length RNA van de vector (uitgedrukt uit de transgenic cassette). De tweede generatie van het systeem van de IDLV-verpakking werd gebruikt, waaronder de regelgevende eiwitten Tat en Rev. Rev expressie is verder aangevuld van een aparte cassette (RSV-REV-plasmide). Abbrev: LTR-lange-terminal herhalen, VSV-G, vesiculaire stomatitis virus G-eiwit, pCMV-cytomegalovirus promotor; Rous Sarcoom virus (RSV) promotor; RRE-(Rev respons-Element). Andere regulerende elementen op de expressie cassette opnemen Sp1-bandplaatsen, Rev Response element (RRE), Woodchuck Hepatitis Virus Posttranscriptional regelgevende Element (WPRE), een kern-rek factor 1α promotor (EFS-NC), de vector-verpakking element ψ (psi), menselijke Cytomegalovirus (hCMV) promotor en menselijke U6 promotor. Klik hier voor een grotere versie van dit cijfer.
Protocol
Representative Results
Discussion
IDLVs zijn begonnen te ontstaan als het voertuig van keuze voor in vivo gen-bewerking, met name in het kader van genetische ziekten, grotendeels als gevolg van het geringe risico van mutagenese gekoppeld aan deze vectoren in vergelijking met de integratie van levering platformen22 , 28. in het huidige manuscript, wij willen het protocol die samenhangen met de opwekking van het verbeterde alles-in-één IDLV-CRISPR/Cas9-systeem, dat onlangs in ons laborat…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We zouden graag bedanken het departement van neurobiologie, Duke University School of Medicine en Dean’s Bureau voor fundamentele wetenschap, Duke University. Wij danken ook leden van de Duke virale-Vector kern voor opmerkingen op het manuscript. Plasmide lolploeg CRISPRv2 was een geschenk van Feng Zhang (brede Instituut). De LV-verpakkingen systeem met inbegrip van de psPAX2 van plasmiden, was VSV-G, pMD2.G en pRSV-Rev een soort geschenk van Didier Trono (EPFL, Zwitserland). Financiële steun voor dit werk werd verstrekt door de University Of South Carolina School Of Medicine, verlenen RDF18080-E202 (B.K).
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

