Ein Protokoll für die Produktion der Integrase-defizienten Lentivirale Vektoren für CRISPR/Cas9-vermittelte Gen Knockout in teilenden Zellen
Summary
Wir beschreiben die Produktionsstrategie der Integrase-defizienten Lentivirale Vektoren (IDLVs) als Vehikel für Zellen CRISPR/Cas9 bereitzustellen. Mit der Fähigkeit, schnell und robust Gene in Zellen bearbeiten zu vermitteln IDLVs präsentieren ein sicherer und ebenso wirksame Vektor Plattform für gen-Lieferung im Vergleich zu Integrase-kompetente Vektoren.
Abstract
Lentivirale Vektoren sind eine ideale Wahl für die Bereitstellung von gen-Bearbeiten von Komponenten auf Zellen aufgrund ihrer Fähigkeit zur stabil transducing einen breiten Bereich von Zellen und hohe Stufen der Genexpression zu vermitteln. Jedoch ihre Fähigkeit zur Integration in das Wirtsgenom Zelle erhöht das Risiko der insertional Mutagenität und damit Sicherheit Bedenken und schränkt ihre Verwendung in klinischen Umgebungen. Weiter, geliefert der anhaltenden Ausdruck der gen-Bearbeiten von Komponenten durch diese Integration zuständigen Lentivirale Vektoren (ICLVs) erhöht die Wahrscheinlichkeit von promiscuous Gene targeting. Als Alternative hat eine neue Generation von Integrase-defizienten Lentivirale Vektoren (IDLVs) entwickelt, die viele dieser Bedenken befasst. Hier Produktionsprotokoll einer neuen und verbesserten IDLV-Plattform für CRISPR-vermittelten gen bearbeiten und Liste der Schritte bei der Reinigung und Konzentration von solchen Vektoren beschrieben und deren Transduktion und gen-Bearbeitung Effizienz mit HEK-293T Zellen zeigte. Dieses Protokoll ist leicht skalierbar und kann verwendet werden, um hohe Titer IDLVs generieren, die in der Lage, transducing Zellen in Vitro und in Vivo. Darüber hinaus kann dieses Protokoll für die Herstellung von ICLVs leicht angepasst werden.
Introduction
Gen präzise Bearbeitung bildet den Grundstein biomedizinische Fortschritte, die die Entwicklung neuer Strategien zur Bekämpfung genetischer Krankheiten betreffen. An die Spitze der gen-Bearbeitungs-Technologien ist die Methode unter Berufung auf die Nutzung der clustered rregelmässig –IchNterspaced sHort pAlindromic REpeats (CRISPR) / Cas9-System, das zunächst identifiziert wurde als Bestandteil des bakteriellen Immunität gegen die Invasion der viralen Erbsubstanz (rezensiert in Referenzen1,2). Ein wesentlicher Vorteil des CRISPR/Cas9-Systems gegenüber anderen gen-editing-Tools, wie z. B. Zinkfinger-Nukleasen (ZFN) und Transkription Aktivator-ähnliche Effektor Nukleasen (TALENs) (rezensiert in Referenz3), ist die relative Einfachheit der Plasmid-Design und Bau von CRISPR-Komponenten – ein Feature, das den Ausbau der gen-Bearbeitung von wenigen Speziallabors für eine viel breitere Forschungsgemeinschaft angetrieben hat. Darüber hinaus haben die Einfachheit der CRISPR/Cas9 Programmierung und seine Kapazität für Multiplex Zielerfassung seine Popularität als eine kostengünstige und einfach zu bedienende Technologie weiter angeheizt. Unter den verschiedenen Methoden zur Verfügung, um Forscher solche gen-Bearbeiten von Komponenten an Zellen zu liefern bleiben virale Vektoren bei weitem die beliebteste und effizientes System.
Lentivirale Vektoren (LVs) entstanden als das Fahrzeug der Wahl für die Komponenten des CRISPR/Cas9 System in Vivo für unterschiedlichste Anwendungen4,5,6,7liefert. Mehrere wichtige Funktionen machen LVs eine beliebte Wahl für diesen Prozess, einschließlich ihrer Fähigkeit, sowohl trennende und nicht teilenden Zellen, niedrige Immunogenität und minimale zelluläre Toxizität (rezensiert in Referenz8) zu infizieren. Infolgedessen wurde LV-vermittelte Gentherapie in der Behandlung von Infektionskrankheiten wie HIV-1, HBV und HSV-1, sowie bei der Korrektur von Mängeln, die zugrunde liegenden menschlichen Erbkrankheiten, wie Mukoviszidose und Neo-Kreislauf Makuladegeneration eingesetzt 4 , 5 , 7 , 9 , 10 , 11. Außerdem, LVs wurden effektiv geändert um Multiplex-gen an unterschiedliche genomic Loci mit einem einzigen Vektor System12Bearbeitung ausführen.
Jedoch kann die inhärente Eigenschaft des LVs in das Wirtsgenom integrieren mutagen und oft behindert ihre Nützlichkeit als Transgen Lieferfahrzeuge, vor allem im klinischen Umfeld. Da zudem stabil integriert LVs ihre transgene auf einem nachhaltig hohen Niveau zum Ausdruck bringen, ist dieses System ungeeignet für die Lieferung von gen-Bearbeiten von Komponenten wie CRISPR/Cas9; Überexpression des Cas9-Guide RNA (gRNA) und ähnliche Proteine wie ZFN, sind verbunden mit erhöhten Konzentrationen von Ziel-Effekte, darunter unerwünschte Mutationen13,14,15,16 , 17 und können potenziell Zytotoxizität18verbessern. Daher unbedingt um präzise zu erreichen gen-Bearbeitung mit minimal Ziel Effekte, Design-Systemen, die für die transiente Expression des Gens, die Bearbeitung von Komponenten ermöglichen.
In den letzten Jahren wurden eine Vielzahl von Plattformen entwickelt, um vorübergehend CRISPR/Cas9 in Zellen16,19,20,21 (rezensiert in Referenz22) zum Ausdruck bringen. Dazu gehören Methoden, die direkt Einführung von gereinigten Cas9 zusammen mit der passenden Guide-RNAs in Zellen, die gezeigt wurde, effektiver auf gezielte gen-Bearbeitung im Vergleich zu Plasmid-vermittelte Transfektion16abhängig. Studien haben gezeigt, dass Ribonucleoprotein (RNP) komplexe, bestehend aus Leitfaden RNA/Cas9 Partikel werden schnell umgedreht nach Vermittlung Spaltung der DNA auf ihre Ziele, was darauf hindeutet, dass kurzfristige Ausdruck dieser Komponenten zu erzielen ist robuste gen16bearbeiten. Denkbar, nicht-Integration von viralen Vektoren Plattformen wie Adeno-assoziierten viralen Vektoren (Flugabwehrpanzer) bieten eine echte Alternative um gen-Bearbeitung Maschinen an Zellen zu liefern. Leider AAV Capsids besitzen deutlich geringere Verpackung Fähigkeit als LVs (< 5kb), die schwer behindert ihre Fähigkeit, das Mehrkomponenten CRISPR-Toolkit innerhalb einer einzigen Vektor (rezensiert in Referenz8) zu verpacken. Es ist erwähnenswert, dass die Zugabe von Verbindungen, die Histon Deacetylases (z.B. Natrium Butyrat23) hemmen oder verhindern den Zellzyklus (z.B. Koffein24) gezeigt worden, um Lentivirale Titer zu erhöhen. Trotz der jüngsten Fortschritte sind die transiente Expression-Systeme so weit entwickelt noch mehrere Mängel auf, z. B. geringere Produktionseffizienz, behindert durch führen zu reduzierten virale Titer und niedrigen Transduktion Effizienz erzeugt durch Viren solche Ansätze25.
Integrase-defizienten Lentivirale Vektoren (IDLVs) stellen einen großen Fortschritt in der Entwicklung von gen-Lieferfahrzeuge, wie sie die Verpackung-Fähigkeit des LVs mit dem zusätzlichen Vorteil der AAV-artigen episomal Wartung in Zellen verbinden. Diese Funktionen helfen IDLVs, die die wichtigsten Probleme im Zusammenhang mit Integration von Vektoren, Vis À Vis kontinuierliche Überexpression von potenziell genotoxische Elementen und Integration-vermittelten Mutagenität weitgehend zu umgehen. Es wurde bereits gezeigt, dass IDLVs erfolgreich zur Verbesserung der episomal gen Ausdruck26,27geändert werden kann. In Bezug auf IDLV-vermittelten CRISPR/Cas9 Lieferung beschränkt niedrige Titer und niedriger Ausdruck von Episome getragen Genomen relativ Integrase-kompetente Lentivirale Systeme ihre Nützlichkeit als Bona Fide -Tools für die Bereitstellung von Genom-Bearbeitung transgene Konstrukte. Wir zeigten kürzlich, dass Transgene Ausdruck und virale Titer IDLV Produktion durch die Einbeziehung von Bindungsstellen für den Transkriptionsfaktor Sp1 in der viralen Ausdruck Kassette28erheblich verbessert werden. Die modifizierte IDLVs unterstützt robust CRISPR-vermittelten gen Bearbeitung sowohl in Vitro (in HEK-293T Zellen) und in Vivo (im Post-mitotische Gehirnneuronen), während minimalste Ziel Mutationen im Vergleich zu den entsprechenden ICLV-vermittelte Induktion Systeme-28. Insgesamt entwickelten wir eine neuartige, kompakte, all-in-One CRISPR-Toolkit auf einer IDLV Plattform durchgeführt und erläutert die verschiedenen Vorteile der Verwendung solch ein Transportvehikel für verbesserte gen zu bearbeiten.
Hier ist das Produktionsprotokoll des IDLV-CRISPR/Cas9-Systems beschrieben, darunter die verschiedenen Schritte in der Montage, Reinigung, Konzentration und Titration von IDLVs sowie Strategien, die gen-Bearbeitung Wirksamkeit dieser Vektoren zu validieren. Dieses Protokoll ist leicht skalierbar, um die Bedürfnisse der verschiedenen Forschern und wurde entwickelt, um erfolgreich LV Vektoren mit Titer im Bereich von 1 x 1010 transducing Einheiten (TU) erzeugen / mL. Die Vektoren generiert durch dieses Protokoll können genutzt werden, um effizient mehrere verschiedene Zelltypen, einschließlich schwer transduzieren embryonale Stammzellen, blutbildenden Zellen (T-Zellen und Makrophagen) und kultiviert und in Vivozu infizieren- eingespritzte Neuronen. Darüber hinaus ist das Protokoll für die Produktion der Integrase-kompetente Lentivirale Vektoren in ähnlichen Mengen gleich gut geeignet.
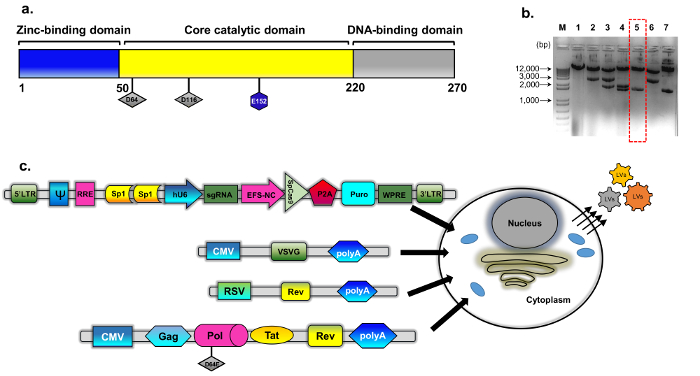
Abbildung 1: IDLV Verpackung. (a) schematische Darstellung der Wildtyp Integrase Protein (b) das modifizierte Plasmid wurde von psPAX2 abgeleitet (siehe Methoden, Plasmid-Konstruktion für Details). Vertreter Agarose Gelbild geschirmt für mutierte Integrase Klone Klone. DNA-Proben mit den standard Plasmid DNA-Isolierung Mini-Kit zubereitet wurden durch Vergärung mit EcoRV und SphI analysiert. Der richtig verdaut Klon (Nummer 5, gestrichelten roten Feld) wurde weiter durch Sequenzierung der direkten (Sanger) für die D64E Substitution in INTüberprüft. Die Integrase-defizienten Verpackung Kassette wurde pBK43 genannt. (c) schematische Darstellung der transiente Transfektion Protokoll eingesetzt, um generieren IDLV-CRISPR/Cas9 Vektoren zeigen, dass 293T Zellen mit VSV-G, Verpackungen und Transgen Kassetten (Sp1-CRISPR/Cas9 all-in-One Plasmid) transfiziert. Viruspartikel, die heraus von der Zellmembran Knospe enthalten die Full-length RNA des Vektors (ausgedrückt aus der Transgen-Kassette). Die zweite Generation der IDLV-Verpackungssystem diente, worunter die regulatorische Proteine Tat und Rev Rev Ausdruck wird von einer separaten Kassette (RSV-REV-Plasmid) ergänzt. Abbrev: LTR Long Terminal repeat, VSV-G, Stomatitis vesicularis Virus G-Protein, pCMV-Zytomegalievirus Veranstalter; Rous Sarkom-Virus (RSV) Veranstalter; RRE-(Rev-Response-Element). Weitere regulatorische Elemente auf der Expressionskassette gehören Sp1-Bindungsstellen, Rev Response-Element (RRE), Murmeltier Hepatitis Virus posttranskritionelle regulatorische Element (WPRE), Kern-Dehnung Faktor 1α Förderer (EFS-NC), der Vektor-Verpackung Element-ψ (Psi), menschliches Cytomegalovirus (hCMV) Veranstalter und menschlichen U6 Förderer. Bitte klicken Sie hier für eine größere Version dieser Figur.
Protocol
Representative Results
Discussion
IDLVs haben damit begonnen, als das Fahrzeug der Wahl für die in-Vivo -gen-Bearbeitung, vor allem im Zusammenhang mit genetischen Erkrankungen, weitgehend aufgrund des geringen Risikos Mutagenese diese Vektoren im Vergleich zu integrieren Lieferung Plattformen22 zugeordnet entstehen , 28. im aktuellen Manuskript, wollten wir ausführlich das Protokoll verbunden mit der Produktion von der verbesserten all-in-One IDLV-CRISPR/Cas9 System, das vor kurzem in…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir möchten die Abteilung für Neurobiologie, Duke University School of Medicine und Dekanat für Grundlagenforschung, Duke University bedanken. Wir danken auch Mitglieder des Duke viraler Vektor Kerns für Kommentare auf das Manuskript. Plasmid pLenti CRISPRv2 war Geschenk von Feng Zhang (Broad Institute). Die LV-Verpackungssystem einschließlich der Plasmide psPAX2, war VSV-G, pMD2.G und pRSV-Rev eine Art Geschenk von Didier Trono (EPFL, Schweiz). Gewähren Sie finanzieller Unterstützung für diese Arbeit von der University Of South Carolina School Of Medicine, zur Verfügung gestellt wurde, RDF18080-E202 (B.K).
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

