細胞分裂に CRISPR/Cas9 を介した遺伝子ノックアウトのインテグラーゼ欠損レンチウイルスベクターの生産のためのプロトコル
Summary
セルに CRISPR/Cas9 を提供するための車としてインテグラーゼ欠損レンチウイルスベクター (IDLVs) の生産戦略について述べる。迅速かつ堅牢な遺伝子がセル内編集を仲介する能力、IDLVs 現在安全と同様に効果的なベクトル遺伝子送達用に比べてベクトル インテグラーゼ主務。
Abstract
レンチウイルスベクターは、安定して高いレベルの遺伝子発現を仲介する細胞の広い範囲を伝達の能力による細胞に遺伝子編集コンポーネントを提供するための理想的な選択肢です。しかし、宿主細胞のゲノムに統合する能力を挿入変異原性のリスクを高めるため安全上の懸念を発生させます、臨床現場での使用を制限します。さらに、これら統合主務レンチウイルスベクター (ICLVs) 増加によって無差別遺伝子ターゲティングの確率を配信遺伝子編集コンポーネントの永続的な表現。代わりは、これらの問題の多くが解決、インテグラーゼ欠損レンチウイルスベクター (IDLVs) の新世代が開発されています。ここ CRISPR 媒介性遺伝子編集およびリストのための新しく、改良された IDLV プラットフォームの生産のプロトコル、精製の手順し、このようなベクトルの濃度が記載されて伝達と HEK 293 t を使用して遺伝子編集効率細胞を示した。このプロトコルは、拡張が容易細胞in vitroとin vivoのことができる高価 IDLVs を生成に使用することができます。さらに、このプロトコルは、ICLVs の生産のために容易に適応することができます。
Introduction
正確な遺伝子編集遺伝的疾患に取り組むために新しい戦略の開発を含む生物医学的進歩の基礎を形成します。遺伝子編集技術の最前線は、 c光沢regularly –私nterspaced s「hort」 palindromic repeats (CRISPR) の使用に依存するメソッド/最初に識別された Cas9 システム(参照1,2レビュー) ウイルスの遺伝物質の侵入に対する細菌免疫のコンポーネント。亜鉛指核酸 (ZFNs) と転写活性化因子のようなエフェクター核酸分解酵素 (TALENs) を (参照3の見直し) などの他の遺伝子編集ツール以上 CRISPR/Cas9 システムの主な利点はプラスミド設計の相対的なシンプルさとCRISPR コンポーネントの構築-の電源が大いにより広い研究コミュニティにいくつかの専門の研究所から遺伝子-編集の拡張機能。また、CRISPR/Cas9 プログラミングの簡潔さと多重ターゲット認識能力さらにコスト効果の高い、簡単に使用できる技術としてその人気を煽っています。ウイルスのベクトル、細胞にこのような遺伝子編集コンポーネントを提供する研究者に利用できるさまざまな方法の中で最も人気のある、効率的なシステムのまま抜いて。
レンチウイルスベクター (LVs) は、CRISPR/Cas9 システム生体内で多様なアプリケーション4,5,6、7のためのコンポーネントを提供する最適な手段として浮上しています。いくつかの主要な機能は、LVs 分割と非分裂細胞、低免疫原性と最小の細胞毒性 (文献8の見直し) の両方に感染する能力を含むこのプロセスの人気のある選択肢を作る。結果として、LV 媒介性遺伝子治療は人間の遺伝性疾患、嚢胞性線維症やネオ血管の加齢黄斑変性などの基礎となる欠陥の補正だけでなく、HIV 1、HBV、HSV-1 などの感染症の治療に採用されています。4,5,7,9,10,11します。 また、LVs を効果的に多重遺伝子の単一のベクトル システム12を使用して異なるゲノム遺伝子編集を実行に変更されています。
しかし、Lv がホストのゲノムに統合するための固有の特性は変異することができます、しばしばハンディキャップとして transgene の配送車、特に臨床の現場での実用。さらに、Lv が安定的に統合された持続可能な高いレベルで自分の遺伝子を表現、以来このシステムは適して CRISPR/Cas9; など遺伝子編集コンポーネントの配信のためCas9 ガイド RNA (gRNA)、および ZFNs など同じような蛋白質の過剰発現は望ましくない突然変異13,14,15,16を含むオフターゲット効果の高いレベルに関連付けられています。,17 18細胞毒性を高めることができる可能性があります。したがって、正確な達成するために最小限のオフターゲット効果と遺伝子編集だ編集コンポーネント遺伝子の一過性発現を可能にするシステムを設計することが不可欠。
近年、一過性細胞16,19,20,21 (参照22レビュー) に CRISPR/Cas9 を表現するさまざまな配信プラットフォームが開発されています。直接ご紹介と共に適切なガイド Rna 精製 Cas9 細胞にトランスフェクションのプラスミド性16と比較する対象となる遺伝子編集でより効果的に示されていたに依存するメソッドが含まれます。そのリボ核タンパク質 (RNP) の調査は示した錯体から成るガイド RNA/Cas9 粒子はそれらの目標は、これらのコンポーネントの短期的な表現が達成するために十分であることを示唆で DNA 切断を仲介後急速にめくって堅牢な遺伝子16を編集します。多分、非統合アデノ随伴ウイルスベクター (通気) などウイルスのベクトルのプラットフォームは細胞に遺伝子編集機械を提供する実行可能な代案を提供できます。残念ながら、AAV カプシドは Lv よりも大幅に低い包装機能を所有している (< 5 kb)、これは深刻な (文献8の見直し) 単一ベクター内多成分 CRISPR ツールキットをパッケージ化する能力を妨げます。それは、レンチ ウイルスの力価を増加する (例えばナトリウム酪酸23) にヒストン脱アセチル化酵素を阻害する、または (例えば、カフェイン24) 細胞周期阻害化合物添加が示されていることは注目に値するです。最近の進歩にもかかわらず、これまで開発した一過性発現システムはまだ生産効率が悪くなどに、減らされたウイルス抗体とウイルスを介して生成された低伝達効率につながる欠点によって妨げ25をのようなアプローチします。
インテグラーゼ欠損レンチウイルスベクター (IDLVs) は、LVs の包装機能を組み合わせて細胞における AAV のような episomal 整備の利点として遺伝子デリバリー車両の開発に大きな進歩を表しています。これらの機能は、主ベクトル、潜在的遺伝毒性要素の統合による変異原性に対して-à-に対して継続的な過剰発現の統合に関連する主要な問題を回避 IDLVs を助けます。以前 episomal 遺伝子式26,27を強化する IDLVs、正常に変更できることが示された.IDLV を介した CRISPR/Cas9 配信に関して低生産価とインテグラーゼ熟練したレンチウイルスベクター システムと比較して episome 由来のゲノムの低い表現制限ゲノム編集を提供するための善意のツールとしての実用遺伝子組換えを構築します。我々 は最近、遺伝子発現および IDLV の生産に関連付けられているウイルスの抗体は、ウイルス発現カセット28内転写因子 Sp1 のためのサイトのバインドを含めることによって大幅に強化を示した。変更された IDLVs は確実 CRISPR 媒介性遺伝子 ICLV を介した対応と比較して最小限のオフのターゲット変異を誘導しながら両方体外(HEK 293 t 細胞)体内(分裂後の脳ニューロン) での編集をサポートシステム28。全体的に、小説を開発した、コンパクト、CRISPR ツールキット IDLV プラットフォームで実施し、強化された遺伝子編集のような配信手段を使用しての様々 な利点を説明しました。
ここでは、IDLV-CRISPR/Cas9 システムの生産プロトコルは述べたように、アセンブリ、浄化、集中、および IDLVs、だけでなく、戦略は、これらのベクトルの遺伝子編集の有効性を検証するための滴定に関連するさまざまな手順を含みます。このプロトコルは別の捜査官のニーズを満たすために拡張が容易と 1 x 10 の10単位 (TU) の伝達の範囲の値と LV ベクトルを正常に生成するように設計/mL。このプロトコルを介して生成されたベクトルを活用すると、効率的にいくつかの異なる種類の細胞、培養および変換困難な胚性幹細胞、造血細胞 (T 細胞やマクロファージ) を含むとin vivo– に感染すること注入されたニューロン。さらに、プロトコルは似たような量のインテグラーゼ有能なレンチウイルスベクターの生産のため均等に適しています。
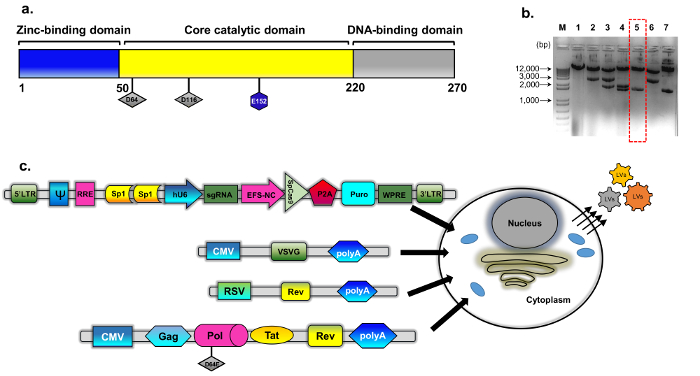
図 1: IDLV 包装します。変更されたプラスミドは、psPAX2 から派生した野生型インテグラーゼ蛋白質(b)の(a)の回路図 (詳細についてはプラスミド構築メソッドを参照してください)。クローン変異インテグラーゼ クローンのスクリーニングの代表 agarose ゲル画像。標準プラスミド DNA 分離ミニ キットを使用して作製した DNA サンプルは、EcoRV と SphI 消化によって分析されました。正しく消化クローン (番号 5、赤い点線) は直通 (サンガー) intD64E 置換シーケンスによってさらに検証されました。インテグラーゼ欠損包装カセット pBK43 に選ばれました。(c)トランスフェクション プロトコルの概略図は VSV G、パッケージング、および遺伝子カセット (Sp1-CRISPR/Cas9 オール イン ワン プラスミッド) をトランスフェクトした 293 t 細胞を示す IDLV-CRISPR/Cas9 ベクトルを生成する採用。細胞膜から芽をウイルス粒子には (遺伝子カセットから表される) ベクトルのフルレングスの RNA が含まれます。Tat の規制のタンパク質が含まれています、IDLV 包装システムの第二世代が使用され、牧師回転式はさらに別のカセット (RSV REV プラスミド) から補われます。略語: LTR 長いターミナル繰り返し、VSV-g の水疱性口内炎ウイルス pCMV サイトメガロ ウイルス プロモーター; G タンパク質ラウス肉腫ウイルス (RSV) プロモーター。RRE – (回転応答要素)。式カセットの他の規制要素があります Sp1 結合部位、Rev 応答要素 (RRE)、ウッド チャック肝炎ウイルス転写規制要素 (WPRE)、コア伸長因子 1 α プロモーター (EFS ノースカロライナ州)、ベクトル包装要素 ψ (psi)、人間のサイトメガロ ウイルス (hCMV) プロモーターおよび人間の U6 プロモーター。この図の拡大版を表示するのにはここをクリックしてください。
Protocol
Representative Results
Discussion
IDLVs は生体内遺伝子編集、配信プラットフォーム22の統合と比較してこれらのベクトルに関連する突然変異誘発のリスクの低いが主因となって、遺伝性疾患のコンテキストで特にのための最適な手段として浮上し始めています。,28.28私たちの研究室で開発された最近改良されたオール ・ イン ・ ワン IDLV-CRISPR/Cas9 システム?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
デューク大学医学部、基礎科学, デューク大学の学部長のオフィス、神経生物学部に感謝したいと思います。私たちも原稿のコメントのデューク ウイルス ベクター コアのメンバーに感謝します。プラスミド pLenti CRISPRv2 は、張鋒 (ブロード研究所) からの贈り物だった。プラスミド psPAX2 を含む LV 包装システム、VSV G、pMD2.G、pRSV Rev はだったディディエ ・ Trono (スイス連邦工科大学、スイス連邦共和国) からの優しい贈り物です。この仕事のための財政支援は、大学のサウスカロライナの医学によって提供された RDF18080 E202 (B.K) を付与します。
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

