ג'ל-seq: שיטת הכנת ספריה סימולטני רצף DNA ו- RNA באמצעות מטריצות הידרוג
Summary
ג’ל-seq מאפשר חוקרים להכין בו זמנית ספריות עבור שניהם הדנ א – ו RNA-seq זניח עלות נוספת החל מ- 100-1000 תאים באמצעות מכשיר הידרוג פשוטה. המאמר מציג גישה מפורטת על הזיוף של המכשיר, כמו גם את הפרוטוקול הביולוגי כדי ליצור ספריות לזווג.
Abstract
היכולת להגביר את רצף ה-DNA או RNA מדגימות התחלה קטנה הושגה רק בחמש השנים האחרונות. למרבה הצער, פרוטוקולים סטנדרטיים עבור יצירת גנומית או ספריות transcriptomic אינם תואמים, חוקרים עליך לבחור אם רצף ה-DNA או RNA מדגם מסוים. ג’ל-seq פותר בעיה זו בכך שהוא מאפשר לחוקרים בו-זמנית להתכונן ספריות הן DNA ו- RNA החל מ 100-1000 תאים באמצעות מכשיר הידרוג פשוטה. המאמר מציג גישה מפורטת על הזיוף של המכשיר, כמו גם את הפרוטוקול הביולוגי כדי ליצור ספריות לזווג. עיצבנו ג’ל-seq. כך זה יכול להיות מיושם בקלות על ידי חוקרים אחרים; מעבדות גנטיקה רבים יש כבר את כל הציוד הדרוש כדי לשחזר את ייצור המכשיר ג’ל-seq. פרוטוקול שלנו מעסיקה נפוצות ערכות עבור שניהם הגברה שלם-תעתיק (של WTA), ספריית הכנה, אשר הם גם נוטים להיות מוכר חוקרים כבר שבקיאים גנומית ושוויונית transcriptomic ספריות. הגישה שלנו מאפשר לחוקרים להביא לשאת את הכוח של רצפי DNA ו- RNA על דוגמה אחת ללא פיצול ועם זניח בתוספת מחיר.
Introduction
הדור הבא רצף (הגדרות) יש לו השפעה עמוקה על הדרך מתנהל מחקר גנטיקה. כאשר חוקרים התמקדו פעם רצף הגנום של זן שלם, עכשיו זה אפשרי את רצף הגנום של גידול יחיד או אפילו תא בודד בניסוי אחד. 1 הגדרות גם הפכה חסכוניות כדי רצף התרשימים RNA נמצא בתוך תא, אוסף של נתונים המכונה של transcriptome. היכולת להגביר את רצף ה-DNA או RNA מדגימות התחלה קטנה הושגה רק בחמש השנים האחרונות. 2 , 3 , 4 . למרבה הצער, פרוטוקולים סטנדרטיים אינם תואמים, חוקרים עליך לבחור אם רצף ה-DNA או RNA דגימה נתון. כאשר מדגם ההתחלתי הוא גדול מספיק, ניתן לפצל אותו לשניים. קשקשים קטנים יותר, עם זאת, אובדן של חומר עקב פיצול דגימות יכולים להשפיע על איכות ספריה ולאחר איגום של דגימות יכול ממוצעים גרסאות מעניינות בין תאים. 5 יתר על כן, החוקרים מעוניינים יותר ויותר בחינת והמדגמים יכולה להתפצל, כגון תאים בודדים או ביופסיות גידול קטן הטרוגנית. 6
כדי לטפל בבעיה זו, שלושה פרוטוקולים לאחרונה פותחו כדי רצף ה-DNA ו- RNA מדגם המוצא זהה: ג’ל-seq7, G & T-seq8ו ד ר-seq9. מאמר זה מציג פרוטוקול מפורט עבור ג’ל-seq, אשר יכול לשמש כדי בו-זמנית ליצור ספריות DNA ו- RNA מתוך כמה כתאי 100 זניח עלות נוספת. ההיבט הרומן של ג’ל-seq היא היכולת להפריד בין DNA ו- RNA המבוסס באופן בלעדי על גודל באמצעות מטריצות הידרוג בעלות נמוכה. החידוש הליבה של פרוטוקול ג’ל-Seq הוא ההפרדה הפיזית של ה-DNA מ- RNA. ההפרדה הזאת מושגת electrophoretically באמצעות שילוב של ממברנות לזיהוי לנצל גודל ההבדלים בין מולקולות אלה. כדי לשים את ההבדלים בגודל בהקשר, שקול כיצד DNA ו- RNA הם צילמו: בעוד דנ א קיים בסולם מיקרון ניתן להציג באמצעות מיקרוסקופים מסורתיים, RNA קיים בסולם ננומטר ולא חייב לדימות תוך שימוש בטכניקות מורכבות כגון אלקטרונים הקפאה מיקרוסקופ. 10
הגישה הפרדת DNA ו- RNA, פרוטוקול זה מוצג באיור1. החלונית השמאלית מציגה DNA ו- RNA חינם צף בפתרון בקרבת קרום. כאשר שדה חשמלי מוחל, כפי שמוצג בלוח הימני, DNA ו- RNA לחוות כוח electrophoretic שגורם העברה דרך הקרום. על-ידי כיוונון המאפיינים ממברנה, יצרנו קרום חדיר למחצה המפריד DNA RNA. מולקולות DNA יידחפו נגד הקרום, אבל להיות מסובכת בקצה בשל גודלם גדולים. מצד שני, מולקולות RNA קטנות, ניתן להגדיר מחדש, לארוג את דרכם דרך הקרום. תהליך זה, המכונה reptation, דומה לאופן בו שנחש עובר דשא. בסופו של דבר אלה מולקולות RNA מופסקים על ידי קרום השני, בצפיפות גבוהה זה קשה מדי עבור פולימרים אפילו קטנה יותר (> 200 זוגות בסיס) להתחמק על דרך. ברגע פיזית מחוזק, DNA ו- RNA יכול להיות התאושש לעבד להפיק מידע על הגנום והן transcriptome. בעוד נוכל להפריד DNA ו- RNA, מצאנו תוצאות טובות יותר מתקבלים אם ה-RNA היא הפוכה עיבד כדי cDNA לפני ההפרדה. היברידים רבים cDNA/RNA יציבים יותר RNA לבד, עדיין יכול לעבור דרך קרום בצפיפות נמוכה.
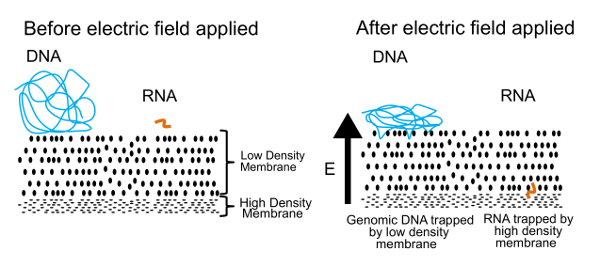
איור 1 . עקרון ההפעלה ג’ל-seq. העיקרון הבסיסי המשמש להפרדת פיזית של DNA ו- RNA. בשדה חשמלי יישומית, מולקולות RNA קטנות להעביר דרך קרום בצפיפות נמוכה אבל מולקולות DNA גדולות לכוד על פני השטח. איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
מאמר זה מתאר בפירוט בשני הזיוף של המכשיר ג’ל-seq, הפרוטוקול הביולוגי ליצירת לזווג ספריות DNA ו- RNA. סקירה כללית של שניהם מוצג באיור2. המכשיר מפוברק על ידי שכבות 3 צפיפות שונה לזיהוי ג’לים אחד על גבי השני בתהליך דומה ליצירת תקן ג’לים הערימה. 11 הפרוטוקול הביולוגי מתחיל עם תאים 100-1000 הושעו ב- PBS. התאים הם lysed, הרנ א מומר cDNA לפני המכשיר משמש להפריד את הדנ א כלאיים cDNA/RNA. לאחר ההפרדה, התאוששות, גנומית, transcriptomic ספריות מוכנות באמצעות תהליך זה מקרוב כדלקמן פרוטוקול ערכת הכנה הספרייה הגנום כולו. ניתן לקרוא פרטים נוספים על פיתוח, אימות של ג’ל-seq במעבדה על פרסום שבב “ג’ל-seq: הגנום כולו ורצף transcriptome על ידי סימולטני קלט נמוך-DNA ו- RNA ספריית הכנה באמצעות מחסומים הידרוג חדיר למחצה .” 7
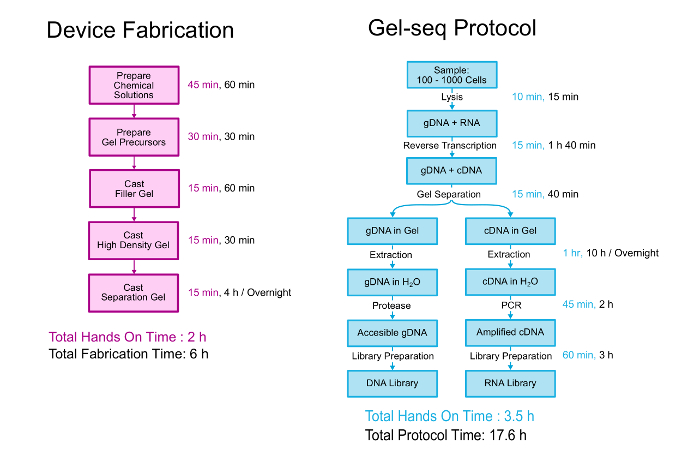
איור 2 . פרוטוקול ג’ל-seq. מבט כולל על הצעדים כדי לפברק את המכשיר ג’ל-seq והפרוטוקול מחוללות לזווג ספריות DNA ו- RNA. חלקים של איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
כדי ליצור ספריות DNA ו- RNA מתוך תאים בודדים, חוקרים, כדאי שתשקול להשתמש או G & T-seq או ד ר-תת סעיף. G & T-Seq, כמו ג’ל-seq, מסתמך על הפרדה פיזית של רנ א מ- DNA גנומי. גישה זו נסמכת על messenger RNA (mRNA) 3′ polyadenylated זנב כמטרה נפתחים. ה-mRNA נלכד על חרוז מגנטי באמצעות פריימר oligo-dT biotinylated. ברגע mRNA כבר נתפס החרוזים הם שנערך במקום עם מגנט, תגובת שיקוע המכיל את הדנ א הגנומי יכול להיות הוסר, הועבר צינור אחר. בתום ההפרדה הפיזית הזאת, ספריות שונות ניתן להפיק מן ה-mRNA ו DNA. 8 גישה זו פועלת היטב אם הרנ א עניין polyadenylated, אולם אין אפשרות להשתמש ללמוד תעתיקים הלא-polyadenylated, כגון ribosomal RNA, tRNA, או RNA של אאוקריוטים.
ד ר-seq מסתמך על צעד הגברה מראש לאן דנ א והן cDNA נגזר מן ה-RNA הם מוגבר בצינור אותו. המדגם ואז לשתיים, מעובד במקביל להכין ספריות seq DNA ו- RNA. כדי להבחין בין DNA גנומי של cDNA נגזר מן ה-RNA, ד ר-seq מקבלת גישה חישובית. רצפים היכן נמצאים רק exons מדוכאים שהמפתחות בנתונים דנ א גנומי, כמו אלה יכול להיות שמקורם מן ה-DNA או RNA. 9 היתרון בגישה זו הוא כי ה-DNA ו cDNA/RNA צריך להיות פיזית מופרדים זאת על ג’ל-seq ו G & T-תת סעיף. החיסרון, לעומת זאת, היא ד ר-seq דורשת ידע א-פריורי של הגנום, transcriptome (כלומר, exons לעומת אינטרונים), לא יהיה אידיאלי עבור יישומים כגון רצף של גרעינים, שבו תעתיקים רבים עדיין אינם לגמרי משולבים, עדיין להכיל אינטרונים. 12
ההיבט הרומן של ג’ל-seq היא היכולת להפריד בין DNA ו- RNA מאות תאים המבוסס באופן בלעדי על גודל. שיטה זו דורשת שום ידע אפריורי של הגנום או transcriptome, היא חזקה נגד שחבור לא שלם, אינה מוגבלת תעתיקים פולי-adenylated. עבור יישומים שבהם חוקר יכול להתחיל עם תאים לפחות 100, ג’ל-seq מספקת גישה ישירה באמצעות חומרים נרחב זמין וזול.
Protocol
Representative Results
Discussion
ישנם מספר שלבים קריטיים המשויך הזיוף התקן ג’ל-seq, כמו גם את הפרוטוקול עצמו. במהלך פבריקציה נוספת, אנו ממליצים החל עוביים השכבה שנקבעו עבור אזורים שונים של הג’ל. בילינו זמן משמעותי בדיקת אפשרויות ייצור שונה ומייצרת הפרוטוקול המתואר כאן ההתקנים הטוב ביותר עבור הקלטות המפורטים בטבלה של חו…
Disclosures
The authors have nothing to disclose.
Acknowledgements
מימון עבור עבודה זו היה בתנאי על-ידי באוניברסיטת סן דייגו, הלאומית למדע קרן בוגר מחקר תכנית העמיתים, NIH להעניק R01-HG007836, ועל ידי קוריאנית משרד המדע, ICT ותכנון העתיד.
גירסאות קודמות של כמה דמויות פורסמו לראשונה ב- “התקליט הזה, ג ד. et al. ג’ל-seq: הגנום כולו, רצף transcriptome על ידי סימולטני קלט נמוך-DNA ו- RNA ספריית הכנה באמצעות מחסומים הידרוג חדיר למחצה. מעבדה על שבב 17, מוריה ברטל-2630, doi:10.1039 / c7lc00430c (2017). ” מעבדה על שבב וההתנהגותיים שימוש חוזר של דמויות בפרסום זה.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

