هلام-seq: طريقة لإعداد مكتبة تسلسل واحد من الحمض النووي والجيش الملكي النيبالي باستخدام مصفوفات المائية
Summary
هلام-seq تمكن الباحثين في نفس الوقت إعداد مكتبات لكلا الحمض النووي–والحمض النووي الريبي-seq في التكلفة المضافة ضئيلة بدءاً من 100-1000 الخلايا باستخدام جهاز هيدروجيل بسيطة. وتعرض هذه الورقة نهجاً مفصلة لتصنيع الجهاز، فضلا عن البروتوكول البيولوجي لإنشاء مكتبات المزدوجة.
Abstract
إلا تحقق القدرة على تضخيم وتسلسل الحمض النووي أو الحمض النووي الريبي من عينات صغيرة في البداية في السنوات الخمس الماضية. ولسوء الحظ، البروتوكولات القياسية لتوليد الجينوم أو مكتبات ترانسكريبتوميك تتنافى والباحثين يجب أن تختار ما إذا كنت تريد تسلسل الحمض النووي الريبي أو لنموذج معين. هلام-seq يحل هذه المشكلة عن طريق تمكين الباحثين في نفس الوقت إعداد مكتبات للحمض النووي والجيش الملكي النيبالي بدءاً من 100-1000 الخلايا باستخدام جهاز هيدروجيل بسيطة. وتعرض هذه الورقة نهجاً مفصلة لتصنيع الجهاز، فضلا عن البروتوكول البيولوجي لإنشاء مكتبات المزدوجة. نحن تصميم seq جل بحيث يمكن تنفيذه بسهولة من الباحثين الآخرين؛ مختبرات علم الوراثة كثيرة لديها بالفعل المعدات اللازمة لإنتاج تصنيع الجهاز جل seq. لدينا بروتوكول يستخدم مجموعات المواد المستخدمة بشكل شائع لكلا التضخيم كل نسخة (الرغبة) وإعداد مكتبة، الذي من المرجح أيضا أن تكون مألوفة للباحثين بالفعل على دراية بتوليد الجينوم وترانسكريبتوميك المكتبات. نهجنا يسمح للباحثين لكي تتحمل سلطة تسلسل الحمض النووي والجيش الملكي النيبالي على عينة واحدة دون تقسيم ومع التكلفة المضافة لا يعتد بها.
Introduction
الجيل التالي التسلسل (خ ع) كان لها تأثير عميق على الطريقة التي تجري بحوث علم الوراثة. حيث مرة ركز الباحثين على تسلسل الجينوم أكمله الأنواع، من الممكن الآن تسلسل الجينوم من ورم واحد أو حتى خلية واحدة في تجربة واحدة. 1 خ ع أيضا جعلها فعالة من حيث التكلفة تسلسل وقائع الجيش الملكي النيبالي وجدت داخل خلية، عبارة عن مجموعة من البيانات المعروفة باسم الترنسكربيتوم. إلا تحقق القدرة على تضخيم وتسلسل الحمض النووي أو الحمض النووي الريبي من عينات صغيرة في البداية في السنوات الخمس الماضية. 2 , 3 , ولسوء الحظ، 4 البروتوكولات القياسية غير متوافقة والباحثين يجب أن تختار ما إذا كنت تريد تسلسل الحمض النووي الريبي أو لنموذج معين. عند انطلاق عينة كبيرة بما يكفي، فإنه يمكن تقسيم في النصف. في جداول صغيرة، ومع ذلك، الخسائر المادية بسبب تقسيم العينات يمكن أن يؤثر على نوعية المكتبة، وتجميع عينات يمكن متوسط من الاختلافات مثيرة للاهتمام بين الخلايا. 5 وعلاوة على ذلك، الباحثين مهتما متزايدة في فحص العينات التي لا يمكن تقسيم، مثل خلايا مفردة أو الخزعات الصغيرة الورم غير متجانسة. 6
ولمعالجة هذه المشكلة، وضعت مؤخرا ثلاثة بروتوكولات لتسلسل الحمض النووي والجيش الملكي النيبالي من نفس العينة انطلاق: جل seq7، ز & T-seq8والدكتور seq9. تعرض هذه المقالة بروتوكول مفصل لجل-seq، التي يمكن استخدامها لإنشاء مكتبات الحمض النووي والجيش الملكي النيبالي في وقت واحد من عدد قليل من الخلايا 100 في التكلفة المضافة لا يعتد بها. الجانب رواية لجل seq هو القدرة على فصل الحمض النووي والجيش الملكي النيبالي تستند حصرا إلى حجم استخدام مصفوفات المائية منخفضة التكلفة. الابتكار الأساسية للبروتوكول Seq جل هو الفصل المادي للحمض النووي من الجيش الملكي النيبالي. ويتحقق هذا الفصل اليكتروفوريتيكالي باستخدام مزيج من الأغشية polyacrylamide التي تستفيد من حجم الاختلافات بين هذه الجزيئات. لوضع هذه الاختلافات في الحجم وفي السياق، تنظر كيف يتم تصويرها الحمض النووي والجيش الملكي النيبالي: بينما الحمض النووي موجود بمقياس ميكرون، ويمكن عرضها باستخدام المجاهر التقليدية، الحمض النووي الريبي موجود بمقياس نانومتر ويجب تصويرها باستخدام تقنيات معقدة مثل البرد-إلكترون الفحص المجهري. 10
ويرد في الشكل 1النهج المتبع في فصل الحمض النووي والجيش الملكي النيبالي في هذا البروتوكول. اللوحة اليسرى يظهر الحمض النووي والجيش الملكي النيبالي حرة عائمة في الحل بالقرب غشاء. عندما يتم تطبيق مجال الكهربائي، كما هو مبين في اللوحة اليمنى، الحمض النووي، والجيش الملكي النيبالي تجربة قوة الغرواني الكهربي الحث على الهجرة من خلال الغشاء. وقد أنشأنا بضبط خصائص الغشاء، غشاء شبه منفذ يفصل الحمض النووي الريبي. جزيئات الحمض النووي يتم الضغط ضد الغشاء، ولكن أصبح متشابكاً في الحافة بسبب حجمها الكبير. جزيئات الحمض النووي الريبي صغيرة، من ناحية أخرى، يمكن تكوين ونسج طريقهم عبر الغشاء. تشبه هذه العملية، المعروفة باسم ريبتيشن، على طريقة ثعبان يتحرك من خلال العشب. في نهاية المطاف توقفت هذه جزيئات الحمض النووي الريبي غشاء عالي الكثافة، والثاني أن من الصعب جداً للبوليمرات حتى أصغر (> 200 قاعدة أزواج) التملص من خلال. مرة واحدة مفصولة ماديا، يمكن استرداد الحمض النووي والجيش الملكي النيبالي وتجهيزها لتوليد معلومات عن الجينوم والترنسكربيتوم على السواء. وفي حين أننا يمكن فصل الحمض النووي والجيش الملكي النيبالي، وجدنا يتم الحصول على نتائج أفضل إذا كان الجيش الملكي النيبالي عكس نسخها إلى كدنا قبل الانفصال. هجن كدنا/الحمض النووي الريبي هي أكثر استقرارا من الحمض النووي الريبي وحدها ولا يزال يمكن أن تمر عبر غشاء منخفضة الكثافة.
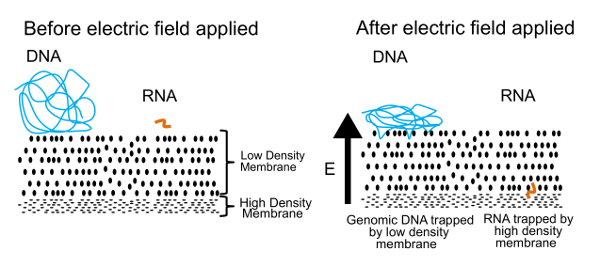
الشكل 1 . مبدأ التشغيل جل seq. والمبدأ الأساسي المستخدمة فعلياً فصل الحمض النووي والجيش الملكي النيبالي. في مجال الكهربائي المطبق، جزيئات الحمض النووي الريبي صغيرة تهاجر عبر غشاء منخفضة الكثافة ولكن جزيئات الحمض النووي كبيرة محاصرون في السطح. واستنسخ هذا الرقم من الرقم 7 بإذن من “المجتمع الملكي للكيمياء”. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
وتصف هذه الورقة بالتفصيل كلا تصنيع الجهاز seq جل وإقران البروتوكول البيولوجي لإنشاء مكتبات الحمض النووي والجيش الملكي النيبالي. ويبين الشكل 2نظرة عامة على حد سواء. الجهاز مختلق من طبقات ثلاثة مختلفة الكثافة polyacrylamide الجل فوق بعضها البعض في عملية مماثلة لإنشاء معيار الجل التراص. 11 البروتوكول البيولوجي يبدأ مع خلايا 100-1000 تعليق في برنامج تلفزيوني. يتم تفكيك الخلايا والحمض النووي الريبي يتم تحويلها إلى كدنا قبل استخدام الجهاز بفصل الدنا الجينومي الهجينة كدنا/الجيش الملكي النيبالي. بعد الانفصال، والانتعاش، والجينوم وترانسكريبتوميك تعد المكتبات باستخدام عملية أن يتابع عن كثب البروتوكول طقم الإعداد القياسي جينوم كل مكتبة. يمكن قراءة المزيد من التفاصيل حول التنمية والتحقق من الصحة لجل seq في مختبر على رقاقة منشور “seq جل: كل الجينوم وتسلسل الترنسكربيتوم عن طريق المتزامنة ذات المدخلات المنخفضة الحمض النووي والجيش الملكي النيبالي مكتبة الإعداد باستخدام الحواجز المائية شبه نفاذية .” 7
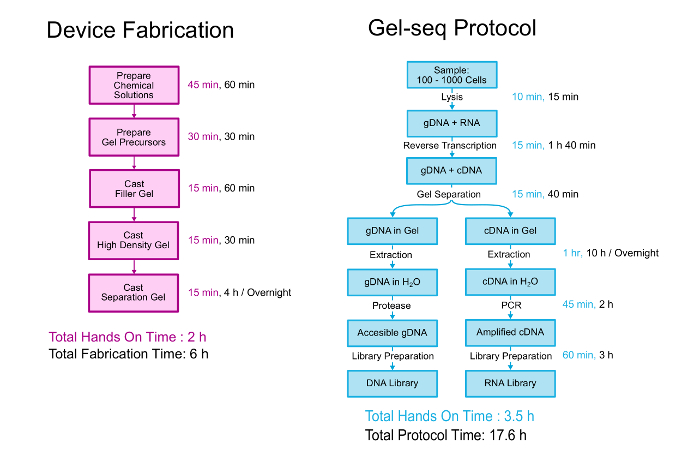
الشكل 2 . بروتوكول جل seq. إقران لمحة عامة عن الخطوات لاختلاق الجهاز seq جل والبروتوكول إنشاء مكتبات الحمض النووي والجيش الملكي النيبالي. استنسخت أجزاء من هذا الرقم من الرقم 7 بإذن من “المجتمع الملكي للكيمياء”. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
لإنشاء مكتبات الحمض النووي والجيش الملكي النيبالي من الخلايا المفردة، وينبغي النظر في استخدام أما ز & T-seq أو ما يليها الدكتور ز & T-Seq، مثل seq جل الباحثين، يعتمد على فصل المادي للجيش الملكي النيبالي من الحمض النووي. هذا النهج يعتمد على رسول رنا (مرناً) 3 ‘ بوليادينيلاتيد الذيل كهدف سحب لأسفل. يتم التقاط مرناً على حبة مغناطيسي باستخدام تمهيدي اليغو-dT بيوتينيلاتيد. متى تم التقاط مرناً الخرز وتعقد في مكان مع مغناطيس ويمكن إزالة المادة طافية تحتوي على الحمض النووي الجينومي وتحويلها إلى أنبوب آخر. بعد الانتهاء من هذا الفصل المادي، يمكن إنشاء مكتبات منفصلة من مرناً والحمض النووي. 8 هذا النهج يعمل جيدا إذا كان الجيش الملكي النيبالي للاهتمام بوليادينيلاتيد، ولكن لا يمكن استخدامه لدراسة النصوص غير بوليادينيلاتيد، مثل الرنا الريباسي، الحمض الريبي النووي النقال، أو الحمض النووي الريبي من بدائيات النوى.
الدكتور seq يعتمد على خطوة ما قبل تضخيم حيث تتضخم الحمض النووي وكدنا المستمدة من الجيش الملكي النيبالي في الأنبوب نفسه. العينة ثم تقسيم في اثنين وتجهيزها بالتوازي مع إعداد مكتبات تسلسل الحمض النووي والجيش الملكي النيبالي. للتمييز بين الحمض النووي وكدنا المستمدة من الجيش الملكي النيبالي، الدكتور seq نهجاً حسابية. تسلسلات التي تتواجد فيها إلا exons تقمع حسابياً في بيانات الحمض النووي الجينوم، كتلك التي يمكن نشأت من الحمض النووي أو الحمض النووي الريبي. ميزة هذا النهج أن الحمض النووي وكدنا/الجيش الملكي النيبالي بحاجة لا يمكن فعلياً فصل كما الحال في جل seq وز & T-ما يليها 9 العيب، ومع ذلك، هو أن الدكتور seq يتطلب معرفة مسبقة بالجينوم والترنسكربيتوم (أي، exons مقابل introns)، وقد لا تكون مثالية لتطبيقات مثل تسلسل نويات، فيها العديد من النصوص لا بعد تماما تقسم ولا يزال يحتوي على إينترونس. 12
الجانب رواية لجل seq هو القدرة على فصل الحمض النووي والجيش الملكي النيبالي في مئات خلايا استناداً إلى حجم حصرا. يتطلب هذا الأسلوب لا المعرفة مسبقاً من الجينوم أو الترنسكربيتوم، هو قوي ضد الربط غير مكتملة، ولا تقتصر على النصوص بولي-أدينيلاتيد. للتطبيقات حيث يمكن أن يبدأ باحث مع خلايا على الأقل 100، يوفر جل seq نهج مباشرة باستخدام مواد رخيصة ومتاحة على نطاق واسع.
Protocol
Representative Results
Discussion
وهناك العديد من الخطوات الهامة المرتبطة بتصنيع جهاز seq هلام، فضلا عن البروتوكول نفسه. أثناء التصنيع، ونحن نوصي بدءاً بسمك الطبقة المحددة لمختلف المناطق من الجل. قضينا قدرا كبيرا من الوقت في اختبار خيارات مختلفة من تلفيق والبروتوكول الموصوفة هنا ينتج أفضل الأجهزة لأشرطة الكاسيت المدرجة ف?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
وكان التمويل اللازم لهذا العمل المقدمة من جامعة سان دييغو، “البرنامج الوطني للعلوم مؤسسة الدراسات العليا بحوث الزمالة”، المعاهد الوطنية للصحة منح R01-HG007836، وعن طريق الوزارة الكورية للعلوم وتكنولوجيا المعلومات والاتصالات وتخطيط المستقبل.
الإصدارات السابقة من عدد من الشخصيات كانت نشرت لأول مرة في “و Hoople، زاي د et al. هلام-seq: الجامع-الجينوم وتسلسل الترنسكربيتوم عن طريق المتزامنة ذات المدخلات المنخفضة الحمض النووي والجيش الملكي النيبالي مكتبة الإعداد باستخدام الحواجز المائية شبه نفاذه. مختبر في 17 رقاقة، 2619-2630، doi:10.1039/c7lc00430c (2017). ” وقد يعاقب مختبر على رقاقة إعادة استخدام الأرقام الواردة في هذا المنشور.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

