ゲル-seq: DNA および RNA のゲルのマトリックスを使用しての同時配列ライブラリの準備の方法
Summary
ゲル seq の両方 DNA と RNA-seq 単純なハイドロゲル デバイスを用いた 100-1000 細胞からごくわずかな追加コストでライブラリを同時に準備することも可能。 にします。対ライブラリを生成する生物のプロトコルと同様に、デバイス作製のための詳細なアプローチを提案する.
Abstract
増幅し、小さい開始サンプルから DNA または RNA をシーケンスする能力は、過去 5 年間で達成されているのみ。残念なことに、ゲノムの生成やトランスクリプトーム ライブラリの標準的なプロトコルに互換性がないと、研究者は特定のサンプルの DNA または RNA をシーケンスするかどうかを選択する必要があります。ゲル seq DNA と単純なハイドロゲル デバイスを用いた 100-1000 細胞から始まる RNA の両方のライブラリを同時に準備する研究者を有効にしてこの問題を解決します。対ライブラリを生成する生物のプロトコルと同様に、デバイス作製のための詳細なアプローチを提案する.他の研究者によって簡単に実装できるようにゲル seq を設計多くの遺伝学研究所はすでにゲル seq デバイス作製を再現する必要な機器を持っています。私達のプロトコルは両方全体転写増幅 (WTA) のための一般的に使用されるキットを採用しています、ライブラリ生成ゲノムおよびトランスクリプトームに精通したライブラリの準備は、既に研究者に精通していることも多い。我々 のアプローチでは、ごくわずかな追加コストと分割せず単一サンプル、DNA と RNA の両方のシーケンスの力をもたらすために研究者をことができます。
Introduction
次世代シークエンシング (NGS) は、遺伝学研究方法の深遠な影響を与えています。ここでの研究者は、一度全体の種のゲノムに焦点を当てて、今単一の腫瘍または 1 つの実験でも単一セルのゲノムを配列することが可能です。1 NGS もしました、トランスクリプトームと呼ばれるデータのコレクションのセル内にある RNA 転写産物をシーケンス処理する費用対効果。増幅し、小さい開始サンプルから DNA または RNA をシーケンスする能力は、過去 5 年間で達成されているのみ。2,3,4は残念ながら、標準のプロトコルに互換性がないと研究者はある特定のサンプルの DNA または RNA をシーケンス処理するかどうかを選択する必要があります。開始サンプルが十分に大きい場合は、半分に分割することができます。ただし、規模が小さいサンプルの分裂のための材料の損失はライブラリの品質に影響を与えるし、細胞間の興味深いバリエーションを平均することができますサンプルのプールします。5さらに、研究者は単一のセルまたは小さな異種腫瘍生検などを分割できないサンプルを調べることに興味を持つ。6
この問題を解決する 3 つのプロトコルが最近開発された同じ開始サンプルから DNA と RNA の両方をシーケンス処理する: ゲル seq7、G & T seq8、および DR seq9。この記事は、ゲル-seq、同時にごくわずかな追加コストで 100 細胞から DNA および RNA のライブラリを生成するための詳細なプロトコルを示します。ゲル seq の小説の側面は、DNA と RNA 低コスト ハイドロゲル行列を使用して大きさだけに基づいて区別する能力です。ゲル Seq プロトコルのコア技術革新は RNA から DNA を物理的に分離します。この分離は、電気泳動によってこれらの分子のサイズ違いの活用のポリアクリルアミドの膜の組み合わせを使用して実現されます。コンテキストでこれらのサイズ違いを置く、DNA と RNA をイメージする方法を検討してください: RNA ナノメートル スケール上に存在し、低温電子などの複雑な技術を使用してイメージを作成する必要があります DNA ミクロン スケール上に存在する、伝統的な顕微鏡を使用して表示することができますが、顕微鏡検査。10
このプロトコルでは DNA と RNA を分離するアプローチは、図 1に示します。左側のパネルは、DNA および RNA の無料ソリューション膜近くに浮かんでいるを示しています。電気フィールドを適用すると、右側のパネルに示すように、DNA および RNA は膜を介して移行を誘導する電気泳動の力を体験します。膜特性を調整することにより、RNA から DNA を分離する半透膜を作成しました。DNA 分子は膜に対して押されるが、サイズが大きいので端につれ。その一方で、小さな RNA 分子を再構成し、できます膜を通して彼らの方法を織る。レプテーション、として知られているこのプロセスはヘビが草を介して移動する方法に似ています。さらに小さいポリマー難しすぎて第二に、高密度の膜によってこれらの RNA 分子を停止する最終的に (> 200 塩基対) をくねらせるため。一度物理的に分離、DNA および RNA 回収し、処理できるゲノムとトランスクリプトームの両方に関する情報を生成します。我々 は DNA と RNA を区切ることができます、我々 は分離前に cDNA に転写される RNA が逆の場合より良い結果が得られますを発見しました。CDNA/RNA 雑種は単独で RNA より安定して、低密度の膜を通して渡すことができます。
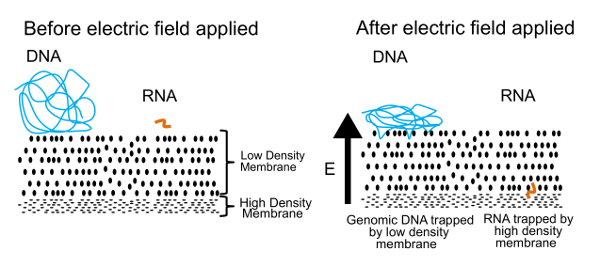
図 1.ゲル seq 動作原理。物理的に DNA と RNA を分離するために使用する基本原理。応用電界で小さな RNA 分子が低密度の膜を通って移行しますが、大きい DNA の分子が表面に閉じ込められています。この図は、王立化学協会の許可を得て参考文献 7 から再現しました。この図の拡大版を表示するのにはここをクリックしてください。
本稿で詳細にゲル seq デバイス作製を生成する生物学的プロトコル ペアに DNA および RNA のライブラリ。両方の概要を図 2に示します。レイヤー 3 つ異なる密度ポリアクリルアミドゲルで互いの上に標準的なスタッキングのゲルを作成する同じプロセスでデバイスを作製しました。11生物学的プロトコルから始まる 100-1000 細胞を PBS で中断します。細胞を分離、cDNA/RNA 雑種からゲノム DNA を分離するデバイスを使用する前に、RNA が cDNA に変換されます。後分離と回復、ゲノム、トランスクリプトーム ライブラリは、標準の全ゲノム ライブラリの準備キットのプロトコルに密着したプロセスを使用して準備されます。チップ用の文書でラボでの開発とゲル seq の検証についてさらに詳細を読むことができます”ゲル seq: 全ゲノムとトランスクリプトーム シーケンス半透性ハイドロゲル障壁を用いた同時低入力 DNA および RNA 図書館準備で.”7
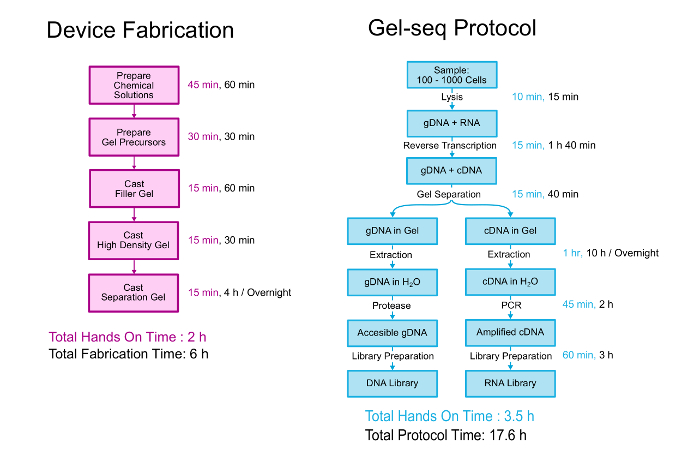
図 2.ゲル seq プロトコル。ゲル seq デバイスおよびプロトコルを生成するための手順の概要については、DNA および RNA のライブラリ ペア。この図の部分は、王立化学協会の許可を得て参考文献 7 から再現されました。この図の拡大版を表示するのにはここをクリックしてください。
単一セルからの DNA および RNA のライブラリを生成するため、研究者は G & T seq や DR seq. G
DR seq は DNA や RNA からの cDNA の両方が同じ管で増幅される中古増幅ステップに依存します。サンプルが 2 つに分割し、DNA と RNA シーケンス ライブラリを準備する並列で処理します。ゲノム DNA と RNA からの cDNA の区別は、DR seq は計算のアプローチを取る。エクソンのみが存在するシーケンスは、それらは DNA や RNA から由来している可能性がありますと計算ゲノム DNA データの抑制されます。9このアプローチの利点は、DNA と cDNA/RNA 区切る必要はありません物理的にゲル seq と G & T seq. で行われるように欠点は、ただしは、DR seq のゲノムとトランスクリプトーム (すなわちイントロンとエクソン)、事前知識を必要とし、多くの転写産物がないまだ完全に核のシーケンスなどのアプリケーションに最適なできない可能性があります。スプライシング ・ イントロンが含まれています。12
ゲル seq の小説の側面は、DNA と RNA のサイズのみに基づいてセルの数百を分離する能力です。このメソッドには、ゲノム、トランスクリプトームの先験的知識は不完全な接続に対してロバストとポリ adenylated の成績証明書に限定されていませんが必要です。研究者が少なくとも 100 セルから始まる、アプリケーション、ゲル seq は安く、広く利用可能な材料を使用して簡単な方法を提供します。
Protocol
Representative Results
Discussion
ゲル seq デバイス作製とプロトコル自体に関連付けられているいくつかの重要な手順があります。製作中にゲルの様々 な地域の所定の層の厚さで始まるをお勧めします。異なる製造オプションをテスト時間を大幅を過ごし、ここで説明したプロトコル [テーブルの材料および試薬カセットの最高のデバイスが生成されます。研究者は、代替カセット システムを使用している場合?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
この仕事のための資金は、国立科学財団大学院研究フェローシップ ・ プログラム、サン ディエゴ大学 NIH R01-HG007836 を与えると科学、ICT および将来計画の韓国大臣によって。
以前のバージョンのいくつかの数字が”・ ザ ・ フープル、g. d.らで出版された最初ゲル-seq: 全ゲノムとトランスクリプトーム シーケンスによる半透性ハイドロゲル障壁を用いた同時低入力 DNA および RNA 図書館準備によって。ラボオン チップ 17、2619-2630 doi:10.1039/c7lc00430c (2017).”ラボオン チップは、このパブリケーション内の図の再利用を認可しています。
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

