Jel-seq: DNA ve RNA hidrojel matrisleri kullanarak eşzamanlı sıralama kitaplığı hazırlanması için bir yöntem
Summary
Jel-seq kitaplıkları için her iki DNA – ve RNA-seq basit hidrojel aygıtı kullanarak 100-1000 hücreden başlayarak ihmal edilebilir ek maliyetle aynı anda hazırlamak araştırmacılar sağlar. Bu kağıt eşleştirilmiş kitaplıkları oluşturmak için biyolojik Protokolü yanı sıra cihazın imalatı için detaylı bir yaklaşım sunar.
Abstract
Yükseltmek ve DNA veya RNA küçük başlangıç örnekleri sıra yeteneği sadece son beş yıl içinde elde edilmiştir. Ne yazık ki, genomik oluşturmadan veya transcriptomic kütüphaneler için standart protokolleri uyumsuzdur ve araştırmacılar DNA veya RNA belirli bir örnek için sıralamak seçmeniz gerekir. Jel-seq aynı anda DNA ve RNA bir basit hidrojel aygıtı kullanarak 100-1000 hücrelerle başlangıç için kitaplıklarına hazırlamak araştırmacılar sağlayarak bu sorunu çözer. Bu kağıt eşleştirilmiş kitaplıkları oluşturmak için biyolojik Protokolü yanı sıra cihazın imalatı için detaylı bir yaklaşım sunar. Biz jel-seq araştırmacılar tarafından kolayca uygulanabilecek tasarlanmıştır; birçok genetik labs zaten jel-seq cihaz imalat çoğaltmak gerekli donanımları var. Bizim iletişim kuralı her iki bütün-transkript amplifikasyon (WTA) için yaygın olarak kullanılan kitleri istihdam ve de zaten araştırmacılar için tanıdık olması olasıdır Kütüphane hazırlık kitaplıkları genomik üreten ve transcriptomic usta. Bizim yaklaşım araştırmacılar DNA ve RNA sıralama gücünü ihmal edilebilir maliyet ve yarma olmadan tek bir örnek üzerinde ayı getirmek için sağlar.
Introduction
Yeni nesil (NGS) sıralama genetik araştırma yaptı yolda derin bir etkiye sahip oldu. Araştırmacılar bir kez tüm türlerin genom sıralama üzerinde duruldu nerede, şimdi tek bir tümör veya bir deneyde bile tek bir hücre genomu sıralamak mümkündür. 1 NGS, aynı zamanda maliyet etkin hücre, transcriptome bilinen veriler topluluğu içindeki bulundu RNA transkript sıralamak için sağlamıştır. Yükseltmek ve DNA veya RNA küçük başlangıç örnekleri sıra yeteneği sadece son beş yıl içinde elde edilmiştir. 2 , 3 , 4 ne yazık ki, standart protokolleri uyumsuzdur ve araştırmacılar DNA veya RNA belirli bir örnek için sıralamak seçmeniz gerekir. Başlangıç bir örnek yeterince büyük olduğunda, ikiye bölünebilir. Daha küçük ölçeklerde ancak, malzeme örnekleri bölme nedeniyle kaybı Kütüphane kalitesini etkileyebilir ve örnekleri havuzu hücreler arasında ilginç varyasyonlar dışarı ortalama olabilir. 5 Ayrıca, araştırmacılar, tek hücre veya küçük heterojen tümör biyopsi gibi bölünemez örnekleri inceleyerek giderek ilgilendi. 6
Bu sorunu çözmek için üç Protokolü son zamanlarda aynı başlangıç örneğinden DNA ve RNA sıralamak için geliştirilmiştir: jel-seq7, G & T-seq8ve DR-seq9. Bu makale aynı anda gelen önemsiz eklenen maliyet 100 hücreler olarak az DNA ve RNA kitaplıkları oluşturmak için kullanılan jel-seq için detaylı bir protokolü sunar. Roman jel-seq DNA ve RNA sadece düşük maliyetli hidrojel matrisleri kullanarak boyutuna göre ayırmak için yetenek yönüdür. RNA fiziksel ayrılması DNA’ın jel-Seq protokolünün çekirdek yeniliktir. Bu ayrılık electrophoretically bu moleküller arasındaki boyut farkları yararlanmak polyacrylamide membranlar kullanarak elde edilir. Bu boyut farkları bağlamda koymak için nasıl DNA ve RNA görüntüsü göz önünde bulundurun: DNA mikron ölçekte var ve geleneksel mikroskoplar kullanılarak görüntülenebilir, RNA nanometre ölçeğinde var ve cryo-elektron gibi karmaşık teknikleri kullanarak yansıma gerekir mikroskobu. 10
DNA ve RNA bu protokol için ayırmak için yaklaşım şekil 1‘ de gösterilen. Sol panelde çözüm bir membran yakın yüzen DNA ve RNA ücretsiz gösterir. Elektrik alan, sağ panelde gösterildiği gibi uygulandığında, DNA ve RNA membran aracılığıyla geçiş indükler bir elektroforetik güç deneyim. Membran özelliklerini ayarlama tarafından biz DNA RNA–dan ayıran yarı geçirgen bir zar oluşturduk. DNA molekülleri membran karşı itti ama kenarındaki büyük boyutlarından dolayı dolaşmış oldu. Küçük RNA molekülleri, öte yandan, yeniden yapılandırabilir ve membran yollarını örgü. Reptation bilinen bu işlem bir yılan çim hamle yol benzer. Sonunda bu RNA molekülleri daha küçük polimerler için çok zor bir ikinci, yüksek yoğunlukta membran tarafından durdurulur (> 200 baz çifti) aracılığıyla sıyrılmak için. Bir kez fiziksel olarak ayrılmış, DNA ve RNA getirilebilir kurtarıldı ve genom ve transcriptome hakkında bilgi oluşturmak için işlenmiş. DNA ve RNA ayırabilirsiniz, biz RNA cDNA ayırma önce transkripsiyonu ters ise daha iyi sonuçlar elde edilen bulduk. CDNA/RNA melez RNA yalnız daha stabildir ve hala düşük yoğunluklu membran geçebilir.
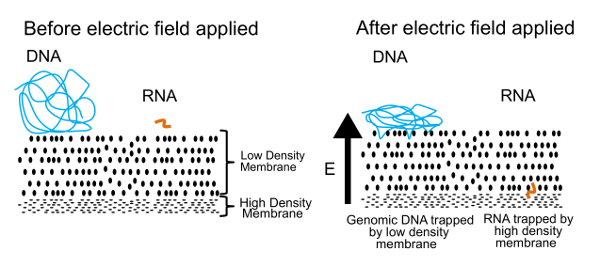
Resim 1 . Jel-seq çalışma prensibi. Fiziksel olarak DNA ve RNA ayırmak için kullanılan temel prensip. Uygulamalı elektrik alan, küçük RNA molekülleri düşük yoğunluklu membran geçiş ama büyük DNA molekülleri yüzeyde sıkışıp kalırlar. Bu rakam Ref. 7 Kimya Royal Society izniyle yeniden. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bu kağıt jel-seq cihazın her iki fabrikasyon ayrıntılı olarak açıklar ve DNA ve RNA kitaplıkları oluşturmak için biyolojik Protokolü eşleştirilmiş. Her ikisi de genel bir bakış Şekil 2‘ de gösterilmiştir. Cihazın katmanlama üç farklı yoğunluk polyacrylamide jeller üst üste çift yığın jelleri oluşturmaya benzer bir işlemde tarafından imal edilmiştir. 11 biyolojik Protokolü PBS içinde askıya 100-1000 hücreleri ile başlar. Hücreleri lysed ve cihazın genomik DNA cDNA/RNA melez ayırmak için kullanılmadan önce RNA cDNA dönüştürülür. Ayrılık ve kurtarma, genomik ve transcriptomic sonra kitaplıkları standart bütün-genom kitaplığı hazırlık seti Protokolü yakından takip eden bir işlem kullanılarak hazırlanır. Geliştirme ve jel-seq doğrulama hakkında daha fazla ayrıntı üzerinde bir çip yayın laboratuarında okumak “jel-seq: Bütün-genom ve yarı geçirgen hidrojel engelleri kullanarak eşzamanlı düşük-girdili DNA ve RNA Kütüphane hazırlık tarafından transcriptome sıralama .” 7
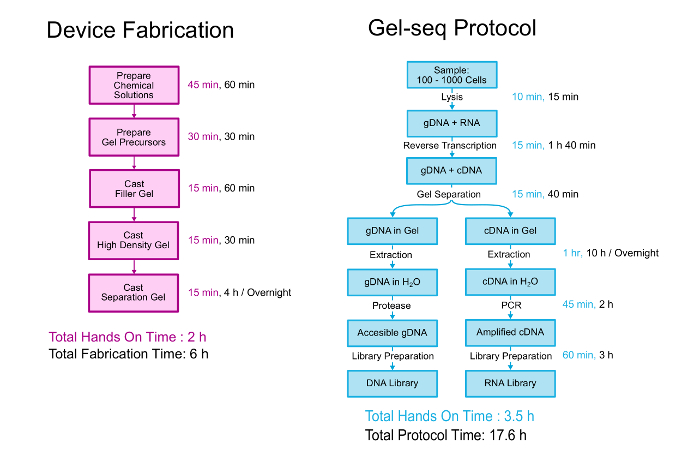
Resim 2 . Jel-seq protokolü. Sonrasında tamamlanması jel-seq aygıt ve iletişim kuralı oluşturulan eşleştirilmiş DNA ve RNA kitaplıklarına imal etmek. Bu rakam bölümlerini Ref. 7 Kimya Royal Society izniyle çoğaltılamaz. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
DNA ve RNA kitaplıkları tek hücreler üretmek için araştırmacılar G & T-seq ya da DR-devamı G & T-Seq, jel-seq gibi kullanmayı dikkate almanız gerekir, genomik DNA RNA’ın fiziksel bir renk kullanır. Bu yaklaşım mesajcı RNA üzerinde’nın (mRNA) dayanır 3’poliadenile kuyruk aşağı açılır hedef olarak. MRNA üzerinde bir manyetik boncuk biotinylated oligo-dT astar kullanarak yakalanır. MRNA yakalandıktan sonra boncuklar bir mıknatıs ile yerde tutulur ve genomik DNA içeren süpernatant kaldırıldı ve olması için başka bir tüp transfer. Bu fiziksel ayırma tamamlandıktan sonra mRNA ve DNA ayrı kütüphaneleri oluşturulabilir. 8 çalışan bu yaklaşım de ilgi RNA poliadenile ise, ancak bu poliadenile transkript, ribozomal RNA, tRNA veya RNA prokaryot üzerinden gibi çalışmak için kullanılamaz.
DR-seq DNA ve RNA türetilmiş cDNA aynı tüp nerede güçlendirilmiş bir öncesi amplifikasyon adım kullanır. Örnek sonra ikiye bölünmüş ve DNA ve RNA seq kütüphaneleri hazırlanmak için paralel olarak işlenebilir. Genomik DNA ve RNA türetilmiş cDNA arasındaki farkı anlamak, DR-seq Hesaplamalı bir yaklaşım. Bu DNA veya RNA kökenli gibi dizileri ekzonlar sadece mevcut nerede hesaplama açısından genomik DNA veri görüntülenmez. 9 cDNA/RNA ve DNA fiziksel olarak jel-seq ve G & T-seq. tamamlandý olarak ayrılması değil ki bu yaklaşımın bir avantajı olduğunu Dezavantaj, ancak, DR-seq genom ve transcriptome (Yani, ekzonlar intron karşı) bir temanın bilgi gerektirir ve çekirdekleri içinde birçok transkript henüz tam değildir, sıralama gibi uygulamalar için ideal olmayabilir olduğunu spliced ve hala intron içeriyor. 12
Roman jel-seq DNA ve RNA hücre sadece boyutuna göre yüzlerce ayırmak için yetenek yönüdür. Bu yöntem hiç bir priori bilgi genom veya transcriptome, eksik Uçbirleştirme karşı sağlamdır ve poli-adenylated transkript için sınırlı değildir gerektirir. Bir araştırmacı en az 100 hücrelerle başlayabileceğiniz uygulamalarda, jel-seq ucuz ve yaygın olarak kullanılan malzemeler kullanılarak basit bir yaklaşım sağlar.
Protocol
Representative Results
Discussion
Jel-seq aygıt uydurma gibi protokol kendisi ile ilişkili çeşitli kritik adımlar vardır. İmalat sırasında jel çeşitli bölgeleri için öngörülen katman kalınlıkları ile başlayan öneririz. Önemli zaman farklı imalat seçenekleri test geçirdim ve en iyi cihazlar malzemeler tablo ve Kimyasallarılistelenen kaset için burada açıklanan protokol üretir. Araştırmacılar bir alternatif kaset sistemi kullanıyorsanız, onlar cihazlar oluştururken kullanılan birimleri tweak için gerek…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Bu iş için fon Üniversitesi San Diego Ulusal Bilim Vakfı lisansüstü araştırma bursları, NIH hibe sağlanan R01-HG007836 ve bilim, ICT ve gelecek planlama Korece Bakanlığı tarafından yapıldı.
Birkaç rakamları önceki sürümleri ilk “Hoople, G. ö ve ark basıldı Jel-seq: Bütün-genom ve yarı geçirgen hidrojel engelleri kullanarak eşzamanlı düşük-girdili DNA ve RNA Kütüphane hazırlık tarafından transcriptome sıralama. Laboratuar bir çip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Laboratuar bir yonga üzerinde rakamlar bu yayındaki yeniden yaptırıma.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

