젤-seq: DNA와 RNA 히드로 매트릭스를 사용 하 여 동시 시퀀싱 라이브러리 준비를 위한 방법
Summary
젤-seq 연구자를 준비가 동시에 라이브러리에 대 한 두 DNA-및 RNA-seq 간단한 히드로 장치를 사용 하 여 100-1000 셀에서 시작 하는 무시할 수 추가 비용 있습니다. 이 종이 짝된 라이브러리를 생성 하는 생물 학적 프로토콜 소자의 제조에 대 한 상세한 접근을 제공 합니다.
Abstract
능력을 증폭 하 고 작은 시작 샘플에서 DNA 또는 RNA 시퀀싱만 지난 5 년간에서 달성 되었습니다. 불행히도, 게놈 생성 또는 transcriptomic 라이브러리에 대 한 표준 프로토콜 호환 되지 않습니다 그리고 연구원은 특정 샘플에 대 한 DNA 또는 RNA 순서를 선택 해야 합니다. 젤-seq 연구원은 DNA와 RNA 100-1000 셀 하이드로 겔 간단한 장치를 사용 하 여 시작에 대 한 라이브러리를 동시에 준비 하 여이 문제를 해결 합니다. 이 종이 짝된 라이브러리를 생성 하는 생물 학적 프로토콜 소자의 제조에 대 한 상세한 접근을 제공 합니다. 우리 설계 젤-seq; 다른 연구자에 의해 쉽게 구현 될 수 있도록 많은 유전학 실험실 이미 젤-seq 장치 제작을 재현 하는 필요한 장비. 우리의 프로토콜 사용 하 여 두 전체 사본 증폭 (WTA)에 대 한 키트 일반적으로 사용 되 고 라이브러리 준비는 연구원에 게 이미 익숙할 것 또한 라이브러리 생성 하 게놈 그리고 transcriptomic에 정통한. 우리의 접근 연구원을 무시할 수 추가 비용와 분할 하지 않고 단일 샘플에 DNA와 RNA 시퀀싱의 힘을가지고 있습니다.
Introduction
차세대 시퀀싱 (NGS) 유전학 연구는 실시 방법에 깊은 영향을 했다. 연구원은 한 번 전체 종의 게놈 시퀀싱에 초점을 맞춘, 단일 종양 또는 심지어 한 실험에 있는 단일 셀의 게놈을 시퀀싱 수 지금이. 1 NGS는 또한 했다 비용 효율적인 셀 데이터는 transcriptome로 알려진 컬렉션 내 RNA 사본 시퀀싱 하. 능력을 증폭 하 고 작은 시작 샘플에서 DNA 또는 RNA 시퀀싱만 지난 5 년간에서 달성 되었습니다. 2 , 3 , 4 는 불행 하 게도, 표준 프로토콜 호환 되지 않습니다 하 고 연구원은 주어진된 샘플에 대 한 DNA 또는 RNA 순서를 선택 해야 합니다. 시작 샘플 충분히 큰 경우 반으로 나눌 수 있습니다. 그러나 작은 스케일에서, 샘플 분할로 인해 재료의 손실 라이브러리 품질에 영향을 하 고 셀 사이의 재미 있는 변화 밖으로 평균 수 샘플 풀링. 5 또한, 연구팀은 단일 세포 또는 작은 다른 유형의 종양 생 검 등 분할 수 없는 샘플 검사에 점점 관심이 있습니다. 6
이 문제를 해결 하려면 세 가지 프로토콜 최근 개발 된 동일한 시작 샘플에서 DNA와 RNA 시퀀싱 하: 젤-seq7,8G & T-seq 및 박사-seq9. 이 문서는 젤-seq, 동시에 몇 가지 사소한 추가 비용 100 셀으로에서 DNA와 RNA 라이브러리를 생성 하는 데 사용할 수 있습니다에 대 한 상세한 프로토콜을 제공 합니다. 젤-seq의 소설 측면 DNA와 RNA 독점적으로 저가 히드로 매트릭스를 사용 하 여 크기에 따라 분리 하는 기능입니다. 젤-Seq 프로토콜의 핵심 혁신은 RNA에서 DNA의 물리적 분리 이다. 이 분리 electrophoretically polyacrylamide 막이이 분자의 크기 차이 활용 하는의 조합을 사용 하 여 이루어집니다. 맥락에서 이러한 크기 차이 넣어, DNA와 RNA의 이미지로 어떻게 하는 것을 고려: DNA 미크론 단위에 존재 하는 동안 전통적인 현미경을 사용 하 여 볼 수 있습니다, RNA 나노미터 규모로 존재 하 고 cryo 전자 등 복잡 한 기술을 사용 하 여 촬영 해야 합니다 현미경 검사 법입니다. 10
이 프로토콜에서 DNA와 RNA를 분리 하는 접근 방식은 그림 1에 표시 됩니다. 왼쪽된 패널 DNA와 RNA 무료 솔루션 막 근처에 떠 있는 것을 보여줍니다. 전기 분야 적용, 오른쪽 패널에 표시 된 것 처럼, DNA와 RNA는 세포 막을 통해 마이그레이션 유도 전기 이동 힘 경험. 막 속성을 조정 하 여 우리는 RNA에서 DNA를 분리 하는 반 투과성 막을 만들었습니다. DNA 분자는 멤브레인에 대 한 적용 됩니다 하지만 그들의 큰 크기 때문에 가장자리에 얽히게 된다. 작은 RNA 분자, 다른 한편으로, 재구성 고 막 통해 그들의 방법을 짜 다 수 있습니다. Reptation로 알려진이 프로세스는 잔디를 통해 뱀 이동 방법와 비슷합니다. 결국 이러한 RNA 분자는 너무 어려운 작은 고분자에 대 한 두 번째, 고밀도 막 중단 되었습니다 (> 200 기본적인 쌍)을 통해 몸부림. 일단 물리적으로 분리, DNA와 RNA 복구 고 게놈 및 transcriptome에 대 한 정보를 생성 하는 처리. 우리는 DNA와 RNA를 분리할 수 있습니다, 하는 동안 우리는 RNA는 역방향 분리 전에 cDNA를 복사할 경우 더 나은 결과 얻을 수 있습니다 나타났습니다. CDNA/RNA 하이브리드 RNA 혼자 보다 더 안정 되며 저밀도 멤브레인 통해 전달할 수 있습니다.
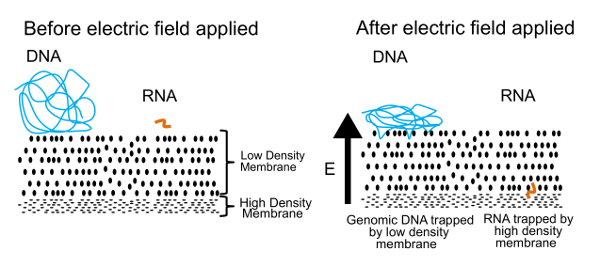
그림 1 . 젤-seq 운영 원칙. 육체적으로 DNA와 RNA를 분리 하는 데 사용 하는 기본 원리. 적용 된 전기 분야에서 작은 RNA 분자는 저밀도 막 통해 마이그레이션할 하지만 큰 DNA 분자 표면에 갇혀 있다. 이 그림은 화학의 왕 사회 허가 참고 7에서 재현 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
이 종이 젤-seq 장치 제조 자세히 설명 하 고 생성 하는 생물 학적 프로토콜 결합 DNA와 RNA 라이브러리. 둘 다에 대 한 개요는 그림 2에 표시 됩니다. 장치는 레이어 링 3 개의 서로 다른 밀도 polyacrylamide 젤 서로 표준 스태킹 젤을 만드는 유사한 과정에 의해 조작 됩니다. 11 생물 학적 프로토콜 100-1000 셀 PBS에 정지로 시작 합니다. 셀 lysed 고 RNA cDNA/RNA 하이브리드에서 게놈 DNA를 분리 하는 장치를 사용 하기 전에 cDNA로 변환 됩니다. 분리 및 복구, 게놈 및 transcriptomic 후 라이브러리 표준 전체 게놈 라이브러리 준비 키트 프로토콜을 다음과 밀접 하 게 하는 프로세스를 사용 하 여 준비가 되어 있습니다. 칩 게시 에 실험실에서 개발 및 젤-seq의 유효성 검사에 대 한 추가 세부 정보를 읽을 수 있습니다 “젤-seq: 전체 게놈 및 동시 낮은 입력 DNA와 RNA 라이브러리 준비 사용 하 여 반 투과성 히드로 장벽을 transcriptome 시퀀싱 .” 7
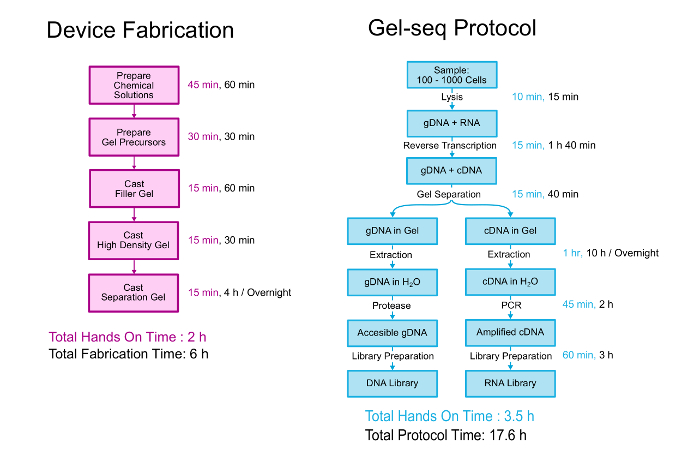
그림 2 . 젤-seq 프로토콜. 젤-seq 장치 및 생성 된 쌍을 이루는 DNA와 RNA 라이브러리에 프로토콜을 조작 하는 단계의 개요입니다. 이 그림의 일부는 화학의 왕 사회 허가 참고 7에서 재현 했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
단일 세포에서 DNA와 RNA 라이브러리를 생성 하기 위해 연구원은 G & T-seq 또는 박사가 G & T-Seq, 젤-seq 같은 사용 하 여 고려해 야, genomic DNA에서 RNA의 물리적 분리에 의존. 메신저 RNA (mRNA)에 의존 하는이 방법은 3 ‘ polyadenylated 꼬리 풀 다운 대상으로. mRNA에 자석 구슬을 biotinylated 올리고 dT 프라이 머를 사용 하 여 캡처됩니다. 일단는 mRNA를 캡처한 구슬 자석와 장소에서 개최 되 고 게놈 DNA를 포함 하는 상쾌한 수 있습니다 제거 하 고 다른 튜브로 전송. 이 물리적 분리 완료 되 면 DNA와 mRNA에서 별도 라이브러리를 생성할 수 있습니다. 그러나 8 이 이렇게 잘 관심의 RNA는 polyadenylated, 그것은 사용할 수 없습니다 ribosomal RNA, tRNA, 또는 RNA 원핵생물에서 같은 비-polyadenylated 증명서를 공부 하 작동 합니다.
박사-seq 같은 튜브에 DNA와 RNA에서 파생 하는 cDNA 증폭 됩니다 사전 증폭 단계에 의존 합니다. 샘플은 다음 둘 하 고 DNA와 RNA seq 라이브러리를 준비 하는 동시에 처리. 게놈 DNA와 RNA에서 파생 된 cDNA 사이 구별, 박사-seq 전산 접근을 걸립니다. 그 중 DNA 또는 RNA에서 유래 수로 exons는 현재 시퀀스 계산 genomic DNA 데이터에 억제 됩니다. 9 는이 방법의 장점은 그 DNA 및 cDNA/RNA 필요 하지 물리적으로 분리 될 젤-seq와 G & T이 이루어집니다. 그러나, 단점은 박사-seq 게놈 및 (즉, introns 대 exons), transcriptome 선험적 지식이 필요 하 고 이상적인 시퀀싱의 핵, 없는 많은 증명서는 아직 완벽 하 게 되지 않을 수도 있습니다. 접합 하 고 여전히 introns를 포함. 12
젤-seq의 소설 측면 DNA와 RNA 크기에 독점적으로 기반으로 셀의 수백에서 분리 하는 기능입니다. 이 메서드는 선험적 지식이 게놈 또는 transcriptome, 불완전 한 접합에 대 한 강력한 이며 폴 리 adenylated 증명서에 국한 되지 않습니다 필요 합니다. 애플리케이션에 연구원 적어도 100 셀 시작할 수 젤-seq 저렴 하 고 널리 사용 가능한 재료를 사용 하 여 간단한 접근을 제공 합니다.
Protocol
Representative Results
Discussion
젤-seq 장치 제조 뿐만 아니라 프로토콜 자체와 관련 된 몇 가지 중요 한 단계가 있습니다. 제조, 중 젤의 다양 한 지역에 대 한 소정의 레이어 두께 함께 시작 하는 것이 좋습니다. 우리는 다른 제조 옵션을 테스트 하는 중요 한 시간을 보냈다 고 여기에 설명 된 프로토콜 카세트에 테이블의 재료 및 시 약에대 한 가장 좋은 장치를 생성 합니다. 연구자는 다른 카세트 시스템을 사용 하는…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 작품에 대 한 자금 제공에 의해 대학의 샌디에고, 국립 과학 재단 대학원 연구 친교 프로그램, NIH R01-HG007836, 부여 하 고 한국 정부 과학, 정보 통신 및 미래 계획에 의해 했다.
여러 인물의 이전 버전 “Hoople, G. D. 외 에 처음 출판 되었다 젤-seq: 전체 게놈 그리고 transcriptome 시퀀싱 동시 낮은 입력 DNA와 RNA 라이브러리 준비 반 투과성 히드로 장벽을 사용 하 여. 실험실에 칩 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” 칩에 실험실이이 발행물에서 그림의 재사용을 허가 했다.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

