Gel-Seq: Eine Methode zur gleichzeitigen Sequenzierung Bibliothek Vorbereitung von DNA und RNA mit Hydrogel Matrizen
Summary
Gel-Seq ermöglicht Forschern gleichzeitig Bibliotheken für beide DNA und RNA-Seq bei vernachlässigbaren Kosten ab 100-1000 Zellen mit einem einfachen Hydrogel-Gerät vorzubereiten. Dieser Beitrag stellt einen detaillierten Ansatz für die Herstellung des Geräts sowie das biologische Protokoll zum gekoppelte Bibliotheken zu generieren.
Abstract
Die Fähigkeit zu verstärken und aus kleinen Start Proben entweder DNA oder RNA-Sequenz wurde erst in den letzten fünf Jahren erreicht. Leider die Standardprotokolle für Erzeugung von genomischen oder transkriptomischen Bibliotheken sind nicht kompatibel und Forschern müssen entscheiden, ob Sie für eine besondere Probe DNA oder RNA-Sequenz. Gel-Seq löst dieses Problem durch Forscher um gleichzeitig Bibliotheken für DNA und RNA beginnend mit 100-1000 Zellen mit einem einfachen Hydrogel Gerät vorzubereiten. Dieser Beitrag stellt einen detaillierten Ansatz für die Herstellung des Geräts sowie das biologische Protokoll zum gekoppelte Bibliotheken zu generieren. Wir konzipiert Gel-Seq so, dass es leicht von anderen Forschern umgesetzt werden könnten; viele Genetik Labore verfügen bereits über die notwendige Ausrüstung, die Gel-Seq-Gerät-Herstellung zu reproduzieren. Unser Protokoll beschäftigt häufig verwendete Kits für beide ganz-Transkript Verstärkung (WTA) und Bibliothek Vorbereitung, die auch bereits Forscher vertraut sein dürften versiert in der Erzeugung von genomischen und transkriptomischen Bibliotheken. Unser Ansatz ermöglicht Forschern, bringen die Kraft der DNA und RNA Sequenzierung auf einer einzigen Probe ohne Spaltung mit unwesentlichen zusätzlichen Kosten zu tragen.
Introduction
Nächste Generation sequencing (NGS) hatte eine profunde Auswirkung auf dem Weg, die Genetik geforscht wird. Wo Forscher einmal auf die Sequenzierung des Genoms eine ganze Spezies konzentriert, ist es nun möglich, das Genom einer einzigen Tumors oder sogar eine einzelne Zelle in einem Experiment sequentiell. 1 NGS hat auch kostengünstig zu den RNA-Transkripte gefunden innerhalb einer Zelle, eine Sammlung von Daten, bekannt als das Transkriptom Sequenzierung gemacht. Die Fähigkeit zu verstärken und aus kleinen Start Proben entweder DNA oder RNA-Sequenz wurde erst in den letzten fünf Jahren erreicht. 2 , 3 , 4 leider Standardprotokolle sind unvereinbar und Forschern müssen entscheiden, ob Sie für eine gegebene Probe DNA oder RNA-Sequenz. Wenn eine Start Stichprobe groß genug ist, kann es in zwei Hälften aufgeteilt werden. Bei kleineren Maßstäben Materialverlust durch Spaltung Proben kann Bibliothek Qualität beeinflussen und Bündelung von Proben kann durchschnittlich interessante Variationen zwischen den Zellen. 5 im übrigen interessieren sich Forscher zunehmend bei der Prüfung von Proben, die nicht können, z. B. Einzelzellen oder kleine heterogene Tumor Biopsien gespalten werden. 6
Um dieses Problem zu beheben, drei Protokolle vor kurzem wurden entwickelt, um aus der gleichen Ausgangspunkt Probe DNA und RNA Sequenz: Gel-Seq7, G & T-Seq8und DR-Seq-9. Dieser Artikel stellt ein detailliertes Protokoll für Gel-Seq, die verwendet werden, um gleichzeitig DNA- und RNA-Bibliotheken von nur 100 Zellen bei vernachlässigbaren Kosten erzeugen kann. Der neue Aspekt der Gel-Seq ist die Fähigkeit, DNA und RNA basiert ausschließlich auf Größe mit low-cost Hydrogel Matrizen zu trennen. Die Kern-Innovation des Gel-Seq-Protokolls ist die physische Trennung der DNA von RNA. Diese Trennung erfolgt elektrophoretisch mit einer Kombination aus Polyacrylamid-Membranen, die die Größenunterschiede zwischen diesen Molekülen nutzen. Um diese Größenunterschiede in Zusammenhang zu setzen, zu prüfen, wie DNA und RNA abgebildet werden: während DNA vorhanden auf der Micron-Skala ist und eingesehen werden traditionelle Mikroskopie, RNA existiert auf der Nanometerskala und komplexe Techniken wie Cryo-Elektron abgebildet werden muss Mikroskopie. 10
Der Ansatz zur Trennung von DNA und RNA in diesem Protokoll ist in Abbildung 1dargestellt. Die linke Tafel zeigt, dass DNA und RNA frei schwebend in Lösung in der Nähe von einer Membran. Wenn ein elektrisches Feld angelegt wird, wie auf der rechten Seite gezeigt, erleben DNA und RNA eine elektrophoretische Kraft, die Migration durch die Membran induziert. Durch die Membran Eigenschaften optimieren, schufen wir eine semipermeable Membran, die DNA von RNA trennt. Die DNA-Moleküle sind gegen die Membran gedrückt, aber verfangen am Rande wegen ihrer großen Größe. Auf der anderen Seite können kleine RNA-Moleküle, konfigurieren und schlängeln sich durch die Membran. Dieser Prozeß, bekannt als Reptation, ähnelt die Art, wie, die eine Schlange durch Rasen bewegt. Schließlich sind diese RNA-Moleküle durch eine zweite, mit hoher Dichte Membran, die zu schwierig für noch kleinere Polymere ist gestoppt (> 200 Basenpaaren), durch Winden. Einmal physisch getrennt, kann um Informationen über das Genom und Transkriptom zu generieren DNA und RNA erholt und verarbeitet werden. Während wir DNA und RNA trennen können, haben wir gefunden, dass bessere Ergebnisse erzielt werden, wenn die RNA transkribiert, cDNA vor der Trennung umgekehrte ist. Die DNA/RNA-Hybriden sind stabiler als RNA allein und können noch durch die Dichte Membran passieren.
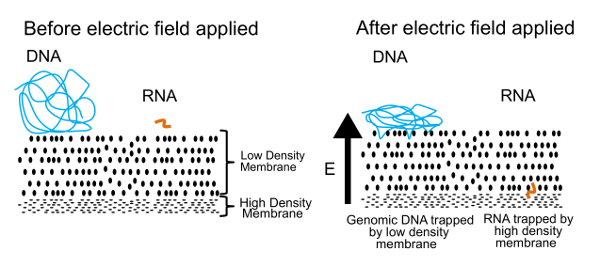
Abbildung 1 . Gel-Seq operative Prinzip. Das zugrunde liegende Prinzip verwendet, um räumliche Trennung von DNA und RNA. In einem angewandten elektrischen Feld kleine RNA-Moleküle Wandern durch die Dichte Membran aber große DNA-Moleküle sind an der Oberfläche gefangen. Diese Zahl wurde mit freundlicher Genehmigung von der Royal Society of Chemistry von ref 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Dieses Papier beschreibt ausführlich sowohl die Herstellung von Gel-Seq-Gerät und das biologische Protokoll generieren gepaart DNA- und RNA-Bibliotheken. Eine Übersicht der beiden ist in Abbildung 2dargestellt. Das Gerät ist durch Schichtung drei unterschiedliche Dichte Polyacrylamid Gele übereinander in einem Prozess ähnlich erstellen standard Stapeln Gele hergestellt. 11 das biologische Protokoll beginnt mit 100-1000 Zellen mit PBS-Puffer suspendiert. Die Zellen lysiert und die RNA in cDNA umgewandelt wird, bevor das Gerät verwendet wird, um die genomische DNA aus der DNA/RNA-Hybriden zu trennen. Nach der Trennung und Verwertung, genomische und transkriptomischen sind Bibliotheken zubereitet einen Prozess, der das gesamte Genom Standardbibliothek Vorbereitung Kit Protokoll verfolgt. Weitere Details über die Entwicklung und Validierung von Gel-Seq abgelesen werden im Labor auf einem Chip-Veröffentlichung “Gel-Seq: Vollständiggenom und Transkriptom Sequenzierung durch gleichzeitige Low-Eingang DNA- und RNA-Bibliothek Vorbereitung mit semi-permeable Hydrogel Barrieren .” 7
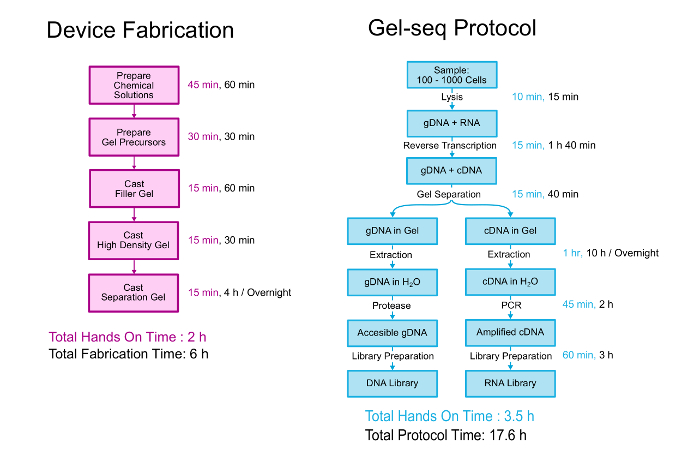
Abbildung 2 . Gel-Seq Protokoll. Eine Übersicht über die Schritte, die Gel-Seq-Gerät und das Protokoll zum generierten gepaarten DNA und RNA Bibliotheken zu fabrizieren. Teile davon wurden mit freundlicher Genehmigung von der Royal Society of Chemistry von Ref. 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Um DNA und RNA Bibliotheken aus einzelnen Zellen zu generieren, Forscher sollten erwägen, entweder G & T-Seq oder DR-f-G & T-FF, wie Gel-Seq, stützt sich auf eine räumliche Trennung von RNA aus genomischer DNA. Dieser Ansatz stützt sich auf Boten-RNA (mRNA) 3′ Polyadenylated Schweif als Pulldown-Ziel. Die mRNA wird auf eine magnetische Perle biotinylierte Oligo-dT Grundierung mit erfasst. Sobald die mRNA eingefangen hat die Perlen mit einem Magneten in Position gehalten werden und der Überstand mit der genomischen DNA entfernt und in einem anderen Gefäß übertragen werden kann. Nachdem diese körperliche Trennung abgeschlossen ist, können separate Bibliotheken aus der mRNA und DNA generiert werden. 8 dieser Ansatz funktioniert gut, wenn die RNA von Interesse Polyadenylated ist, kann nicht jedoch es verwendet werden, nicht Polyadenylated Abschriften, wie ribosomaler RNA, tRNA oder RNA von Prokaryoten zu studieren.
DR-Seq stützt sich auf eine Vorverstärkung Schritt wo DNA und cDNA abgeleitet von RNA in der gleichen Röhre verstärkt werden. Die Probe wird dann in zwei Teile gespalten und parallel, DNA und RNA-Seq Bibliotheken vorzubereiten verarbeitet. Zur Unterscheidung zwischen genomischer DNA und die cDNA abgeleitet von RNA, nimmt DR-Seq einen rechnerischen Ansatz. Sequenzen, in denen nur Exons vorhanden sind werden rechnerisch in der genomischen DNA-Daten, unterdrückt, wie diejenigen aus entweder DNA oder RNA entstanden sein könnte. 9 ein Vorteil dieses Ansatzes ist, dass die DNA und DNA/RNA nicht räumlich getrennt sein müssen wie in Gel-Seq und G & T-seq. Der Nachteil ist jedoch, dass DR-Seq a-priori Kenntnis des Genoms und Transkriptom (d.h. Exons und Introns erfordert), und möglicherweise nicht ideal für Anwendungen wie z. B. Sequenzierung von Kernen, in denen viele Transkripte noch nicht vollständig sind gespleißt und weiterhin enthalten Introns. 12
Der neuartige Aspekt des Gel-Seq ist die Fähigkeit, DNA und RNA in Hunderten von Zellen ausschließlich anhand der Größe trennen. Diese Methode erfordert kein priori Kenntnis des Genoms oder Transkriptom, ist robust gegen unvollständige Spleißen und beschränkt sich nicht auf Poly-Adenylated-Protokolle. Für Anwendungen, wo ein Forscher mit mindestens 100 Zellen beginnen kann, bietet Gel-Seq ein einfaches Verfahren mit billigen und allgemein zugänglichen Materialien.
Protocol
Representative Results
Discussion
Es gibt mehrere wichtige Schritte, die mit der Gel-Seq-Gerät-Fertigung sowie das Protokoll selbst verbunden. Während der Fertigung empfiehlt es sich, beginnend mit der vorgeschriebenen Schichtdicken für die verschiedenen Regionen des Gels. Wir verbrachten viel Zeit verschiedene Herstellung Testmöglichkeiten und das hier beschriebene Protokoll produziert die besten Geräte für die Kassetten, die in der Tabelle der Werkstoffe und Reagenzienaufgeführt. Wenn Forscher eine alternative Kassettensystem ve…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Mittel für diese Arbeit wurde von der Universität von San Diego, die National Science Foundation Graduate Research Fellowship Program, NIH R01-HG007836 gewähren, und vom koreanischen Ministerium für Wissenschaft, IKT und Zukunft planen.
Frühere Versionen von ein paar Zahlen wurden zuerst in “Hoople, G. D. Et Al. veröffentlicht. Gel-Seq: Vollständiggenom und Transkriptom Sequenzierung durch gleichzeitige Low-Eingang DNA- und RNA-Bibliothek Vorbereitung mit semi-permeable Hydrogel Barrieren. Lab-on-Chip 17, 2619-2630, Doi:10.1039 / c7lc00430c (2017). ” Labor auf einem Chip hat die Wiederverwendung von Abbildungen in dieser Publikation sanktioniert.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

