Gel-seq: Um método para a preparação de biblioteca simultânea de sequenciamento de DNA e RNA usando matrizes de hidrogel
Summary
Gel-seq permite aos investigadores simultaneamente preparar bibliotecas para ambos DNA – e RNA-seq insignificante custo adicionado a partir de 100-1000 células usando um dispositivo simples de hidrogel. Este trabalho apresenta uma abordagem detalhada para a fabricação do dispositivo, bem como o protocolo biológico para gerar bibliotecas emparelhadas.
Abstract
A capacidade de amplificar e sequenciar o DNA ou RNA de pequenas amostras de partida só foi alcançada nos últimos cinco anos. Infelizmente, os protocolos padrão para gerar genômica ou transcriptomic bibliotecas são incompatíveis e pesquisadores devem escolher-se para sequenciar o DNA ou RNA para uma determinada amostra. Gel-seq resolve esse problema permitindo que pesquisadores preparar simultaneamente bibliotecas para DNA e RNA, começando com 100-1000 células usando um dispositivo simples de hidrogel. Este trabalho apresenta uma abordagem detalhada para a fabricação do dispositivo, bem como o protocolo biológico para gerar bibliotecas emparelhadas. Nós projetamos Gel-seq para que ele pode ser facilmente implementado por outros pesquisadores; muitos laboratórios de genética já tem o equipamento necessário para reproduzir a fabricação do dispositivo de Gel-seq. Nosso protocolo emprega comumente usados kits para ambos os todo-transcrição de amplificação (WTA) e preparação da biblioteca, que também são susceptíveis de ser familiar para os investigadores já versado na geração de genômica e transcriptomic bibliotecas. Nossa abordagem permite que os pesquisadores trazer para suportar o poder de sequenciamento de DNA e o RNA em uma única amostra sem divisão e com custo insignificante adicionado.
Introduction
Próxima geração (NGS) de sequenciamento teve um profundo impacto no caminho a pesquisa genética é realizada. Onde pesquisadores uma vez focada no sequenciamento do genoma de uma espécie inteira, é agora possível sequenciar o genoma de um tumor único ou até mesmo uma única célula em um experimento. 1 NGS também tornou rentável para sequenciar os transcritos de RNA encontrados dentro de uma célula, uma coleção de dados conhecidos como a transcriptoma. A capacidade de amplificar e sequenciar o DNA ou RNA de pequenas amostras de partida só foi alcançada nos últimos cinco anos. 2 , 3 , 4 infelizmente, protocolos padrão são incompatíveis e pesquisadores devem escolher-se para sequenciar o DNA ou RNA para uma determinada amostra. Quando uma amostra inicial é grande o suficiente, ele pode ser dividido ao meio. Em escalas menores, no entanto, perda de material devido a separação de amostras pode afetar a qualidade da biblioteca, e pool de amostras pode média variações interessantes entre as células. 5 além disso, os pesquisadores estão cada vez mais interessados em examinar as amostras que não podem ser divididas, como células únicas ou biópsias de pequeno tumor heterogêneo. 6
Para resolver este problema, recentemente foram desenvolvidos três protocolos para sequenciar o DNA e o RNA da mesma amostra inicial: Gel-seq7, G & T-seq8e DR-seq9. Este artigo apresenta um protocolo detalhado para Gel-seq, que pode ser usado para gerar simultaneamente bibliotecas de DNA e RNA de apenas 100 células no custo adicionado insignificante. O aspecto inovador do Gel-seq é a capacidade de separar o DNA e RNA baseado exclusivamente no tamanho usando matrizes de hidrogel de baixo custo. A inovação do núcleo do protocolo Gel-Seq é a separação física do DNA do RNA. Esta separação é alcançada electrophoretically usando uma combinação de membranas de poliacrilamida que aproveitam as diferenças de tamanho entre estas moléculas. Para colocar estas diferenças de tamanho em contexto, considere como o DNA e RNA são fotografada: enquanto o ADN existe na escala de mícron e pode ser visualizado usando microscópios tradicionais, RNA existe na escala de nanômetros e deve ser fotografado usando técnicas complexas como cryo-elétron microscopia. 10
A abordagem para separação de DNA e RNA neste protocolo é mostrada na Figura 1. O painel esquerdo mostra DNA e RNA livram flutuação em solução perto de uma membrana. Quando um campo elétrico é aplicado, como mostrado no painel da direita, DNA e RNA experimentam uma força eletroforética que induz a migração através da membrana. Ajustando as propriedades da membrana, nós criamos uma membrana semipermeável que separa o DNA do RNA. As moléculas de DNA são empurradas contra a membrana, mas tornar-se enredado na borda, devido ao seu grande tamanho. Pequenas moléculas de RNA, por outro lado, podem reconfigurar e tecer sua maneira através da membrana. Este processo, conhecido como reptation, é similar à maneira que uma cobra se move pela grama. Eventualmente, estas moléculas de RNA são interrompidas por uma membrana de alta densidade, segunda que é muito difícil para polímeros ainda menores (> 200 pares de bases) de esquivar-se através de. Uma vez que fisicamente separados, DNA e RNA podem ser recuperado e processadas para gerar informação sobre o genoma e transcriptoma. Enquanto podemos separar DNA e RNA, encontramos melhores resultados são obtidos se o RNA é reverso transcrito de cDNA antes da separação. Os híbridos de cDNA/RNA são mais estáveis que o RNA sozinho e ainda podem passar através da membrana de baixa densidade.
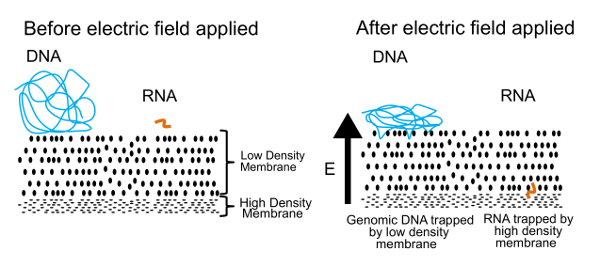
Figura 1 . Princípio de funcionamento de gel-seq. O princípio subjacente usado para separar fisicamente o DNA e RNA. Em um campo elétrico aplicado, pequenas moléculas de RNA migram através da membrana de baixa densidade, mas grandes moléculas de DNA estão presas na superfície. Esta figura foi reproduzida da ref. 7 com a permissão da Real Sociedade de química. Clique aqui para ver uma versão maior desta figura.
Este paper descreve detalhadamente tanto a fabricação do Gel-seq dispositivo e o protocolo biológico para gerar emparelhado bibliotecas de DNA e RNA. Uma visão geral de ambos é mostrada na Figura 2. O dispositivo é fabricado pela estratificação três diferentes densidade geles de polyacrylamide em cima do outro em um processo semelhante à criação de gel de empilhamento padrão. 11 o protocolo biológico começa com 100-1000 células suspendidas em PBS. As células lysed e o RNA é convertido em cDNA antes que o dispositivo é usado para separar os híbridos de cDNA/RNA o DNA genômico. Após a separação e recuperação, genômica e transcriptomic bibliotecas são preparadas com um processo que segue de perto o protocolo de kit de preparação de biblioteca padrão do inteiro-genoma. Mais detalhes sobre o desenvolvimento e validação de Gel-seq podem ser lido no laboratório em uma publicação de Chip “Gel-seq: do inteiro-genoma e transcriptoma sequenciamento por simultânea baixa-entrada DNA e RNA biblioteca preparação usando barreiras semi-permeáveis hidrogel .” 7
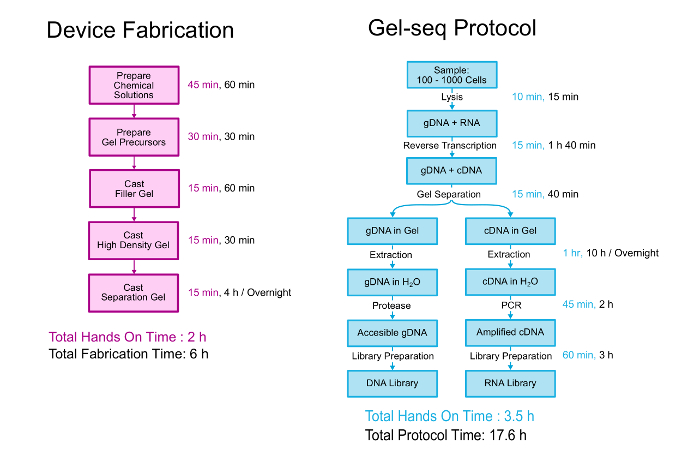
Figura 2 . Protocolo de gel-seq. Uma visão geral das etapas para fabricar o dispositivo Gel-seq e o protocolo gerado emparelhado bibliotecas de DNA e RNA. Porções desta figura foram reproduzidas de ref. 7 com a permissão da Real Sociedade de química. Clique aqui para ver uma versão maior desta figura.
Para gerar bibliotecas de DNA e RNA de células únicas, pesquisadores devem considerar o uso de G & T-seq ou Seq. DR G & T-Seq, como Gel-seq, se baseia em uma separação física do RNA do DNA genômico. Esta abordagem baseia-se no RNA mensageiro (mRNA) 3 ‘ polyadenylated cauda como um destino de suspenso. O mRNA é capturada em um grânulo magnético usando um primer de oligo-dT biotinilado. Uma vez que o mRNA foi capturado os grânulos são prendidos no lugar com um imã e o sobrenadante contendo o DNA genômico pode ser removido e transferido para outro tubo. Após concluir esta separação física, bibliotecas separadas podem ser geradas do mRNA e DNA. 8 esta abordagem funciona bem se o RNA de interesse é polyadenylated, porém não pode ser usado para estudar não-polyadenylated transcrições, tais como o RNA ribossomal, tRNA ou RNA de procariontes.
DR-seq baseia-se em uma etapa de pré-amplificação onde tanto o DNA e o cDNA derivados de RNA são amplificados no mesmo tubo. A amostra é então dividida em dois e processada em paralelo para preparar as bibliotecas de DNA e RNA-seq. Para distinguir entre DNA genômico e o cDNA derivados de RNA, DR-seq adota uma abordagem computacional. Sequências onde apenas exões estão presentes são computacionalmente suprimidas nos dados de DNA genômicos, como aqueles poderiam ter se originado de DNA ou RNA. 9 uma vantagem dessa abordagem é que o DNA e RNA de cDNA/precisam não ser fisicamente separados como é feito em Gel-seq e G & T-Seq A desvantagem, no entanto, é que DR-seq requer conhecimento a priori do genoma e transcriptoma (ou seja, exões contra os intrões) e talvez não seja ideal para aplicações tais como sequenciamento dos núcleos, no qual muitas transcrições ainda não são totalmente emendados e ainda contêm intrões. 12
O aspecto inovador do Gel-seq é a capacidade de separar o DNA e RNA em centenas de células com base exclusivamente em tamanho. Este método requer um priori nenhum conhecimento do genoma ou do transcriptoma, é robusto contra emenda incompleta e não está limitado a transcrições de poli-adenylated. Para aplicações onde um pesquisador pode começar com pelo menos 100 células, Gel-seq fornece uma abordagem simples, usando materiais baratos e amplamente disponível.
Protocol
Representative Results
Discussion
Existem vários passos críticos associados com a fabricação do dispositivo de Gel-seq, bem como o protocolo em si. Durante a fabricação, recomendamos começar com as espessuras de camada prescritos para as diversas regiões do gel. Passamos um tempo significativo teste opções de fabricação diferentes e o protocolo descrito aqui produz os melhores dispositivos para as fitas listadas na tabela de materiais e reagentes. Se pesquisadores usam um sistema alternativo de gaveta, eles podem ser necessá…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Financiamento para este trabalho foi fornecido pela Universidade de San Diego, a National Science Foundation Graduate Research Fellowship Program, NIH grant R01-HG007836 e pelo Ministério da ciência, TIC e planejamento futuro coreano.
Versões anteriores de uma várias figuras foram publicadas primeiramente em “Hoople, G. D. et al . Gel-seq: do inteiro-genoma e transcriptoma sequenciamento por simultânea baixa-entrada DNA e RNA biblioteca preparação usando barreiras semi-permeáveis hidrogel. Laboratório em um Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Laboratório em um Chip sancionou a reutilização das figuras nesta publicação.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

