分子弹簧常量分析由生物元原力探针光谱
Summary
生物元力探针 (BFP) 是一种 原位 动态力光谱 (DFS) 技术。BFP可用于测量活细胞分子相互作用的弹簧常数。此协议对 BFP 检测到的分子键进行弹簧常量分析。
Abstract
生物元原力探针(BFP)最近成为一种原生细胞表面或 原位 动态力光谱(DFS)纳米图,可以测量单分子结合动力学,评估配体受体相互作用的机械特性,可视化蛋白质动态构象变化,更令人振奋地阐明受体介导细胞机械增效机制。最近,BFP被用来测量分子键的弹簧常数。此协议描述了执行分子弹簧恒定 DFS 分析的分步过程。具体来说,讨论了两种 BFP 操作模式,即珠子单元和珠珠模式。此协议侧重于从 DFS 原始数据中提取分子键和细胞的弹簧常数。
Introduction
作为活细胞DFS技术,BFP工程人类红血球(RBC:图1)进入一个超敏感和可调谐的力量传感器与兼容的弹簧恒定范围在0.1-3 pN/nm1,2,3。为了探测配体受体相互作用,BFP 使 DFS 测量在 +1 pN (10-12 N), +3 nm (10-9米) 和 +0.5 ms (10-3 s) 生效, 空间和时间分辨率4,5.其实验配置由两个对立的微管,即探测器和目标组成。探针微管吸气RBC和珠子粘在其顶点通过生物素-链球菌素的相互作用。珠子上涂有感兴趣的配体(图1A)。目标微管吸气细胞或带有利益受体的珠子,分别对应珠细胞(图1B)和珠珠(图1C)模式。
BFP的建造,组装和DFS实验协议被详细描述之前1,6。简言之,BFP 触摸周期由 5 个阶段组成:方法、因平格、接触、缩回和分离(图 1D)。水平 RBC 顶点位置表示为+xRBC。在开始时,未应力(零力)RBC 变形=xRBC为 0(表 1)。然后,目标由压电翻译驱动,以冲压和缩回从探针珠(图1D)。RBC 探头首先由目标压缩,具有负 RBC 变形+xRBC < 0。在债券事件中,缩回阶段从压缩阶段过渡到拉伸阶段,正 RBC 变形=xRBC > 0 (图 2C和D)。根据胡克定律,BFP 承载力可以测量为F = k RBC ×xRBC,其中kRBC (表1)是 BFP 的 RBC 弹簧常数。当键破裂并完成一个触摸周期后,探针珠返回到零力位置与+xRBC = 0 (图1D)。
为了确定kRBC,我们测量并记录探针微管内孔(Rp)、RBC(R0)和 RBC 和探针珠之间的圆形接触区域(Rc)的半径(图1A)。然后kRBC根据埃文的模型(Eq.1)7,8使用实验室VIEW程序,作为一个虚拟仪器(VI)来操作BFP(图S1A)8,9。
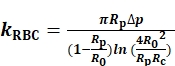 (Eq. 1)
(Eq. 1)
通过建立 BFP 和获得 DFS 原始数据,我们介绍如何分析配体受体对或细胞的弹簧常数。DFS关于糖化蛋白Thy-1和K562细胞轴承集成蛋白相互作用的原始数据α 5 β 1(Thy-1-α 5 β 1: 数字3A和3B)10和纤维蛋白和珠涂层的集成物α IIb β 3(FGN-α IIb β 3: 图3C)11,12已分别用于演示珠细胞和珠珠分析模式。
BFP 实验准备
有关 BFP 实验制备和仪器的详细信息,请参阅先前发布的协议3。简言之,人类RBC在碳/碳酸氢盐缓冲中使用了生物素-PEG3500-NHS进行生物素化。感兴趣的蛋白质与磷酸盐缓冲器中使用MAL-PEG3500-NHS的玻硅酸盐玻璃珠共价耦合。为了附着在生物素化RBC上,探针珠还使用MAL-SA涂上链球菌素(SA)。请参阅材料表和表2。
为了组装BFP(图1,左),第三个称为”帮手”的微管将用于交付探针珠,并粘附到RBC的顶点1,3。SA 涂层探针珠和生物镀金 RBC 之间的共价相互作用比配体受体的利益纽带强得多。因此,分离阶段可以解释为配体受体键破裂,而不是探针珠从RBC分离。
Protocol
Representative Results
Discussion
总之,我们提供了详细的数据分析协议,用于在 BFP 珠子和珠子细胞分析模式下对 DFS 原始数据进行预处理并衍生分子弹簧常数。介绍了确定分子和细胞弹簧常数所需的生物力学模型和方程。尽管研究的集成物不同,但珠珠模式和珠细胞模式测量的kmol具有显著的范围差异(图3A与图3C)。值得注意的是,与珠珠模式,受体是共同连接到玻?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
我们感谢纪劳姆·特罗亚德茨的有益讨论,王子豪的硬件咨询,悉尼制造中心,格雷格·苏宁和西蒙·林格支持我们的实验室启动。这项工作得到了澳大利亚研究理事会发现项目(DP200101970 – 洛杉矶J.),新南威尔士州心血管能力建设计划(中早期职业研究员格兰特 – 洛杉矶J.),悉尼研究加速器奖(SOAR – 洛杉矶),拉马乔蒂的支持 基金会健康投资赠款(2020HIG76 – 洛杉矶J.)、国家健康和医学研究理事会创意赠款(APP2003904 – L.A.J.)和悉尼大学工程学院启动基金和主要设备计划(L.A.J.)。李宁·阿诺德·朱是澳大利亚研究理事会DECRA研究员(DE190100609)。
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

