Moleculaire veerconstante analyse door biomembrane force probe spectroscopie
Summary
Een biomembrane force probe (BFP) is een in situ dynamic force spectroscopy (DFS) techniek. BFP kan worden gebruikt om de veerconstante van moleculaire interacties op levende cellen te meten. Dit protocol presenteert veerconstante analyse voor moleculaire bindingen gedetecteerd door BFP.
Abstract
Een biomembrane force probe (BFP) is onlangs naar voren gekomen als een native-cell-surface of in situ dynamic force spectroscopy (DFS) nanotool die single-molecular binding kinetics kan meten, mechanische eigenschappen van ligand-receptor interacties kan beoordelen, eiwitdynamische conformatieveranderingen kan visualiseren en receptor gemedieerde celmechanosensingmechanismen spannender kan ophelderen. Meer recent is BFP gebruikt om de veerconstante van moleculaire bindingen te meten. Dit protocol beschrijft de stapsgewijze procedure voor het uitvoeren van moleculaire veerconstante DFS-analyse. Specifiek worden twee BFP-werkingsmodi besproken, namelijk de Bead-Cell- en Bead-Bead-modi. Dit protocol richt zich op het afleiden van veerconstanten van de moleculaire binding en cel uit DFS-ruwe gegevens.
Introduction
Als een live-cell DFS-techniek ontwikkelt BFP een menselijke rode bloedcel (RBC; Figuur 1) in een ultragevoelige en afstembare krachtopnemer met een compatibel veerconstantebereik bij 0,1-3 pN/nm1,2,3. Om ligand-receptorinteractie te onderzoeken, maakt BFP DFS-metingen mogelijk bij ~ 1 pN (10-12 N), ~ 3 nm(10 -9 m) en ~ 0,5 ms (10-3 s) in kracht, ruimtelijke en temporele resolutie4,5. De experimentele configuratie bestaat uit twee tegengestelde micropipettes, namelijk de Probe en het doelwit. De Probe micropipette aspirateert een RBC en een kraal wordt aan de top gelijmd via een biotine-streptavidin interactie. De kraal is bedekt met het ligand van belang (figuur 1A). De doelmicropipette aspirateert een cel of een kraal met de receptor van belang, overeenkomend met de modi Bead-Cell (Figuur 1B) en Bead-Bead (Figuur 1C) respectievelijk5.
BFP constructie, assemblage en de DFS experimentele protocollen werden eerder in detail beschreven1,6. In het kort bestaat een BFP-aanraakcyclus uit 5 fasen: Approach, Impinge, Contact, Retract en Dissociate(Figuur 1D). De horizontale RBC-toppositie wordt aangeduid als ΔxRBC. In het begin is de onbelaste (nulkracht) RBC-vervorming ΔxRBC 0 (tabel 1). Het doel wordt vervolgens aangedreven door een piëzotranslator om de sondekraal te impeneren en terug te trekken(figuur 1D). De RBC-sonde wordt eerst gecomprimeerd door het doel met negatieve RBC-vervorming ΔxRBC < 0. In een Bond-gebeurtenis gaat de Retract-fase over van een druk- naar een trekfase met positieve RBC-vervorming ΔxRBC > 0 (figuur 2C en D). Volgens de wet van Hooke kan de BFP-draagkracht worden gemeten als F = kRBC × ΔxRBC, waarbij kRBC ( tabel1) de RBC-veerconstante van de BFP is. Na het scheuren van de binding en het voltooien van de one-touch cyclus, keert de sondekraal terug naar de nulkrachtpositie met ΔxRBC = 0 (Figuur 1D).
Om de kRBCte bepalen, meten en registreren we de radii van de binnenste opening van de sondemicropipette (Rp), de RBC (R0) en het cirkelvormige contactgebied (Rc) tussen de RBC en de sondekraal ( Figuur1A). Vervolgens wordt kRBC berekend volgens het model van Evan (Eq. 1)7,8 met behulp van een LabVIEW-programma dat fungeert als een virtueel instrument (VI) om de BFP te bedienen (Figuur S1A)8,9.
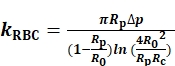 (Eq. 1)
(Eq. 1)
Met een BFP vastgesteld en DFS ruwe gegevens verkregen, presenteren we hierbij hoe de veerconstante van ligand-receptorpaar of cellen te analyseren. De DFS ruwe gegevens over de interactie van het geglycosyleerde eiwit Thy-1 en K562 celdragende integrine α5β1 (Thy-1-α5β1; Figuren 3A en 3B)10 en die van fibrinogeen en met kralen bekleed integrine αIIbβ3 (FGN-αIIbβ3; Figuur 3C) 11,12 zijn gebruikt om respectievelijk de Bead-Cell en Bead-Bead analysemodi te demonstreren.
BFP Experimentele Voorbereiding
Voor meer informatie over de experimentele voorbereiding en instrumentatie van BFP verwijzen wij u naar de eerder gepubliceerde protocollen3. Kortom, humane RBC is gebiotinyleerd met behulp van de Biotine-PEG3500-NHS in de koolstof / bicarbonaatbuffer. Interessante eiwitten zijn covalent gekoppeld aan de borosilicaatglasparels met behulp van MAL-PEG3500-NHS in de fosfaatbuffer. Om zich aan de gebiotinyleerde RBC te hechten, wordt de sondekraal ook bedekt met streptavidin (SA) met behulp van de MAL-SA. Zie de tabel met materialen en tabel 2.
Om de BFP te assembleren(figuur 1, links),zal de derde micropipette met de term ‘Helper’ worden gebruikt om de sondekraal af te leveren en te lijmen op de top1,3vande RBC. De covalente interactie tussen de SA-gecoate sondekraal en gebiotinyleerde RBC is veel sterker dan de ligand-receptorbinding van belang. Het dissociate stadium kan dus worden geïnterpreteerd als de ligand-receptorbindingsruptuur in plaats van de loslating van probekraal van de RBC.
Protocol
Representative Results
Discussion
Samenvattend hebben we een gedetailleerd data-analyseprotocol geleverd voor het voorbewerken van de DFS-ruwe gegevens en het afleiden van moleculaire veerconstanten in de BFP Bead-Bead en Bead-Cell analysemodi. Biomechanische modellen en vergelijkingen die nodig zijn voor het bepalen van moleculaire en cellulaire veerconstanten worden gepresenteerd. Hoewel verschillende integrinen worden bestudeerd, heeft de kmol gemeten door de Bead-Bead-modus en de Bead-Cell-modus significante bereiksverschillen ( F…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We bedanken Guillaume Troadec voor de nuttige discussie, Zihao Wang voor hardwareconsultatie en Sydney Manufacturing Hub, Gregg Suaning en Simon Ringer voor ondersteuning van onze labstart-up. Dit werk werd ondersteund door het Australian Research Council Discovery Project (DP200101970 – L.A.J.), NSW Cardiovascular Capacity Building Program (Early-Mid Career Researcher Grant – L.A.J.), Sydney Research Accelerator prize (SOAR – L.A.J.), Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.), en The University of Sydney Faculty of Engineering Startup Fund and Major Equipment Scheme (L.A.J.). Lining Arnold Ju is een Decra fellow van de Australian Research Council (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

