Molekulare Federkonstantenanalyse mittels Biomembran-Kraftsondenspektroskopie
Summary
Eine Biomembrankraftsonde (BFP) ist eine In-situ-Technik der dynamischen Kraftspektroskopie (DFS). BFP kann verwendet werden, um die Federkonstante molekularer Wechselwirkungen auf lebenden Zellen zu messen. Dieses Protokoll präsentiert eine Federkonstantenanalyse für molekulare Bindungen, die von BFP nachgewiesen wurden.
Abstract
Eine Biomembrankraftsonde (BFP) hat sich kürzlich als Nanowerkzeug für die native Zelloberfläche oder die dynamische Kraftspektroskopie (DFS) mit nativer Zelloberfläche oder in situ entwickelt, das die einzelmolekulare Bindungskinetik messen, mechanische Eigenschaften von Liganden-Rezeptor-Interaktionen bewerten, proteindynamische Konformationsänderungen visualisieren und rezeptorvermittelte Zellmechanosensormechanismen aufregender aufklären kann. In jüngerer Zeit wurde BFP verwendet, um die Federkonstante molekularer Bindungen zu messen. Dieses Protokoll beschreibt das schrittweise Verfahren zur Durchführung der molekularen Federkonstanten-DFS-Analyse. Konkret werden zwei BFP-Betriebsmodi diskutiert, nämlich die Bead-Cell- und Bead-Bead-Modi. Dieses Protokoll konzentriert sich auf die Ableitung von Federkonstanten der molekularen Bindung und Zelle aus DFS-Rohdaten.
Introduction
Als Lebendzell-DFS-Technik entwickelt BFP ein menschliches rotes Blutkörperchen (RBC; Abbildung 1) in einen ultraempfindlichen und abstimmbaren Kraftaufnehmer mit einem kompatiblen Federkonstantenbereich bei 0,1-3 pN/nm1,2,3. Um die Liganden-Rezeptor-Interaktion zu untersuchen, ermöglicht BFP DFS-Messungen bei ~1 pN (10-12 N), ~3 nm (10-9 m) und ~0,5 ms (10-3 s) in Kraft, räumlicher und zeitlicher Auflösung4,5. Seine experimentelle Konfiguration besteht aus zwei gegenüberliegenden Mikropipetten, nämlich der Sonde und dem Ziel. Die Sonden-Mikropipette saugt ein RBC an und eine Perle wird an ihrer Spitze über eine Biotin-Streptavidin-Wechselwirkung geklebt. Die Perle ist mit dem interessierenden Liganden beschichtet (Abbildung 1A). Die Target-Mikropipette aspirat entweder eine Zelle oder eine Perle mit dem interessierenden Rezeptor, entsprechend den Modi Bead-Cell (Abbildung 1B) und Bead-Bead (Abbildung 1C) bzw.5.
BFP-Aufbau, Montage und die DFS-Versuchsprotokolle wurden zuvor ausführlich beschrieben1,6. Kurz gesagt, ein BFP-Touch-Zyklus besteht aus 5 Stufen: Annäherung, Impinge, Kontakt, Rückzug und Dissoziieren (Abbildung 1D). Die horizontale RBC-Spitzenposition wird als ΔxRBC bezeichnet. Zu Beginn ist die unbelastete (kraftfreie) RBC-Verformung ΔxRBC 0 (Tabelle 1). Das Ziel wird dann von einem Piezotranslator angetrieben, um auf die Sondenperle einzufallen und sich von ihr zurückzuziehen (Abbildung 1D). Die RBC-Sonde wird zunächst vom Target mit negativer RBC-Verformung ΔxRBC-< 0 komprimiert. In einem Bond-Ereignis geht die Retract-Stufe von einer Druck- in eine Zugphase mit positiver RBC-Verformung ΔxRBC > 0 über(Abbildung 2C und D). Nach dem Hookeschen Gesetz kann die BFP-Lagerkraft als F = kRBC × ΔxRBCgemessenwerden, wobei kRBC ( Tabelle1) die RBC-Federkonstante des BFP ist. Nach Abschluss eines Berührungszyklus kehrt die Sondenperle mit Δ xRBC = 0 in die Nullkraftposition zurück (Abbildung 1D).
Zur Bestimmung des kRBCmessen und erfassen wir die Radien der Sondenmikropipetten-Innenöffnung (Rp), der RBC (R0) und der kreisförmigen Kontaktfläche (Rc) zwischen dem RBC und der Sondenperle ( Abbildung1A). Dann wird kRBC nach dem Evan’s Modell (Eq. 1)7,8 unter Verwendung eines LabVIEW-Programms berechnet, das als virtuelles Instrument (VI) zum Betrieb des BFP fungiert (Abbildung S1A)8,9.
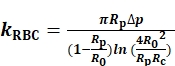 (Gleichung 1)
(Gleichung 1)
Mit einem BFP etabliert und DFS Rohdaten erhalten, präsentieren wir hiermit, wie die Federkonstante von Liganden-Rezeptor-Paaren oder Zellen analysiert werden kann. Die DFS-Rohdaten zur Wechselwirkung des glykosylierten Proteins Thy-1 und K562 zelltragendes Integrin α5β1 (Thy-1-α5β1; Abbildungen 3A und 3B)10 und die des fibrinogen- und perlenbeschichteten Integrins αIIbβ3 (FGN-αIIbβ3; Abbildung 3C) 11,12 wurden verwendet, um die Analysemodi Bead-Cell bzw. Bead-Bead zu demonstrieren.
BFP Versuchsvorbereitung
Einzelheiten zur experimentellen Vorbereitung und Instrumentierung von BFP finden Sie in den zuvor veröffentlichten Protokollen3. Kurz gesagt, humanes RBC wurde mit dem Biotin-PEG3500-NHS im Kohlenstoff-/Bicarbonatpuffer biotinyliert. Interessante Proteine wurden kovalent mit den Borosilikatglasperlen unter Verwendung von MAL-PEG3500-NHS im Phosphatpuffer gekoppelt. Zur Bindung an das biotinylierte RBC wird die Sondenperle auch mit Streptavidin (SA) unter Verwendung des MAL-SA beschichtet. Bitte beachten Sie die Materialtabelle und Tabelle 2.
Um den BFP (Abbildung 1, links) zusammenzubauen, wird die dritte Mikropipette, die als “Helper” bezeichnet wird, verwendet, um die Sondenperle zu liefern und sie an die Spitze1,3des RBC zu kleben. Die kovalente Wechselwirkung zwischen der SA-beschichteten Sondenperle und dem biotinylierten RBC ist viel stärker als die interessierende Ligandenrezeptorbindung. Daher kann das Dissoziierungsstadium als Liganden-Rezeptor-Bindungsbruch und nicht als Ablösung der Sondenperle vom RBC interpretiert werden.
Protocol
Representative Results
Discussion
Zusammenfassend haben wir ein detailliertes Datenanalyseprotokoll zur Vorverarbeitung der DFS-Rohdaten und zur Ableitung molekularer Federkonstanten in den BFP-Analysemodi Bead-Bead und Bead-Cell bereitgestellt. Biomechanische Modelle und Gleichungen, die zur Bestimmung molekularer und zellulärer Federkonstanten erforderlich sind, werden vorgestellt. Obwohl verschiedene Integrine untersucht werden, besitzt das kmol, gemessen durch den Bead-Bead-Modus und den Bead-Cell-Modus signifikante Reichweitenun…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken Guillaume Troadec für die hilfreiche Diskussion, Zihao Wang für die Hardware-Beratung und Sydney Manufacturing Hub, Gregg Suaning und Simon Ringer für die Unterstützung unseres Labor-Startups. Diese Arbeit wurde unterstützt durch das Australian Research Council Discovery Project (DP200101970 – L.A.J.), das NSW Cardiovascular Capacity Building Program (Early-Mid Career Researcher Grant – L.A.J.), den Sydney Research Accelerator Prize (SOAR – L.A.J.), den Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), den National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.) und den University of Sydney Faculty of Engineering Startup Fund and Major Equipment Scheme (L.A.J.). Lining Arnold Ju ist deCRA-Fellow des Australian Research Council (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

