Анализ молекулярной пружинной константы с помощью зондовой спектроскопии биомембранных сил
Summary
Биомембранный силовой зонд (BFP) – это метод динамической силовой спектроскопии in situ (DFS). BFP может быть использован для измерения пружинной постоянной молекулярных взаимодействий на живых клетках. Этот протокол представляет весенний постоянный анализ молекулярных связей, обнаруженных BFP.
Abstract
Биомембранный силовой зонд (BFP) недавно появился как наноинструмент для нативной клеточной поверхности или динамической силовой спектроскопии in situ (DFS), который может измерять кинетику одномолекулярного связывания, оценивать механические свойства взаимодействий лиганд-рецептор, визуализировать динамические конформационные изменения белка и более захватывающе разъяснять механизмы механозонирования клеток, опосредованные рецепторами. Совсем недавно BFP был использован для измерения пружинной постоянной молекулярных связей. Этот протокол описывает пошаговую процедуру выполнения анализа DFS с константой молекулярной пружины. В частности, обсуждаются два режима работы BFP, а именно режимы Bead-Cell и Bead-Bead. Этот протокол фокусируется на получении пружинных констант молекулярной связи и клетки из необработанных данных DFS.
Introduction
В качестве метода DFS с живыми клетками BFP разрабатывает эритроцит человека (RBC; Рисунок 1) в сверхчувствительный и настраиваемый преобразователь силы с совместимым постоянным диапазоном пружин при 0,1-3 пН/нм1,2,3. Для зондирования взаимодействия лиганд-рецептор BFP позволяет проводить измерения DFS при ~1 пН (10-12 Н), ~3 нм (10-9м) и ~0,5 мс (10-3 с) в силе, пространственном и временном разрешении 4,5. Его экспериментальная конфигурация состоит из двух противоположных микропипет, а именно зонда и мишени. Микропипетка Probe аспирирует эрицит, и шарик приклеивается на его вершине посредством взаимодействия биотин-стрептавидин. Шарик покрыт интересующих лигандами(рисунок 1А). Микропипетка-мишень аспирирует либо клетку, либо бусину, несущую интересующих рецептор, соответствующий режимам Бисероплетение(Рисунок 1В)и Бусина-Бусина(Рисунок 1С),соответственно5.
Построение, сборка и экспериментальные протоколы DFS были подробно описаныранее 1,6. Вкратце, сенсорный цикл BFP состоит из 5 этапов: подход, ущемление, контакт, втягивание и диссоциат(рисунок 1D). Горизонтальная вершинная позиция RBC обозначается как ΔxRBC. В начале безударная (с нулевой силой) деформация RBC ΔxRBC равна 0(таблица 1). Затем мишень приводится в движение пьезотранслятором для впрызания и втягивания из шарика зонда(рисунок 1D). Зонд RBC сначала сжимается мишенью с отрицательной деформацией RBC ΔxRBC < 0. В событии Связи стадия втягивания переходит из сжимающей в растягиваемую фазу с положительной деформацией RBC ΔxRBC > 0(рис. 2C и D). Согласно закону Гука, несущая сила BFP может быть измерена как F = kRBC × ΔxRBC, где kRBC (Таблица 1)— константа пружины RBC BFP. При разрыве связи и завершении цикла одного касания шарик зонда возвращается в положение с нулевой силой при ΔxRBC = 0(рисунок 1D).
Чтобы определить kRBC,мы измеряем и регистрим радиусы внутреннего отверстия микропипетки зонда(Rp),RBC(R0)и круговой области контакта(Rc)между RBC и шариком зонда(рисунок 1A). Затем kRBC вычисляется по модели Эвана (Eq. 1) 7,8 с использованием программы LabVIEW, которая действует как виртуальный инструмент (VI) для работы BFP(рисунок S1A)8,9.
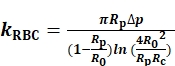 (Экв. 1)
(Экв. 1)
Установив БФП и получив исходные данные DFS, мы представляем, как анализировать пружинную константу пары лиганд-рецептор или клеток. Исходные данные DFS о взаимодействии гликозилированного белка Thy-1 и клетки K562, несущей интегрин, α5β1 (Thy-1-α5β1; Фиг.3А и 3В)10и фибриноген и интегрин с шариковым покрытием αIIbβ3 (FGN-αIIbβ3; Рисунок 3C) 11,12 были использованы для демонстрации режимов анализа Бисероплетняя клетка и Бисероплета, соответственно.
Экспериментальная подготовка BFP
Для получения подробной информации об экспериментальной подготовке и приборах BFP, пожалуйста, обратитесь к ранее опубликованным протоколам3. Короче говоря, человеческий эриртикат был биотинилирован с использованием Biotin-PEG3500-NHS в буфере углерода / бикарбоната. Интересуемые белки были ковалентно соединены с боросиликатными стеклянными шариками с использованием MAL-PEG3500-NHS в фосфатном буфере. Для прикрепления к биотинилированному эрициту шарик зонда также покрывают стрептавидином (SA) с использованием MAL-SA. Пожалуйста, ознакомьтесь с Таблицей материалов и Таблицей 2.
Для сборки BFP(рисунок 1, слева)третья микропипетка, названная «Помощник», будет использоваться для доставки шарика зонда и приклеивания его к вершинеRBC 1,3. Ковалентное взаимодействие между шариком зонда с покрытием SA и биотинилированным RBC намного сильнее, чем интересуящее связь лиганд-рецептор. Таким образом, диссоциатная стадия может быть интерпретирована как разрыв связи лиганд-рецептор, а не отделение шарика зонда от эрицита.
Protocol
Representative Results
Discussion
Таким образом, мы предоставили подробный протокол анализа данных для предварительной обработки необработанных данных DFS и получения констант молекулярной пружины в режимах анализа BFP Bead-Bead и Bead-Cell. Представлены биомеханические модели и уравнения, необходимые для определения молекуля?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы благодарим Гийома Троадека за полезную дискуссию, Zihao Wang за консультации по аппаратному обеспечению и Sydney Manufacturing Hub, Грегга Суанинга и Саймона Рингера за поддержку нашего лабораторного стартапа. Эта работа была поддержана Австралийским исследовательским советом Discovery Project (DP200101970 – L.A.J.), Программой наращивания сердечно-сосудистого потенциала Нового Южного Уэльса (грант исследователя в начале и середине карьеры – L.A.J.), премией Сиднейского исследовательского акселератора (SOAR – L.A.J.), Инвестиционным грантом Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), Грантом идей Национального совета по здравоохранению и медицинским исследованиям (APP2003904 – L.A.J.) и Фондом стартапов факультета инженерных разработок Университета Сиднея и схемой крупного оборудования (L.A.J.). Лайнинг Арнольд Джу является членом Австралийского исследовательского совета DECRA (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

