生体膜力プローブ分光法による分子ばね定数解析
Summary
バイオメンブレン力プローブ(BFP)は、イン ・ス・ ダイナミック・フォース分光法(DFS)の技術です。BFPは、生細胞上の分子相互作用のばね定数を測定するために使用することができる。このプロトコルは、BFPによって検出された分子結合に対するばね定数分析を提示する。
Abstract
生体膜力プローブ(BFP)は、最近、単一分子結合性キネティクスを測定し、リガンド-受容体相互作用の機械的特性を評価し、タンパク質の動的構造変化を視覚化し、よりエキサイティングに受容体媒介細胞メカノセンシング機構を解明することができる、ネイティブ細胞表面または その場 動的力分光(DFS)ナノツールとして出現しました。最近では、BFPは分子結合のばね定数を測定するために使用されています。このプロトコルは、分子ばね定数DFS分析を実行するためのステップバイステップの手順を説明する。具体的には、2つのBFP動作モード、すなわちビードセルとビードビードモードについて説明します。このプロトコルは、DFS生データから分子結合と細胞のばね定数を導き出することに焦点を当てています。
Introduction
ライブセルDFS技術として、BFPはヒト赤血球(RBC;)図1)0.1-3pN/nm1、2、3で互換性のあるばね定数範囲を持つ超高感度で調整可能な力トランスデューサーに。リガンドと受容体の相互作用をプローブするために、BFPは、約1pN(10-12 N)、〜3nm(10-9 m)、および〜0.5 ms(10-3 s)の力、空間、および時間分解能4、5でDFS測定を可能にする。その実験的構成は、2つの対向するマイクロピペット、すなわちプローブとターゲットで構成されています。プローブマイクロピペットはRBCを吸引し、ビーズはビオチンとストレプトアビジンの相互作用を介して頂点に接着されます。ビーズは、目的のリガンドでコーティングされている(図1A)。標的マイクロピペットは、対象の受容体を担う細胞またはビーズのいずれかを吸引し、ビーズ細胞(図1B)およびビーズビーズ(図1C)モードにそれぞれ対応し、それぞれ5。
BFP の構築、アセンブリ、DFS 実験プロトコルについては、前に1,6を詳しく説明しました。簡単に言えば、BFPタッチサイクルは、アプローチ、インピンジ、接触、後退および解化の5つの段階で構成されています(図1D)。水平 RBC 頂点位置は ΔxRBCと示されます。冒頭で、無ストレス(ゼロフォース)RBC変形ΔxRBCは0(表1)である。ターゲットは、ピエゾトランスレータによって駆動され、プローブビードを押さえ、引き込まれるようにします(図1D)。RBCプローブは、まず負のRBC変形ΔxRBC<0でターゲットによって圧縮されます。ボンドイベントでは、リトラクトステージは、正のRBC変形ΔxRBC>0(図2CおよびD)を有する圧縮相から引張り相に移行する。フックの法則によれば、BFP軸受力は、F = kRBC×Δ xRBCとして測定することができ、k RBC(表1)はBFPのRBCばね定数である。ボンド破裂と1回のタッチサイクルの完了時に、プローブビーズはΔxRBC = 0(図1D)でゼロフォース位置に戻ります。
kRBCを決定するために、プローブマイクロピペット内オリフィス(R p)、RBC(R0)、およびRBCとプローブビーズの間の円形接触領域(Rc)の半径を測定し、記録する(図1A)。次いで、KRBCは、BFP(図S1A)8,9を操作する仮想計測器(VI)として機能するLabVIEWプログラムを用いて、Evanのモデル(Eq. 1)7,8に従って計算される。
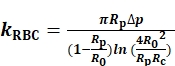 (Eq. 1)
(Eq. 1)
BFPが確立され、DFS生データが得られたことで、リガンドとレセプターの対または細胞のばね定数を分析する方法を紹介します。グリコシル化タンパク質Thy-1とK562細胞を持つインテグリンの相互作用に関するDFS生データα 5 β 1 (thy-1-α5β1;図3A及び3B)10及びフィブリノーゲン及びビーズ αのそのとIIb β 3(FGN-α IIb β 3; 図3C)11,12は、ビーズセルおよびビードビーズ分析モードをそれぞれ実証するために使用されています。
BFP実験準備
BFP実験の準備と計測の詳細については、前に公開したプロトコル3を参照してください。簡単に言えば、ヒトRBCは、炭素/重炭酸塩緩衝液中のビオチン-PEG3500-NHSを用いてビオチン化されている。目的のタンパク質は、リン酸緩衝液中のMAL-PEG3500-NHSを用いてホウケイ酸ガラスビーズに共有結合されている。ビオチン化RBCに付着するために、プローブビーズはまた、MAL-SAを使用してストレプトアビジン(SA)でコーティングされています。 資料表 と 表2を参照してください。
BFP(図1、左)を組み立てるには、プローブビーズを送達し、RBCの頂点1、3に接着するために「ヘルパー」と呼ぶ3番目のマイクロピペットを使用します。SAコーティングプローブビーズとビオチン化RBCの共有結合相互作用は、対象のリガンド-受容体結合よりもはるかに強い。したがって、解離ステージは、RBCからのプローブビーズの剥離ではなく、リガンド-受容体結合破裂と解釈することができる。
Protocol
Representative Results
Discussion
要約すると、BFPビーズビーズおよびビードセル解析モードでDFS生データを前処理し、分子ばね定数を導出するための詳細なデータ分析プロトコルを提供しました。分子および細胞のばね定数を決定するために必要な生体力学的モデルと方程式が提示される。異なるインテグリンが研究されているにもかかわらず、ビーズ・ビーズ・モードとビーズ・セル・モードで測定された kモル…
Disclosures
The authors have nothing to disclose.
Acknowledgements
ギヨーム・トロアデックは、有益な議論、ハードウェアコンサルテーションのためのZihao Wang、シドニー・マニュファクチャリング・ハブ、グレッグ・スアンイン、サイモン・リンガーのラボスタートアップのサポートに感謝します。この研究は、オーストラリア研究評議会ディスカバリープロジェクト(DP200101970 – L.A.J.)、NSW心血管能力構築プログラム(早期キャリア研究者グラント – L.A.J.)、シドニー研究アクセラレータ賞(SOAR – L.A.J.)、ラマシオッティ財団によって支援されました 健康投資助成金(2020HIG76 – L.A.J.)、国民保健医療研究評議会アイデアグラント(APP2003904 – L.A.J.)、シドニー大学工学部スタートアップファンドと主要機器スキーム(L.A.J.)。ライニングアーノルドジュは、オーストラリア研究評議会DECRAフェロー(DE190100609)です。
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

