Analisi della costante molecolare della molla mediante spettroscopia a sonda a forza biomembrana
Summary
Una sonda di forza biomembrana (BFP) è una tecnica di spettroscopia di forza dinamica in situ (DFS). La BFP può essere utilizzata per misurare la costante di molla delle interazioni molecolari sulle cellule viventi. Questo protocollo presenta l’analisi costante a molla per i legami molecolari rilevati da BFP.
Abstract
Una sonda di forza biomembrana (BFP) è recentemente emersa come nanostrutto per spettroscopia di forza dinamica (DFS) a superficie cellulare nativa o in situ in grado di misurare la cinetica di legame molecolare singolo, valutare le proprietà meccaniche delle interazioni ligando-recettore, visualizzare i cambiamenti conformazionali dinamici della proteina e chiarire in modo più eccitante i meccanismi di meccanosensing cellulare mediati dal recettore. Più recentemente, la BFP è stata utilizzata per misurare la costante di molla dei legami molecolari. Questo protocollo descrive la procedura passo-passo per eseguire l’analisi DFS a molla molecolare costante. In particolare, vengono discusse due modalità operative BFP, vale a dire le modalità Bead-Cell e Bead-Bead. Questo protocollo si concentra sulla derivazione di costanti molle del legame molecolare e della cellula dai dati grezzi DFS.
Introduction
Come tecnica DFS a cellule vive, BFP progetta un globuli rossi umani (RBC; Figura 1) in un trasduttore di forza ultrasensibile e sintonizzabile con un intervallo costante di molla compatibile a 0,1-3 pN/nm1,2,3. Per sondare l’interazione ligando-recettore, BFP consente misurazioni DFS a ~ 1 pN(10 -12 N), ~ 3 nm(10 -9 m) e ~ 0,5 ms(10 -3 s) in forza, risoluzione spaziale e temporale4,5. La sua configurazione sperimentale è costituita da due micropipette opposte, ovvero la sonda e il bersaglio. La micropipetta Probe aspira un RBC e una perle viene incollata al suo apice tramite un’interazione biotina-streptavitina. Il perno è rivestito con il ligando di interesse (Figura 1A). La micropipetta target aspira una cellula o una perna che porta il recettore di interesse, corrispondente rispettivamente alle modalità Bead-Cell (Figura 1B) e Bead-Bead (Figura 1C), rispettivamente5.
La costruzione, l’assemblaggio e i protocolli sperimentali DFS BFP sono stati descritti in dettaglio in precedenza1,6. In breve, un ciclo tattile BFP è costituito da 5 fasi: Approccio, Impinge, Contatto, Retract e Dissociate (Figura 1D). La posizione dell’apice RBC orizzontale è indicata come ΔxRBC. All’inizio, la deformazione RBC non accentata (forza zero) ΔxRBC è 0 (Tabella 1). Il bersaglio viene quindi guidato da un piezotraduatore per impattarsi e ritrarsi dal perla della sonda (Figura 1D). La sonda RBC viene prima compressa dal Target con deformazione RBC negativa ΔxRBC < 0. In un evento di legame, lo stadio di retrazione passa da una fase di compressione a una fase di trazione con deformazione RBC positiva ΔxRBC > 0 (Figura 2C e D). Secondo la legge di Hooke, la forza portante BFP può essere misurata come F = kRBC × ΔxRBC, dove kRBC ( Tabella1) è la costante della molla RBC del BFP. Dopo la rottura del legame e il completamento di un ciclo di tocco, il perle della sonda ritorna in posizione di forza zero con ΔxRBC = 0 (Figura 1D).
Per determinare il kRBC,misuriamo e registriamo i raggi dell’orifizio interno della micropipetta della sonda (Rp), l’RBC (R0) e l’area di contatto circolare (Rc) tra l’RBC e il perlo della sonda ( Figura1A). Quindi kRBC viene calcolato secondo il modello di Evan (Eq. 1)7,8 utilizzando un programma LabVIEW che funge da strumento virtuale (VI) per far funzionare il BFP(Figura S1A)8,9.
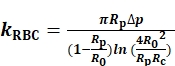 (Eq. 1)
(Eq. 1)
Con un BFP stabilito e i dati grezzi DFS ottenuti, presentiamo come analizzare la costante di molla della coppia o delle cellule ligando-recettore. I dati grezzi DFS sull’interazione della proteina glicosilata Thy-1 e della cellula K562 portante integrina α5β1 (Thy-1-α5β1; Figure 3A e 3B)10 e quella dell’integrina rivestita di fibrinogeno e perle αIIbβ3 (FGN-αIIbβ3; Figura 3C) 11,12 sono stati utilizzati per dimostrare le modalità di analisi Bead-Cell e Bead-Bead, rispettivamente.
Preparazione sperimentale BFP
Per i dettagli sulla preparazione sperimentale e la strumentazione BFP, fare riferimento ai protocolli3precedentemente pubblicati. In breve, l’RBC umano è stato biotinilato utilizzando la Biotina-PEG3500-NHS nel tampone di carbonio / bicarbonato. Le proteine di interesse sono state accoppiate covalentemente alle perle di vetro borosilicato utilizzando MAL-PEG3500-NHS nel tampone fosfato. Per attaccarsi al RBC biotinilato, il perla della sonda è anche rivestito con streptavitina (SA) utilizzando il MAL-SA. Si prega di consultare la Tabella dei materiali e la Tabella 2.
Per assemblare il BFP (Figura 1, a sinistra), la terza micropipetta denominata ‘Helper’ verrà utilizzata per consegnare la sonda e incollarla all’apice del RBC1,3. L’interazione covalente tra il perle della sonda rivestito sa e l’RBC biotinilato è molto più forte del legame ligando-recettore di interesse. Pertanto, lo stadio di dissociato può essere interpretato come la rottura del legame ligando-recettore piuttosto che il distacco del perlo della sonda dal RBC.
Protocol
Representative Results
Discussion
In sintesi, abbiamo fornito un protocollo di analisi dei dati dettagliato per la pre-elaborazione dei dati grezzi DFS e la derivazione di costanti molecolari a molla nelle modalità di analisi BFP Bead-Bead e Bead-Cell. Vengono presentati i modelli biomeccanici e le equazioni necessarie per determinare le costanti molecolari e cellulari della molla. Sebbene siano studiate integrine diverse, la kmol misurata dal modo Bead-Bead e il modo Bead-Cell possiedono differenze di gamma significative ( Figura<st…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo Guillaume Troadec per l’utile discussione, Zihao Wang per la consulenza hardware e Sydney Manufacturing Hub, Gregg Suaning e Simon Ringer per il supporto della nostra startup di laboratorio. Questo lavoro è stato supportato dall’Australian Research Council Discovery Project (DP200101970 – L.A.J.), nsw Cardiovascular Capacity Building Program (Early-Mid Career Researcher Grant – L.A.J.), Sydney Research Accelerator prize (SOAR – L.A.J.), Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.) e The University of Sydney Faculty of Engineering Startup Fund and Major Equipment Scheme (L.A.J.). Lining Arnold Ju è un borsista DECRA dell’Australian Research Council (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

