Analyse de la constante de ressort moléculaire par spectroscopie de sonde de force de biomembrane
Summary
Une sonde de force biombrane (BFP) est une technique de spectroscopie de force dynamique (DFS) in situ. BFP peut être utilisé pour mesurer la constante de ressort des interactions moléculaires sur les cellules vivantes. Ce protocole présente une analyse de la constante de ressort pour les liaisons moléculaires détectées par BFP.
Abstract
Une sonde de force biomébrarane (BFP) a récemment émergé en tant que nanooutil de spectroscopie de force dynamique (DFS) à surface cellulaire native ou in situ qui peut mesurer la cinétique de liaison moléculaire unique, évaluer les propriétés mécaniques des interactions ligand-récepteur, visualiser les changements conformationnels dynamiques des protéines et élucider de manière plus excitante les mécanismes de mécanosension cellulaire médiés par les récepteurs. Plus récemment, le BFP a été utilisé pour mesurer la constante de ressort des liaisons moléculaires. Ce protocole décrit la procédure étape par étape pour effectuer une analyse DFS de la constante de ressort moléculaire. Plus précisément, deux modes de fonctionnement BFP sont discutés, à savoir les modes Bead-Cell et Bead-Bead. Ce protocole se concentre sur la dérivation des constantes de ressort de la liaison moléculaire et de la cellule à partir de données brutes DFS.
Introduction
En tant que technique DFS à cellules vivantes, BFP conçoit un globule rouge humain (RBC; Figure 1) dans un transducteur de force ultrasensible et accordable avec une plage constante de ressort compatible à 0,1-3 pN/nm1,2,3. Pour sonder l’interaction ligand-récepteur, BFP permet des mesures DFS à ~1 pN(10 -12 N), ~3 nm(10 -9 m) et ~0,5 ms (10-3 s) en force, résolution spatiale et temporelle4,5. Sa configuration expérimentale se compose de deux micropipettes opposées, à savoir la sonde et la cible. La micropipette Probe aspire un globules biologiques et une perle est collée à son sommet via une interaction biotine-streptavidine. La perle est recouverte du ligand d’intérêt (Figure 1A). La micropipette cible aspire soit une cellule, soit une perle portant le récepteur d’intérêt, correspondant aux modes Bead-Cell(Figure 1B)et Bead-Bead(Figure 1C),respectivement5.
La construction BFP, l’assemblage et les protocoles expérimentaux DFS ont été décrits en détail précédemment1,6. En bref, un cycle tactile BFP se compose de 5 étapes : Approcher, Impinge, Contact, Retract et Dissocier(Figure 1D). La position horizontale de l’apex RBC est notée ΔxRBC. Au début, la déformation RBC non étayée (force nulle) ΔxRBC est 0(tableau 1). La cible est ensuite entraînée par un piézotranslateur pour empiéter et se rétracter sur le cordon de la sonde(Figure 1D). La sonde RBC est d’abord comprimée par la cible avec une déformation RBC négative ΔxRBC < 0. Dans un cas de liaison, l’étage de rétraction passe d’une phase de compression à une phase de traction avec une déformation RBC positive ΔxRBC > 0(Figure 2C et D). Selon la loi de Hooke, la force de roulement BFP peut être mesurée comme F = kRBC × ΔxRBC, où kRBC ( Tableau1) est la constante de ressort RBC du BFP. À la rupture de la liaison et à la fin d’un cycle tactile, le bille de sonde revient à la position de force nulle avec ΔxRBC = 0(Figure 1D).
Pour déterminer le kRBC,nous mesurons et enregistrons les rayons de l’orifice interne de la micropipette de sonde (Rp), du RBC (R0) et de la zone de contact circulaire (Rc) entre le RBC et le bille de sonde ( Figure1A). Ensuite, kRBC est calculé selon le modèle d’Evan (Eq. 1)7,8 à l’aide d’un programme LabVIEW qui agit comme un instrument virtuel (VI) pour faire fonctionner le BFP(Figure S1A)8,9.
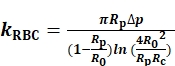 (Eq. 1)
(Eq. 1)
Avec un BFP établi et des données brutes DFS obtenues, nous présentons ici comment analyser la constante de ressort de la paire ligand-récepteur ou des cellules. Les données brutes DFS sur l’interaction de la protéine glycosylée Thy-1 et de la cellule K562 portant l’intégrine α5β1 (Thy-1-α5β1; Figures 3A et 3B)10 et celle de l’intégrine enduite de fibrinogène et de billes αIIbβ3 (FGN-αIIbβ3; Figure 3C) 11,12 ont été utilisés pour démontrer les modes d’analyse Bead-Cell et Bead-Bead, respectivement.
Préparation expérimentale BFP
Pour plus de détails sur la préparation expérimentale et l’instrumentation du BFP, veuillez vous référer aux protocoles précédemment publiés3. En bref, les globules biologiques humains ont été biotinylés à l’aide du Biotin-PEG3500-NHS dans le tampon carbone/bicarbonate. Les protéines d’intérêt ont été couplées de manière covalente aux billes de verre borosilicate en utilisant MAL-PEG3500-NHS dans le tampon de phosphate. Pour se fixer au globule rouge biotinylé, la bille de sonde est également recouverte de streptavidine (SA) à l’aide du MAL-SA. Veuillez consulter le tableau des matériaux et le tableau 2.
Pour assembler le BFP(Figure 1, à gauche),la troisième micropipette appelée « Helper » sera utilisée pour délivrer la bille de sonde et la coller à l’apex du RBC1,3. L’interaction covalente entre la bille de sonde revêtue de SA et les globules rouges biotinylés est beaucoup plus forte que la liaison ligand-récepteur d’intérêt. Ainsi, l’étape dissociée peut être interprétée comme la rupture de la liaison ligand-récepteur plutôt que le détachement de la perle de sonde du globule rouge.
Protocol
Representative Results
Discussion
En résumé, nous avons fourni un protocole d’analyse de données détaillé pour le prétraitement des données brutes DFS et la dérivation des constantes de ressort moléculaire dans les modes d’analyse BFP Bead-Bead et Bead-Cell. Des modèles biomécaniques et des équations nécessaires à la détermination des constantes de ressort moléculaires et cellulaires sont présentés. Bien que différentes intégrines soient étudiées, le kmol mesuré par le mode Perle-Perle et le mode Perle-Cellu…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions Guillaume Troadec pour la discussion utile, Zihao Wang pour la consultation sur le matériel, et Sydney Manufacturing Hub, Gregg Suaning et Simon Ringer pour le soutien de notre démarrage de laboratoire. Ce travail a été soutenu par l’Australian Research Council Discovery Project (DP200101970 – L.A.J.), le NSW Cardiovascular Capacity Building Program (Early-Mid Career Researcher Grant – L.A.J.), le Sydney Research Accelerator prize (SOAR – L.A.J.), ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), la National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.) et le Fonds de démarrage et le programme d’équipement majeur de la Faculté d’ingénierie de l’Université de Sydney (L.A.J.). Lining Arnold Ju est un boursier DECRA de l’Australian Research Council (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

