Molekylær vårkonstantanalyse av Biomembrane Force Probe Spectroscopy
Summary
En biomembrane force probe (BFP) er en in situ dynamic force spektroskopi (DFS) teknikk. BFP kan brukes til å måle vårkonstanten av molekylære interaksjoner på levende celler. Denne protokollen presenterer vårkonstantanalyse for molekylære bindinger oppdaget av BFP.
Abstract
En biomembrane force probe (BFP) har nylig dukket opp som en innfødt-celle-overflate eller in situ dynamisk kraft spektroskopi (DFS) nanoverktøy som kan måle enkeltmolekylære binding kinetikk, vurdere mekaniske egenskaper av ligand-reseptor interaksjoner, visualisere protein dynamiske konformasjonsendringer og mer spennende belyse reseptormediert celle mechanosensing mekanismer. Mer nylig har BFP blitt brukt til å måle vårkonstanten av molekylære bindinger. Denne protokollen beskriver den trinnvise prosedyren for å utføre molekylær vårkonstant DFS-analyse. Nærmere bestemt diskuteres to BFP-operasjonsmoduser, nemlig Bead-Cell- og Bead-Bead-modusene. Denne protokollen fokuserer på å utlede vårkonstanter av molekylærbindingen og cellen fra DFS-rådata.
Introduction
Som en live-celle DFS-teknikk utvikler BFP en menneskelig rød blodlegeme (RBC; Figur 1) inn i en ultrasensitiv og justerbar krafttransduser med et kompatibelt fjærkonstantområde på 0,1-3 pN/nm1,2,3. For å undersøke ligandreseptorinteraksjon aktiverer BFP DFS-målinger ved ~1 pN (10-12 N), ~3 nm (10-9 m) og ~0,5 ms (10-3 s) i kraft, romlig og tidsmessig oppløsning4,5. Den eksperimentelle konfigurasjonen består av to motsatte mikropipetter, nemlig sonden og målet. Probemikropipetten aspirerer en RBC og en perle limes på toppen via en biotin-streptavidin interaksjon. Perlen er belagt med ligaen av interesse (Figur 1A). Målmikropipetten aspirerer enten en celle eller en perle som bærer reseptoren av interesse, tilsvarende bead-celle (figur 1B) og perle(figur 1C) modi, henholdsvis5.
BFP-konstruksjon, montering og DFS eksperimentelle protokoller ble beskrevet i detalj tidligere1,6. Kort sagt består en BFP-berøringssyklus av 5 trinn: Approach, Impinge, Contact, Retract and Dissociate (Figur 1D). Den horisontale RBC apex-posisjonen er betegnet som ΔxRBC. I begynnelsen er den ubenyttede (nullkrafts) RBC-deformasjonen ΔxRBC 0 (Tabell 1). Målet drives deretter av en piezotranslator for å hindre og trekke seg tilbake fra sondeperlen (figur 1D). RBC-sonden komprimeres først av Target med negativ RBC-deformasjon ΔxRBC < 0. I en Bond-hendelse går tilbaketrekningsfasen fra et kompressiv til en strekkfase med positiv RBC-deformasjon ΔxRBC > 0 (Figur 2C og D). I henhold til Hookes lov kan BFP-lagerkraften måles som F = kRBC × ΔxRBC, der kRBC ( tabell1) er RBC-fjærkonstanten til BFP. Ved bindingsbrudd og fullføring av en berøringssyklus går sondeperlen tilbake til nullkraftposisjon med ΔxRBC = 0 (Figur 1D).
For å bestemme kRBCmåler og registrerer vi radiene til sondens mikropipette indre åpning (Rp), RBC (R0) og det sirkulære kontaktområdet (Rc) mellom RBC og sondeperlen ( figur1A). Deretter beregnes kRBC i henhold til Evans modell (Eq. 1)7,8 ved hjelp av et LabVIEW-program som fungerer som et virtuelt instrument (VI) for å betjene BFP ( FigurS1A)8,9.
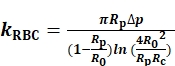 (Eq. 1)
(Eq. 1)
Med en BFP etablert og DFS rådata oppnådd, presenterer vi herved hvordan vi analyserer vårkonstanten til ligandreseptorpar eller celler. DFS-rådataene om samspillet mellom det glykosylerte proteinet Thy-1 og K562 cellebærende integrin α5β1 (Thy-1-α5β1; Figur 3A og 3B)10 og fibrinogen og perle belagt integrin αIIbβ3 (FGN-αIIbβ3; Figur 3C) 11,12 har blitt brukt til å demonstrere bead-cell og perle analyse moduser, henholdsvis.
BFP eksperimentell forberedelse
Hvis du vil ha mer informasjon om BFP eksperimentell forberedelse og instrumentering, kan du se de tidligere publiserte protokollene3. Kort fortalt har human RBC blitt biotinylert ved hjelp av Biotin-PEG3500-NHS i karbon/bikarbonatbufferen. Proteiner av interesse har blitt kovalent koblet til borosilikatglassperler ved hjelp av MAL-PEG3500-NHS i fosfatbufferen. For å feste til den biotinylerte RBC, er sondeperlen også belagt med streptavidin (SA) ved hjelp av MAL-SA. Se Materialtabellen og tabell 2.
For å montere BFP (Figur 1, venstre), vil den tredje mikropipetten kalt ‘Hjelper’ bli brukt til å levere sondeperlen og lime den til RBCs apex1,3. Den kovalente interaksjonen mellom SA-belagt sondeperle og biotinylert RBC er mye sterkere enn den ligandreseptorbindingen av interesse. Dermed kan Dissociate-scenen tolkes som ligandreseptorbindingsbruddet i stedet for løsrivelse av sondeperle fra RBC.
Protocol
Representative Results
Discussion
Oppsummert har vi gitt en detaljert dataanalyseprotokoll for forhåndsbehandling av DFS-rådata og avlede molekylære vårkonstanter i analysemodusene BFP Bead-Bead og Bead-Cell. Biomekaniske modeller og ligninger som kreves for å bestemme molekylære og cellulære vårkonstanter presenteres. Selv om forskjellige integriner studeres, har kmol målt ved Bead-Bead-modus og Bead-Cell-modus betydelige områdeforskjeller ( Figur3A vs. Figur 3C). …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi takker Guillaume Troadec for nyttig diskusjon, Zihao Wang for maskinvarekonsultasjon, og Sydney Manufacturing Hub, Gregg Suaning og Simon Ringer for støtte til vår lab-oppstart. Dette arbeidet ble støttet av Australian Research Council Discovery Project (DP200101970 – L.A.J.), NSW Cardiovascular Capacity Building Program (Early-Mid Career Researcher Grant – L.A.J.), Sydney Research Accelerator Prize (SOAR – L.A.J.), Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.), og University of Sydney Faculty of Engineering Startup Fund og Major Equipment Scheme (L.A.J.). Lining Arnold Ju er en australsk forskningsråd DECRA fellow (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
References
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

