Proteomica della spettrometria di massa guidata da livrea cellulare nell'embrione in via di sviluppo (rana)
Summary
Qui descriviamo una caratterizzazione proteomica basata sulla spettrometria di massa di linee cellulari con destini tissutali noti nell’embrione vertebrato di Xenopus laevis .
Abstract
La caratterizzazione degli eventi molecolari quando le cellule danno origine a tessuti e organi aumenta il potenziale per comprendere meglio lo sviluppo normale e progettare rimedi efficaci per le malattie. Le tecnologie che consentono l’identificazione e la quantificazione accurate di diversi tipi e di un gran numero di proteine fornirebbero informazioni ancora mancanti sui meccanismi molecolari che orchestrano lo sviluppo di tessuti e organismi nello spazio e nel tempo. Qui, presentiamo un protocollo basato sulla spettrometria di massa che consente la misurazione di migliaia di proteine in linee cellulari identificate in embrioni di Xenopus laevis (rana). L’approccio si basa su mappe riproducibili del destino cellulare e metodi consolidati per identificare, etichettare, tracciare e campionare in modo fluorescente le cellule e la loro progenie (cloni) da questo modello di sviluppo dei vertebrati. Dopo aver raccolto il contenuto cellulare utilizzando il microcampionamento o isolando le cellule mediante dissezione o selezione cellulare attivata dalla fluorescenza, le proteine vengono estratte e processate per l’analisi proteomica bottom-up. La cromatografia liquida e l’elettroforesi capillare vengono utilizzate per fornire una separazione scalabile per il rilevamento e la quantificazione delle proteine con spettrometria di massa ad alta risoluzione (HRMS). Vengono forniti esempi rappresentativi per la caratterizzazione proteomica delle cellule adipose del tessuto neurale. La proteomica HRMS guidata dalla linea cellulare è adattabile a diversi tessuti e organismi. È sufficientemente sensibile, specifico e quantitativo per scrutare le dinamiche spazio-temporali del proteoma durante lo sviluppo dei vertebrati.
Introduction
La nostra comprensione della differenziazione cellulare e della genesi di tessuti e organi è il risultato di decenni di elaborati screening mirati di geni e dei loro prodotti. Aumentare la nostra conoscenza di tutte le biomolecole e delle loro quantità durante importanti eventi cellulari aiuterebbe a svelare i meccanismi molecolari che controllano il modello spaziale e temporale del piano corporeo dei vertebrati. Le tecnologie che consentono l’amplificazione molecolare e il sequenziamento sono ora in grado di riferire regolarmente su un gran numero di geni e trascrizioni, supportando studi basati su ipotesi nella ricerca biologica e traslazionale di base. Per comprendere i sistemi in via di sviluppo, una complessa relazione tra trascrizione e traduzione sostiene l’analisi diretta di più proteine e le loro modificazioni post-traduzionali. La proteomica globale che utilizza sistemi biologici in vitro, come le cellule staminali pluripotenti indotte, ha iniziato a delineare i meccanismi di induzione tissutale 1,2. Negli organismi complessi, come l’embrione vertebrato, lo sviluppo si basa su gradienti morfogeni nel contesto dello spazio e del tempo3. Ne consegue che acquisire conoscenze sui cambiamenti proteomici man mano che le cellule si differenziano per formare tessuti specializzati, come i tessuti neurali, offre una chiave per sbloccare programmi molecolari che controllano lo sviluppo normale e difettoso e guidare le terapie di prossima generazione.
La rana artigliata sudafricana vertebrata (Xenopus laevis) è un modello consolidato nella biologia cellulare e dello sviluppo, neuro e rigenerativa. Il Premio Nobel 2012 per la Fisiologia ola Medicina 4,5 di Sir John Gurdon per la scoperta della pluripotenza del nucleo somatico ha evidenziato l’importanza di questo modello per le scoperte negli studi di base e traslazionali. Gli embrioni di Xenopus si sviluppano esternamente alla madre, facilitando così la manipolazione diretta delle cellule, dei cloni cellulari e dell’espressione genica nelle varie fasi di sviluppo. La pigmentazione asimmetrica e le divisioni cellulari stereotipate hanno permesso di tracciare mappe del destino riproducibili dall’embrione a 7,8 e 16-6e 32 cellule. Per la proteomica basata sulla spettrometria di massa ad alta risoluzione (HRMS), ulteriori vantaggi del modello includono dimensioni relativamente grandi (~ 1 mm di diametro), che producono un abbondante contenuto proteico per l’analisi (~ 130 μg negli embrioni in fase di scissione precoce, ~ 10 μg di contenuto proteico in singole cellule dell’embrione a 16 cellule)9,10.
Attualmente, HRMS è la tecnologia leader di scelta per la rilevazione delle proteine. Questa tecnologia consente il rilevamento e la quantificazione diretti, sensibili e specifici di proteine multiple, di solito da centinaia a migliaia di proteine diverse11. La proteomica bottom-up di HRMS comporta una serie di passaggi interconnessi. Dopo l’estrazione dal campione di cellula/tessuto, le proteine vengono digerite con un enzima proteolitico, come la tripsina (proteomica bottom-up). I peptidi risultanti vengono separati in base alle loro diverse proprietà fisico-chimiche, tra cui idrofobicità (cromatografia liquida a fase inversa, LC), carica netta (cromatografia a scambio ionico), dimensioni (cromatografia di esclusione dimensionale) o mobilità elettroforetica (elettroforesi capillare, CE). I peptidi vengono quindi caricati (ionizzati), tipicamente utilizzando la ionizzazione elettrospray (ESI), e gli ioni peptide vengono rilevati e sequenziati tramite frammentazione in fase gassosa mediante HRMS tandem. I dati peptidici risultanti sono mappati sul proteoma dell’organismo studiato. Con l’intensità del segnale dello ione peptidico specifica per proteine (proteotipica) correlata alla concentrazione, la quantificazione delle proteine può essere eseguita senza etichetta o basata sull’etichetta (quantificazione multiplexing). La proteomica HRMS fornisce una ricca risorsa di informazioni sullo stato molecolare del sistema in studio, consentendo la generazione di ipotesi e studi funzionali di follow-up.
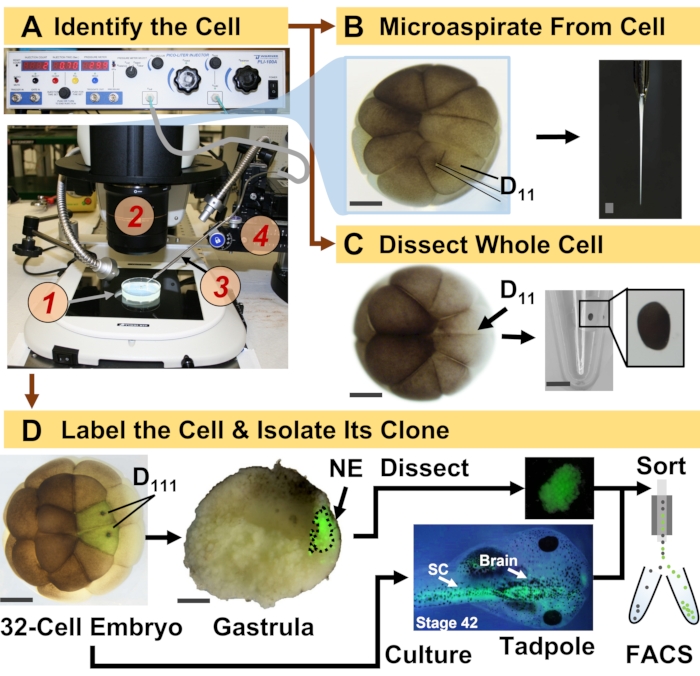
Figura 1: Proteomica spaziotemporalmente scalabile che consente la proteomica HRMS guidata dalla linea cellulare nell’embrione in via di sviluppo (rana). (A) Visualizzazione del campione (1) utilizzando uno stereomicroscopio (2) per l’iniezione di una cellula identificata (inserto), utilizzando una micropipetta fabbricata (3) sotto controllo da uno stadio di traslazione (4). (B) Campionamento subcellulare della cellula D11 sinistra identificata in un embrione a 16 cellule. (C) Dissezione di un’intera cellula D11 da un embrione a 16 cellule. (D) Tracciamento fluorescente (verde) delle progenie D111 sinistra e destra da un embrione a 32 cellule per guidare la dissezione dell’ectoderma neurale (NE) nella gastrula (stadio 10) e l’isolamento del tessuto discendente dal girino mediante FACS. Barre della scala: 200 μm per gli embrioni, 1,25 mm per la fiala. Le figure sono state adattate con il permesso dei riferimenti 15,19,21,59. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Il protocollo qui presentato consente la quantificazione basata su HRMS di un gran numero di proteine in cellule/tessuti identificati nello sviluppo di embrioni di X. laevis. L’approccio si basa su un’accurata identificazione cellulare, mappe del destino cellulare riproducibili e metodologie consolidate per tracciare le linee cellulari in questo modello biologico 6,7,8. Come mostrato nella Figura 1, studiamo i proteomi da singole cellule impiegando la dissezione dell’intera cellula o il microcampionamento capillare per aspirare il contenuto cellulare. Il monitoraggio del lignaggio di una cellula ci permette di studiare l’evoluzione spaziotemporale del proteoma quando le cellule formano i tessuti durante la gastrulazione. La progenie cellulare è marcata in modo fluorescente iniettando un fluoroforo coniugato a destrano inerte o mRNA per proteine fluorescenti (ad esempio, proteina fluorescente verde o GFP). La progenie marcata è isolata nei punti temporali di sviluppo desiderati. Durante la gastrulazione, i cloni cellulari che sono strettamente raggruppati possono essere isolati per dissezione. Dopo la gastrulazione, i cloni cellulari possono essere distribuiti all’interno dell’embrione a causa di movimenti migratori e possono essere isolati dai tessuti dissociati mediante selezione cellulare attivata dalla fluorescenza (FACS). Le proteine in queste cellule e tessuti sono misurate tramite proteomica bottom-up che impiega HPLC o CE per la separazione e HRMS tandem ESI per l’identificazione. La proteomica HRMS guidata dalla linea cellulare è scalabile a diverse dimensioni cellulari e linee cellulari all’interno dell’embrione ed è specifica, sensibile e quantitativa. Attraverso esempi selezionati mostrati qui, dimostriamo anche che questo protocollo è scalabile e ampiamente adattabile a diversi tipi di cellule e linee cellulari.
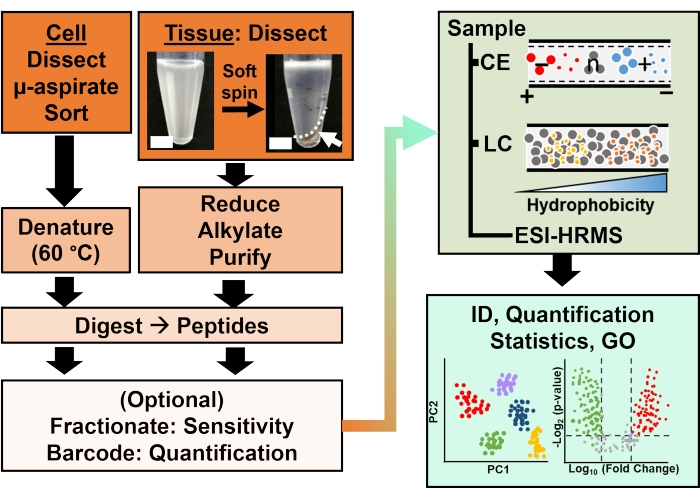
Figura 2: Il flusso di lavoro bioanalitico. Micro-dissezione e aspirazione capillare, o FACS ha facilitato il campionamento del contenuto proteico cellulare e clonale. Deplezione delle abbondanti proteine del tuorlo e separazione mediante elettroforesi capillare (CE) o cromatografia liquida a nanoflusso (LC) con sensibilità di identificazione migliorata (ID) mediante spettrometria di massa ad alta risoluzione (HRMS) a ionizzazione elettrospray (ESI). La quantificazione ha rivelato la disregolazione, fornendo nuove informazioni per studi basati su ipotesi in combinazione con le informazioni disponibili dall’ontologia genica (GO). Le cifre sono state adattate con il permesso del riferimento15. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocol
Representative Results
Discussion
Questo protocollo consente la caratterizzazione dell’espressione proteica in linee cellulari identificate in embrioni della specie Xenopus . Derivando dalla HRMS, la metodologia combina una squisita specificità nell’identificazione molecolare, capacità di rilevamento multi-proteina senza sonde molecolari (di solito centinaia o migliaia di proteine diverse) e una capacità di quantificazione. L’adattabilità agli strumenti e ai flussi di lavoro classici nella (neuro)biologia cellulare e dello sviluppo espande l…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Siamo grati a Jie Li (University of Maryland, College Park) per le preziose discussioni sulla dissociazione embrionale e sulla FACS. Ringraziamo Vi M. Quach e Camille Lombard-Banek per l’assistenza nella preparazione dei campioni e nella raccolta dei dati in studi precedenti che esemplificano le applicazioni proteomiche evidenziate in questo protocollo. Parti di questo lavoro sono state sostenute dalla National Science Foundation con il numero di premio IOS-1832968 CAREER (a P.N.), dal National Institutes of Health con il numero di premio R35GM124755 (a P.N.), dall’Università del Maryland-National Cancer Institute Partnership Program (a P.N.) e dai premi di ricerca della COSMOS Club Foundation (ad A.B.B. e L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. 发育生物学. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. 发育生物学. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. 发育生物学. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

