Direkte måling af KDM1A Target engagement ved hjælp af Chemoprobe-baserede Immunoassays
Summary
Her præsenterer vi en protokol til måling KDM1A Target engagement i en menneske-eller dyrecelle, væv eller blodprøver behandlet med KDM1A-hæmmere. Protokollen anvender chemoprobe-mærkning af det frie KDM1A enzym og direkte kvantificering af målbesættelsen ved hjælp af chemoprobe-baserede immunassays og kan anvendes i prækliniske og kliniske studier.
Abstract
Vurderingen af Target engagement, defineret som samspillet mellem et lægemiddel med det protein, det var designet til, er et grundlæggende krav for fortolkningen af den biologiske aktivitet af enhver forbindelse i narkotika udvikling eller i grundlæggende forskningsprojekter. I Epigenetik, Target engagement er oftest vurderes af analysen af proxy markører i stedet for at måle Unionen af forbindelsen til målet. Downstream biologiske udlæsninger, der er blevet analyseret omfatter Histon Mark modulation eller genekspression ændringer. KDM1A er en lysin demethylase, der fjerner methylgrupper fra mono-og dimethylerede H3K4, en ændring forbundet med hæmning af genekspression. Graduering af proxy markører er afhængig af celletype og funktion af den genetiske sammenstilling af de undersøgte celler, hvilket kan gøre fortolkning og Cross-case sammenligning ganske vanskeligt. For at omgå disse problemer, en alsidig protokol præsenteres for at vurdere dosis effekter og dynamikken i den direkte KDM1A Target engagement. Den beskrevne analyse gør brug af en KDM1A chemoprobe til at indfange og kvantificere uhæmmet enzym, kan anvendes bredt på celler eller vævsprøver uden behov for genetisk modifikation, har et fremragende vindue til påvisning, og kan bruges både til grundforskning og analyse af kliniske prøver.
Introduction
Lysin specifik demethylase 1 (KDM1A)1 er en demethylase involveret i kontrollen af gen transskription. Dette protein er dukket op som en kandidat farmakologiske mål2 i onkologi; herunder akut myeloid Leukemia3 (AML), Myelodysplasi syndrom (MDS)4, myelofibrose (MF)5,6, småcellet lungekræft (SCLC)7; i seglcellesygdom (SCD)8,9, og i centrale nervesystem sygdomme, herunder Alzheimers sygdom (ad), multipel sklerose (MS); og i aggression10.
De fleste af de KDM1A hæmmende forbindelser i klinisk udvikling er cyclopropylamin derivater og hæmmer proteinet ved kovalent binding til dets Flavin adenin dinucleotid (fad) cofaktor11. Hæmning af KDM1A inducerer genekspression ændringer, men disse ændringer varierer enormt på tværs af væv, celletyper, eller sygdomstilfælde. Hæmning af KDM1A ændrer også Histon mærker12, men disse ændringer er generelt produceres lokalt på et bestemt sted i genomet, og er igen, meget væv og celle specifikke.
Protokollen blev udviklet til direkte at måle KDM1A Target engagement i biologiske prøver og er blevet optimeret til brug med cyclopropylamin afledte hæmmere. Analysen er baseret på ELISA-teknologi og analyserer parallelt, samlet og gratis (dvs. ubundet af inhibitor) KDM1A i et indfødt proteinekstrakt fra en biologisk prøve i en solid fase analyse. Som et første skridt lyses den biologiske prøve i nærværelse af den biotinylerede KDM1A selektive chemoprobe og-88113,14, afledt af den selektive KDM1A inhibitor ORY-1001 (iadademstat), en potent hæmmer af KDM1A i kliniske udvikling til behandling af onkologiske sygdomme. Chemoprobe har en IC50 for KDM1A af 120 nm og omfatter en fad bindende delen forbundet med en biotinyleret polyethylenglycol (peg)-hale. Chemoprobe binder udelukkende til den frie KDM1A, men ikke til den inhibitor bundne KDM1A i prøven. Efter chemoprobe bindingen optages de KDM1A, der indeholder komplekser i prøven, på mikrotiterplader med streptavidin belagt overflade for at bestemme fri KDM1A eller på plader belagt med et monoklonalt anti-KDM1A Capture-antistof for at bestemme total KDM1A. Efter vask inkuberet begge plader med anti-KDM1A-detektionsanti stof, vaskes igen og inkuberet med et sekundært HRP-konjugeret æsel anti-kanin IgG-antistof til påvisning ved hjælp af et luminescerende substrat og kvantificering ved måling af relative lysenheder (RLU) i et luminometer (figur 1).
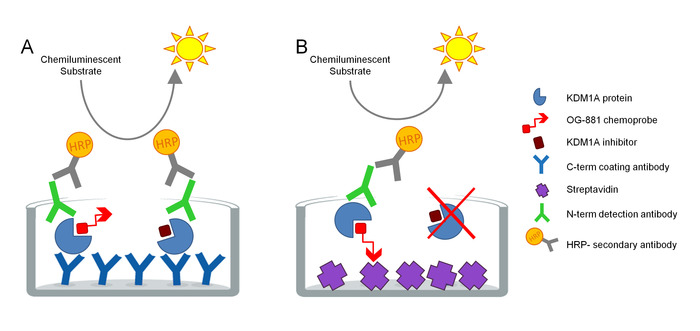
Figur 1. Skema af ELISA enzym forbundet chemoprobe immunoabsorber ende assay for KDM1A Target engagement: A) bestemmelse af total KDM1A ved hjælp af sandwich Elisa og B) bestemmelse af gratis KDM1A ved hjælp af chemoprobe Elisa. Venligst klik her for at se en større version af dette tal.
En standardkurve er inkluderet i begge ELISA-plader for at verificere lineariteten af hver analyse. Bestemmelsen af KDM1A Target engagement i hver prøve beregnes derefter som en relativ værdi til den præ-dosis eller køretøj behandlet prøve.
Protocol
Representative Results
Discussion
Protokollen præsenteres her blev udviklet til direkte at måle KDM1A Target engagement ved hjælp af en roman KDM1A chemoprobe Capture baseret ELISA. Metoden er blevet valideret på dyrkede humane cellelinjer og ex vivo-prøver fra menneske, rotte og mus og bavian (herunder Pbm’er, lunge, hjerne, hud, tumorer), men kan let anvendes på andre arter, hvor KDM1A antistof Target epitoper og katalytisk Center er bevaret. Da OG-881 er en aktivitet baseret chemoprobe, er prøve kvaliteten vigtig og korrekt manipulation og beva…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne undersøgelse blev finansieret af Oryzon Genomics. S.A., Hoffman-La Roche, og delvist understøttet af CIIP-20152001 og RETOS Collaboration program RTC-2015-3332-1.
Materials
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
References
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601 (2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54 (2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905 (2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Gonz#225;lez, E. C., Maes, T., Crusat, C. M., Mu#241;oz, A. O. Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000 (2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192 (2017).

