Medição direta do KDM1A Target Engagement usando imunoensaios baseados em Quimiosonda
Summary
Aqui, apresentamos um protocolo para medir KDM1A engajamento alvo em uma célula humana ou animal, tecido ou amostras de sangue tratadas com inibidores da KDM1A. O protocolo emprega a marcação do chemoprobe da enzima livre de KDM1A e a quantificação direta da ocupação do alvo usando immunoensaios quimioprobe-baseados e pode ser usada em estudos pré-clínicos e clínicos.
Abstract
A avaliação do engajamento alvo, definida como a interação de um fármaco com a proteína para a qual foi projetada, é um requisito básico para a interpretação da atividade biológica de qualquer composto no desenvolvimento de fármacos ou em projetos de pesquisa básica. No Epigenetics, o acoplamento do alvo é avaliado o mais frequentemente pela análise de marcadores do proxy em vez de medir a União do composto ao alvo. Os leituras biológicos a jusante que foram analisados incluem a modulação da marca do histona ou as mudanças da expressão de Gene. KDM1A é uma desmetililase de lisina que remove grupos metilo de mono e H3K4 dimetilizados, uma modificação associada ao silenciamento da expressão gênica. A modulação dos marcadores do proxy é dependente do tipo e da função da pilha da make-up genética das pilhas investigadas, que pode fazer a interpretação e a comparação do Cruz-caso completamente difícil. Para contornar estes problemas, um protocolo versátil é apresentado para avaliar os efeitos da dose e a dinâmica do acoplamento direto do alvo de KDM1A. O ensaio descrito faz uso de uma quimiosonda KDM1A para capturar e quantificar a enzima desinibida, pode ser amplamente aplicada a células ou amostras de tecido sem a necessidade de modificação genética, tem uma excelente janela de detecção, e pode ser usado tanto para pesquisa básica e análise de amostras clínicas.
Introduction
O demetilase específico 1 de lysine (KDM1A)1 é um demetilase envolvido no controle da transcrição do gene. Esta proteína emergiu como um alvo farmacológico candidato2 em Oncologia; incluindo leucemia mielóide aguda3 (AML), síndrome de mielodisplasia (MDS)4, mielofibrose (MF)5,6, câncer de pulmão de pequenas células (SCLC)7; em doença falciforme (SCD)8,9, e em doenças do sistema nervoso central, incluindo a doença de Alzheimer (AD), esclerose múltipla (MS); e na agressão10.
A maioria dos compostos inibidores da KDM1A no desenvolvimento clínico são derivados da ciclopropilamina e inibem a proteína por ligação covalente ao seu cofator11de flavina-dinucleotídeo (FAD). A inibição da KDM1A induz alterações na expressão gênica, mas essas alterações variam enormemente nos tecidos, tipos de células ou casos de doença. A inibição da KDM1A também altera as marcas de histona12, mas essas mudanças são geralmente produzidas localmente em um local específico no genoma, e são novamente, altamente tecido e células específicas.
O protocolo foi desenvolvido para medir diretamente o engajamento alvo de KDM1A em amostras biológicas e foi otimizado para o uso com inibidores derivados da ciclopropilamina. O ensaio é baseado na tecnologia ELISA e analisa, em paralelo, total e livre (ou seja, não acoplado por inibidor) KDM1A em um extrato de proteína nativa de uma amostra biológica em um ensaio de fase sólida. Como primeiro passo, a amostra biológica é lisada na presença da quimiosonda seletiva biotinilada KDM1A og-88113,14, derivada do inibidor seletivo KDM1A Ory-1001 (iadademstat), um potente inibidor da KDM1A em clínica desenvolvimento para o tratamento da doença oncológica. O chemoprobe tem um IC50 para KDM1A de 120 nanômetro e inclui um metade obrigatório do FAD lig a um polietileno biotinylated glicol (PEG)-cauda. A quimiosonda liga-se exclusivamente à KDM1A livre, mas não à KDM1A associada ao inibidor na amostra. Após a ligação do chemoprobe, os KDM1A que contêm complexos na amostra são capturados em placas do de microtitulação com a superfície revestida estreptavidina para determinar o KDM1A livre, ou em placas revestidas com um anticorpo KDM1A monoclonal da captação para determinar o KDM1A total. Após a lavagem, ambas as placas são incubadas com um anticorpo de detecção KDM1A, lavadas novamente, e incubadas com um anticorpo de IgG anticoelho conjugado HRP secundário para detecção usando um substrato luminescente e quantificação por medição relativa de luz (RLU) num luminômetro (Figura 1).
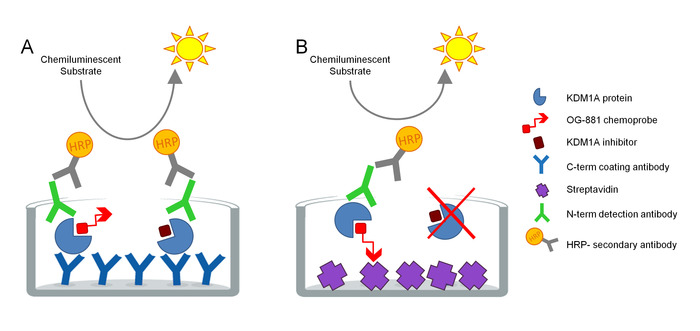
Figura 1. Esquema de ELISA enzima lig o ensaio immunoabsorvente do chemoprobe para o acoplamento do alvo KDM1A: A) determinação do total KDM1A usando o sanduíche ELISA e B) determinação do KDM1A livre usando o quimioprobe Elisa. Por favor clique aqui para ver uma versão maior desta figura.
Uma curva padrão é incluída em ambas as placas ELISA para verificar a linearidade de cada ensaio. A determinação do engajamento alvo de KDM1A em cada amostra é então calculada como um valor relativo para a amostra pré-dose ou tratada pelo veículo.
Protocol
Representative Results
Discussion
O protocolo apresentado aqui foi desenvolvido para medir diretamente o engajamento alvo de KDM1A usando um romance KDM1A com base na captura de quimiosonda ELISA. O método foi validado em linhagens de células humanas cultivadas e amostras ex vivo de humano, rato e rato e babuíno (incluindo PBMCs, pulmão, cérebro, pele, tumores), mas pode ser prontamente aplicada a outras espécies em que o anticorpo KDM1A resumos alvo e catalítico Centro são conservadas. Como OG-881 é um quimioprobe baseado na atividade, a qualid…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Este estudo foi financiado pela ORYZON Genomics. S.A., Hoffman-la Roche, e parcialmente apoiada pelo programa de colaboração CIIP-20152001 e RETOS RTC-2015-3332-1.
Materials
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
References
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601 (2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54 (2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905 (2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Gonz#225;lez, E. C., Maes, T., Crusat, C. M., Mu#241;oz, A. O. Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000 (2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192 (2017).

