Misurazione diretta del coinvolgimento target KDM1A utilizzando Immunoassays basate su Chemoprobe
Summary
Qui, presentiamo un protocollo per misurare kDM1A target engagement in una cellula umana o animale, tessuto o campioni di sangue trattati con inibitori KDM1A. Il protocollo utilizza l’etichettatura chemioprobe dell’enzima KDM1A gratuito e la quantificazione diretta dell’occupazione target utilizzando immunoasisti basati sulla chemiospa e può essere utilizzato in studi preclinici e clinici.
Abstract
La valutazione dell’impegno target, definita come l’interazione di un farmaco con la proteina per cui è stato progettato, è un requisito fondamentale per l’interpretazione dell’attività biologica di qualsiasi composto nello sviluppo di farmaci o nei progetti di ricerca di base. Nell’epigenetica, l’impegno target è più spesso valutato mediante l’analisi dei marcatori proxy invece di misurare l’unione del composto al bersaglio. Le letture biologiche a valle che sono state analizzate includono la modulazione del marchio dell’istorio o i cambiamenti dell’espressione genica. KDM1A è una lisila demetila si riparte da H3K4 mono e dimetilata, una modifica associata al silenziamento dell’espressione genica. La modulazione dei marcatori proxy dipende dal tipo di cellula e dalla funzione della composizione genetica delle cellule studiate, il che può rendere piuttosto difficile l’interpretazione e il confronto tra maiuscole e minuscole. Per aggirare questi problemi, viene presentato un protocollo versatile per valutare gli effetti della dose e la dinamica del coinvolgimento diretto del target KDM1A. L’analisi descritta fa uso di una chemoprobe KDM1A per catturare e quantificare l’enzima disinibito, può essere ampiamente applicato a cellule o campioni di tessuto senza necessità di modificazione genetica, ha un’eccellente finestra di rilevamento e può essere utilizzato sia per la ricerca di base e l’analisi di campioni clinici.
Introduction
La lisina demetilasi specifica 1 (KDM1A)1 è un demetilare coinvolto nel controllo della trascrizione genica. Questa proteina è emersa come bersaglio farmacologico candidato2 in oncologia; tra cui acuta mieloide Leucemia3 (AML), Sindrome da Mielodisplasia (MDS)4, Mielofibrosi (MF)5,6, Cancro polmonare a piccole cellule (SCLC)7; nella malattia delle cellule falciformi (SCD)8,9, e nelle malattie del sistema nervoso centrale tra cui il morbo di Alzheimer (AD), la sclerosi multipla (MS); e nell’aggressione10.
La maggior parte dei composti inibitori di KDM1A nello sviluppo clinico sono derivati della ciclopropilaree e inibiscono la proteina legandosi covalente al suo cofattore dinucleotide di flavin adenina (FAD)11. L’inibizione di KDM1A induce cambiamenti nell’espressione genica, ma questi cambiamenti variano enormemente tra i tessuti, i tipi di cellule o i casi di malattia. L’inibizione di KDM1A cambia anche i segni itoni12, ma questi cambiamenti sono generalmente prodotti localmente in un sito specifico del genoma, e sono ancora una volta, altamente tessuto e cellule specifiche.
Il protocollo è stato sviluppato per misurare direttamente l’impegno del target KDM1A in campioni biologici ed è stato ottimizzato per l’uso con inibitori derivati dalla ciclopropile. Il saggio si basa sulla tecnologia ELISA e analizza, in parallelo, Total e Free (cioè non legato dall’inibitore) KDM1A in un estratto di proteina autoctona da un campione biologico in un saggio in fase solida. Come primo passo, il campione biologico viene lizzato in presenza della chemoprobe selettiva KDM1A biomutlata OG-88113,14, derivata dall’inibitore selettivo di KDM1A ORY-1001 (iadademstat), un potente inibitore di KDM1A in sviluppo per il trattamento della malattia oncologica. La chemiosche ha un IC50 per KDM1A di 120 nM e comprende una moiety legante FAD collegata a una coda di glicole di polietilene biotinyato (PEG). La chemiosa si lega esclusivamente al KDM1A gratuito, ma non al KDM1A inibitore nel campione. Dopo la rilegatura della chemiosa, il KDM1A contenente complessi nel campione vengono catturati su piastre di microtitro con superficie rivestita di streptavidin per determinare kDM1A libero, o su piastre rivestite con un anticorpo di cattura anti-KDM1A monoclonale per determinare il totale KDM1A. Dopo il lavaggio, entrambe le piastre vengono incubate con un anticorpo anti-rilevamento anti-KDM1A, lavate di nuovo e incubate con un anticorpo igG con asino concatenato HRP secondario per la rilevazione utilizzando un substrato luminescente e la quantificazione misurando unità di luce (RLU) in un luminometro (Figura 1).
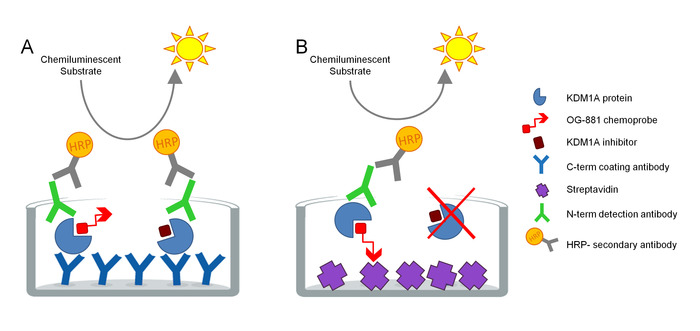
come illustrato nella Figura 1. Schema di ELISA Enzima collegato chemioprobeata saggio immunoassorante per KDM1A target engagement: A) Determinazione del totale KDM1A utilizzando sandwich ELISA e B) Determinazione di KDM1A gratuito utilizzando chemioprobe ELISA. Fare clic qui per visualizzare una versione più grande di questa figura.
In entrambe le piastre ELISA è inclusa una curva standard per verificare la linearità di ogni analisi. La determinazione dell’impegno target KDM1A in ogni campione viene quindi calcolata come valore relativo al campione pre-dose o al veicolo trattato.
Protocol
Representative Results
Discussion
Il protocollo qui presentato è stato sviluppato per misurare direttamente il coinvolgimento del target KDM1A utilizzando una nuova chemoprobe KDM1A che coglie la base ELISA. Il metodo è stato convalidato su linee cellulari umane coltivate e campioni di ex vivo da esseri umani, ratti e topi e babbuini (compresi PMC, polmone, cervello, pelle, tumori), ma può essere facilmente applicato ad altre specie in cui l’anticorpo KDM1A bersaglio epitopi e cataliti centro sono conservati. Poiché OG-881 è una chemioprobe basata s…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo studio è stato finanziato da Oryzon Genomics. S.A., Hoffman-La Roche e parzialmente supportati dal programma di collaborazione CIIP-20152001 e RETOS RTC-2015-3332-1.
Materials
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
References
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601 (2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54 (2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905 (2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Gonz#225;lez, E. C., Maes, T., Crusat, C. M., Mu#241;oz, A. O. Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000 (2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192 (2017).

