Direkte måling av KDM1A Target Engagement ved hjelp av Chemoprobe-baserte immunanalyser
Summary
Her presenterer vi en protokoll for å måle KDM1A mål engasjement i et menneske eller dyr celle, vev eller blodprøver behandlet med KDM1A-hemmere. Protokollen sysselsetter chemoprobe merking av det frie KDM1A-enzymet og direkte kvantifisering av mål okkupasjonen ved hjelp av chemoprobe-baserte immunanalyser og kan brukes i prekliniske og kliniske studier.
Abstract
Vurderingen av målet engasjement, definert som samspillet av et medikament med proteinet den ble utformet for, er et grunnleggende krav for tolkningen av biologisk aktivitet av enhver forbindelse i narkotika utvikling eller i grunnleggende forskningsprosjekter. I epigenetikk, mål engasjement er oftest vurdert av analysen av proxy markører i stedet for å måle foreningen av sammensatte til målet. Nedstrøms biologiske readouts som har blitt analysert inkluderer histone merke modulering eller genuttrykk endringer. KDM1A er en lysin-demethylase som fjerner metyl-grupper fra mono-og dimethylated H3K4, en modifikasjon knyttet til deaktivering av genuttrykk. Modulering av proxy markører er avhengig av celle type og funksjon av genetisk make-up av cellene undersøkt, som kan gjøre tolkning og kryss-saken sammenligning ganske vanskelig. For å omgå disse problemene, presenteres en allsidig protokoll for å vurdere dose effekter og dynamikk av direkte KDM1A mål engasjement. Analysen beskrevet gjør bruk av en KDM1A chemoprobe å fange og kvantifisere uhemmet enzym, kan grovt brukes på celler eller vevsprøver uten behov for genetisk modifisering, har et utmerket vindu for deteksjon, og kan brukes både for grunnleggende forskning analyse av kliniske prøver.
Introduction
Lysin spesifikke demethylase 1 (KDM1A)1 er en demethylase involvert i kontroll av genet transkripsjon. Dette proteinet har dukket opp som en kandidat farmakologisk mål2 i onkologi; inkludert akutt myelogen leukemi3 (AML), myelodysplasi syndrom (MDS)4, myelofibrose (MF)5,6, liten celle lungekreft (SCLC)7; i sigdcellesykdom (SCD)8,9, og i sentrale nervesystemet sykdommer inkludert Alzheimers sykdom (AD), multippel sklerose (MS); og i aggresjon10.
Mesteparten av KDM1A hemme forbindelser i klinisk utvikling er cyclopropylamine derivater og hemme proteinet ved kovalente binding til sin Flavin adenine dinucleotide (FAD) kofaktor11. Hemming av KDM1A induserer genuttrykk endringer, men disse endringene varierer enormt på tvers av vev, celletyper, eller sykdomstilfeller. Hemming av KDM1A også endringer histone merker12, men disse endringene er vanligvis produsert lokalt på et bestemt sted i Genova, og er igjen, svært vev og celle spesifikke.
Protokollen ble utviklet for å direkte måle KDM1A mål engasjement i biologiske prøver og har blitt optimalisert for bruk med cyclopropylamine avledet hemmere. Analysen er basert på ELISA teknologi og analyser, parallelt, total og gratis (dvs. ubundet av inhibitor) KDM1A i en innfødt protein ekstrakt fra en biologisk prøve i en solid fase analysen. Som et første skritt er den biologiske prøven lysert i nærvær av biotinylated KDM1A selektive chemoprobe og-88113,14, avledet fra den selektive KDM1A inhibitor ORY-1001 (iadademstat), en potent hemmer av KDM1A i klinisk utvikling for behandling av onkologiske sykdom. Den chemoprobe har en IC50 for KDM1A av 120 nM og inkluderer en kjepphest bindende moiety knyttet til en biotinylated polyetylen GLYKOL (PEG)-tail. Chemoprobe binder utelukkende til den frie KDM1A, men ikke til den hemmer-bundne KDM1A i prøven. Etter den chemoprobe bindingen blir KDM1A som inneholder komplekser i prøven fanget opp på mikrotiterbrønnene plater med streptavidin belagt overflate for å bestemme fri KDM1A, eller på plater belagt med et monoklonale anti-KDM1A fangst antistoff for å fastslå total KDM1A. Etter vask er begge platene inkubert med et anti-KDM1A deteksjon antistoff, vasket igjen, og inkubert med et sekundært HRP-bøyd esel anti-kanin IgG antistoff for påvisning ved hjelp av et selvlysende substrat og kvantifisering ved å måle relative lys enheter (RLU) i en luminometeret (figur 1).
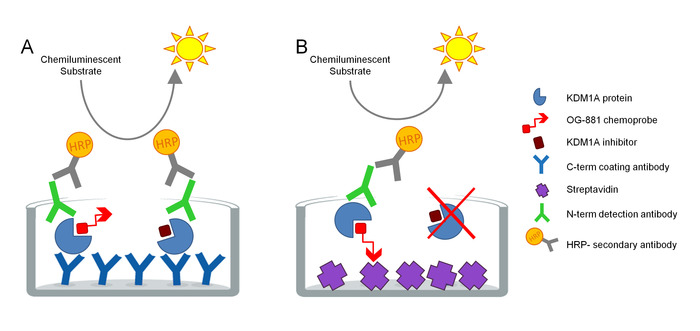
Figur 1. Skjema av ELISA enzym knyttet chemoprobe immunoabsorbent analysen for KDM1A Target engasjement: A) fastsettelse av total KDM1A bruker sandwich Elisa og B) fastsettelse av gratis KDM1A bruker CHEMOPROBE Elisa. Vennligst klikk her for å se en større versjon av dette tallet.
En standard kurve er inkludert i begge ELISA-platene for å verifisere linearitet til hver analyse. Fastsettelse av KDM1A mål engasjement i hver prøve blir deretter beregnet som en relativ verdi til pre-dose eller kjøretøy behandlet prøven.
Protocol
Representative Results
Discussion
Protokollen som presenteres her ble utviklet for å direkte måle KDM1A mål engasjement ved hjelp av en roman KDM1A chemoprobe fangst basert ELISA. Metoden har blitt validert for kulturperler menneskelige cellelinjer og ex vivo prøver fra menneske, rotte og mus og bavian (inkludert Pbmc, lunge, hjerne, hud, svulster), men kan lett brukes til andre arter der KDM1A antistoff målet epitopes og katalysator senteret er bevart. AS OG-881 er en aktivitet basert chemoprobe, er utvalget kvaliteten viktig og forsvarlig manipula…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne studien ble finansiert av Oryzon Genomics. S.A., Hoffman-La Roche, og delvis støttes av CIIP-20152001 og RETOS samarbeid programmet RTC-2015-3332-1.
Materials
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
References
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601 (2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54 (2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905 (2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Gonz#225;lez, E. C., Maes, T., Crusat, C. M., Mu#241;oz, A. O. Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000 (2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192 (2017).

