Direkt mätning av KDM1A mål engagemang med Chemoprobe-baserade Immunoassays
Summary
Här presenterar vi ett protokoll för att mäta KDM1A mål engagemang i en människa eller djur cell, vävnad eller blodprov som behandlats med KDM1A-hämmare. Protokollet sysselsätter chemoprobe märkning av den fria KDM1A enzym och direkt kvantifiering av mål ockupationen med chemoprobe-baserade immunanalyser och kan användas i prekliniska och kliniska studier.
Abstract
Bedömningen av målet engagemang, definierad som samspelet mellan ett läkemedel med proteinet det utformades för, är ett grundläggande krav för tolkningen av den biologiska aktiviteten hos någon förening i läkemedelsutveckling eller i grundläggande forskningsprojekt. I epigenetik bedöms mål engagemanget oftast av analysen av proxymarkörer istället för att mäta föreningen av substansen till målet. Nedströms biologiska avläsningar som har analyserats inkluderar Histon mark modulering eller genuttryck förändringar. KDM1A är en lysin-demetyleras som avlägsnar metylgrupper från mono-och dimetylerade H3K4, en modifiering som är förknippad med ljuddämpning av genuttryck. Modulering av proxy markörer är beroende av celltyp och funktion av den genetiska make-up av de undersökta cellerna, som kan göra tolkning och Cross-Case jämförelse ganska svårt. För att kringgå dessa problem, ett mångsidigt protokoll presenteras för att bedöma dos effekter och dynamik i direkt KDM1A mål engagemang. Analysen beskrivs gör användning av en KDM1A chemoprobe att fånga och kvantifiera ohämmade enzym, kan i stort sett tillämpas på celler eller vävnadsprover utan behov av genetisk modifiering, har ett utmärkt fönster för detektion, och kan användas både för grundforskning och analys av kliniska prover.
Introduction
Lysin-specifika demetyleras 1 (KDM1A)1 är en demetyleras inblandade i kontrollen av gentranskription. Detta protein har vuxit fram som en kandidat farmakologisk mål2 i onkologi; inklusive akut myeloisk leukemi3 (AML), myelodysplasia syndrom (MDS)4, myelofibros (MF)5,6, småcellig Lung cancer (SCLC)7; i sicklecellanemi (SCD)8,9, och i centralanervsystemet sjukdomar inklusive Alzheimers sjukdom (AD), multipel skleros (MS); och i aggression10.
De flesta av de KDM1A hämmande föreningar i klinisk utveckling är cyklopropylamin derivat och hämmar proteinet genom kovalenta bindning till dess Flavin adenin–dinukleotid (FAD) kofaktor11. Hämning av KDM1A inducerar förändringar av genuttryck, men dessa förändringar varierar enormt mellan vävnader, celltyper eller sjukdoms fall. Hämning av KDM1A ändrar också Histon Marks12, men dessa förändringar är i allmänhet produceras lokalt på en specifik plats i genomet, och är återigen, mycket vävnad och cell specifika.
Protokollet utvecklades för att direkt mäta KDM1A mål engagemang i biologiska prover och har optimerats för användning med cyklopropylamin härledda hämmare. Analysen är baserad på ELISA-teknik och analyserar, parallellt, totalt och gratis (dvs. obunden av inhibitor) KDM1A i ett inhemskt proteinextrakt från ett biologiskt prov i en solid fas analys. Som ett första steg, är det biologiska provet lyserat i närvaro av en KDM1A selektiva chemoprobe og-88113,14, härrör från selektiv KDM1A inhibitor ORY-1001 (iadademstat), en potent hämmare av KDM1A i kliniska utveckling för behandling av onkologisk sjukdom. Den chemoprobe har en IC50 för KDM1A av 120 Nm och innehåller en modefluga bindande del kopplad till en en polyetylenglykol (PEG)-svans. Chemosonden binder exklusivt till den fria KDM1A, men inte till inhibitoren-begränsar KDM1A i ta prov. Efter chemoprobe bindning, de KDM1A som innehåller komplex i provet fångas på mikrotiterplattor med konjugerat belagd yta för att bestämma fri KDM1A, eller på plattor belagda med en monoklonal anti-KDM1A Capture antikropp för att bestämma totala KDM1A. Efter tvättning inkuberas båda plattorna med en anti-KDM1A detektionsantikropp, tvättas igen, och inkuberas med en sekundär HRP-konjugerad åsna anti-kanin IgG antikropp för detektion med hjälp av ett självlysande substrat och kvantifiering genom att mäta relativa ljusenheter (RLU) i en luminometern (figur 1).
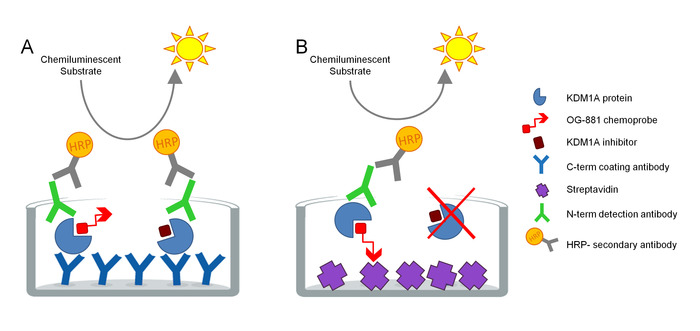
Figur 1. Schema för ELISA enzym länkade chemoprobe immunoabsorberande test för KDM1A Target Engagement: A) bestämning av total KDM1A med Sandwich-Elisa och B) bestämning av fri KDM1A med hjälp av chemoprobe Elisa. Vänligen klicka här för att se en större version av denna siffra.
En standardkurva ingår i båda ELISA-plattorna för att kontrollera Lineariteten hos varje analys. Bestämning av KDM1A-målengagemang i varje prov beräknas sedan som ett relativt värde för det före dos-eller vehikel-behandlade provet.
Protocol
Representative Results
Discussion
Det protokoll som presenteras här utvecklades för att direkt mäta KDM1A mål engagemang med hjälp av en roman KDM1A chemoprobe Capture baserade ELISA. Metoden har validerats på odlade humana cellinjer och ex vivo prover från människa, råtta och mus och babian (inklusive pbmcs, lung, hjärna, hud, tumörer), men kan lätt appliceras på andra arter där KDM1A antikropp mål epitoper och katalytisk Center bevaras. Som OG-881 är en verksamhet baserad chemoprobe, provet kvalitet är viktigt och korrekt manipulation …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denna studie finansierades av Oryzon Genomics. S.A., Hoffman-La Roche, och delvis med stöd av CIIP-20152001 och RETOS Collaboration program RTC-2015-3332-1.
Materials
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
References
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601 (2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54 (2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905 (2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Gonz#225;lez, E. C., Maes, T., Crusat, C. M., Mu#241;oz, A. O. Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000 (2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192 (2017).

