Brug af enkelt molekyle fluorescerende i Situ hybridisering (SM-fisk) til tal og Oversæt mRNAs i Murine oocyter
Summary
Hvis du vil reproducerbar tælle antallet af mRNAs i individuelle oocytter, enkelt molekyle RNA fluorescens in situ var hybridisering (RNA-fisk) optimeret til ikke-tilhænger celler. Oocytter blev indsamlet, hybridiseret med udskrift specifikke sonder og kvantificeres ved hjælp af et billede kvantificering software.
Abstract
Nuværende metoder, der rutinemæssigt anvendes til at kvantificere mRNA i oocytter og embryoner omfatter digitale reverse-transskription polymerase kædereaktion (dPCR), kvantitative, real-time RT-PCR (RT-qPCR) og RNA sekvensering. Da disse teknikker er udført ved hjælp af en enkelt oocyt eller embryo, er lav-kopi mRNAs ikke pålideligt registreret. For at løse dette problem, kan æg eller embryoner samles sammen til analyse. men dette fører ofte til stor variation blandt prøver. I denne protokol beskriver vi brugen af fluorescens i situ hybridisering (FISH) ved hjælp af forgrenede DNA kemi. Denne teknik identificerer den rumlige mønster af mRNAs i individuelle celler. Når teknikken er kombineret med stedet at finde og spore computersoftware, kan overfloden af mRNAs i cellen også kvantificeres. Brug af denne teknik, der er nedsat variabilitet i en eksperimenterende gruppe og færre oocytter og embryoner er forpligtet til at påvise betydelige forskelle mellem eksperimentelle grupper. Kommercielt tilgængelige forgrenet DNA SM-fisk kits har været optimeret til at registrere mRNAs i sektioneret væv eller vedhængende celler på dias. Men oocyter effektivt overholder ikke dias og nogle reagenser i sættet var for hårdt, hvilket resulterer i oocyt lysering. For at forhindre denne lysis, blev flere ændringer foretaget til fisk kit. Specifikt, erstattet oocyt permeabilization og vask buffere designet til immunfluorescens af oocytter og embryoner leverandørejet bufferne. Permeabilization, vasker og inkubationer med sonder og forstærker blev udført i 6-godt plader og oocyter blev placeret på dias i slutningen af protokollen ved hjælp af montering medier. Disse ændringer var i stand til at overvinde begrænsningerne af den kommercielt tilgængeligt kit, navnlig oocyt lysering. For at præcist og reproducerbar tælle antallet af mRNAs i individuelle oocytter, blev computersoftware brugt. Sammen, repræsenterer denne protokol et alternativ til PCR og sekventering at sammenligne udtryk for specifikke udskrifter i enkelte celler.
Introduction
Reverse transkriptase Polymerasekædereaktionen (PCR) har været guldstandarden til mRNA-kvantificering. To assays, digital PCR (dPCR)1 og kvantitative, real time PCR (qPCR)2 bruges aktuelt. Af de to PCR teknikker har dPCR større følsomhed end qPCR tyder på, at det kunne bruges til at måle mRNA overflod i enkelte celler. Men i vores hænder, dPCR analyse af lav overflod mRNAs i puljer af 5 til 10 oocyter pr. hver eksperimentel prøve har produceret data med lav reproducerbarhed og høj variation3. Dette er sandsynligvis på grund af eksperimentelle fejl forbundet med RNA udvinding og reverse transkription effektivitet. RNA sekventering har også udført ved hjælp af en enkelt mus og menneskelige oocyter4,5. Denne teknik kræver cDNA forstærkning trin, der kræves for den bibliotek generation, hvilket sandsynligvis øger variation inden for en eksperimentel musikgruppe. Endvidere må lav overflod udskrifter ikke kunne påvises. Selvom sekventering priser er gået ned de sidste par år, kan det stadig være omkostningseffektivt uoverkommelige på grund af de høje udgifter til Bioinformatik analyser. Endelig, mRNA lokalisering er en dynamisk proces med rumlige ændringer bidrager til protein funktion6. Derfor, vi satte sig for at vedtage en teknik, der vil producere nøjagtige og reproducerbare kvantitative foranstaltninger og lokalisering af individuelle mRNAs i enkelt oocyter.
Forgrenede DNA koblet til fluorescens i situ hybridisering forstærker fluorescens signal snarere end forstærkende RNA/cDNA muliggør påvisning af enkelt mRNAs i enkelte celler 7,8,9. Analysen udføres gennem en række hybridisering, forstærkning (ved hjælp af forgrenede DNA) og fluorescens mærkning skridt for at forstærke fluorescens signal7. Teknikken, der begynder med bindingen af 18 – 25 base oligonukleotid sonde-par, der supplerer en specifik mRNA3,8,10. Femten til tyve sonde par er designet til hver afskrift sikring specificitet for target udskrift. MRNA-specifikke hybridisering er efterfulgt af pre-forstærker og forstærker sonder, der danner et forgrenet konfiguration. Ca, 400 etiket fluorophores binder til hver forstærker, hvilket resulterer i en 8000-fold stigning i fluorescens giver mulighed for påvisning af individuelle mRNAs (figur 1)11.
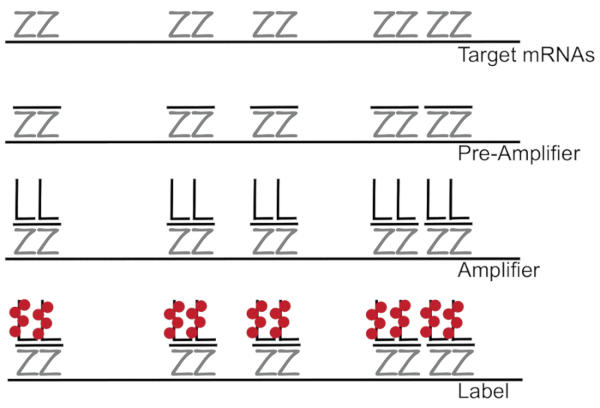
Figur 1: skematisk af SM-fisk protokol. Sekventiel hybridisering af udskrift specifikke sonde, forgrenet DNA forstærker og fluorophore til et mål mRNA er vist. Venligst klik her for at se en større version af dette tal.
Tidligere undersøgelser ved hjælp af enkelt molekyle fluorescens i situ hybridisering (SM-fisk) lokaliseret β-actin mRNAs i individuelle neuroner12 og humant papillomvirus DNA i livmoderhalskræft celle linjer7. Computersoftware stedet at finde og programmet til sporing af identificerer enkelte punktformet fluorescerende signal og held har været anvendt til at opgøre antallet af mRNAs i hver celle3,13.
Baseret på resultaterne af mRNA påvisning i neuroner12, hypotese vi at SM-fisk ville vise sig at være et nyttigt redskab til at kvantificere udskrift niveauer i murine oocytter og embryoner herunder lav overflod mRNAs. Men teknikken er optimeret til brug med vedhængende fast celler og formaldehyd fast paraffin indlejret (FFPE) væv sektioner. Oocyter kan ikke overholde et dias, selv når de er belagt med Poly-L-lysin. Derudover er de mere skrøbelige end somatiske celler og væv sektioner resulterer i celle lysis når de udsættes for nogle af leverandørejet bufferne i kommercielt tilgængelige kits3. For at overvinde disse udfordringer, oocyter fast og manuelt overført mellem dråber af buffere. Desuden blev permeabilization og vask buffere i kits udskiftet for at reducere celle lysering. Foruddesignede sonder er købt sammen med fisk kit eller specifikke udskrifter kan bestilles. Hver proprietære sonde sæt er tilgængelige i en af tre fluorescens kanaler (C1, C2 og C3) at tillade multiplexing. I den aktuelle eksperiment var murine oocyter dual-farvede og kvantificeret ved hjælp af en C2 Nanog sonde og en C3 Pou5f1 sonde. Disse prober blev udvalgt på baggrund af de rapporterede udtryk for Nanog og Pou5f1 i oocytter og embryoner. Ved afslutningen af hybridisering trin blev oocyter placeret i dråber af anti-fade montering medier for ansøgning til histologisk dias. Konfokal billeder blev brugt til at opgøre antallet af punktformet fluorescerende signaler, som repræsenterer individuelle mRNAs. Ud over at kvantificere mRNAs, imaging viste også den geografiske fordeling af den specifikke mRNA i cellen, er som andre RNA kvantificering metoder ude af stand til at opnå. Denne teknik vist sig for at have lav variation inden for en eksperimentel musikgruppe der tillader anvendelse af mindre antal oocyter i hver eksperimentelle gruppe for at identificere væsentlige forskelle mellem eksperimentelle grupper3.
Protocol
Representative Results
Discussion
En række mindre trin under protokollen vil sikre vellykkede fluorescens og nøjagtige optællinger af mRNAs. Først skal protokollen udføres straks efter samling og fiksering af oocyter. Bemærk, at PVP er tilføjet til 4% PARAFORMALDEHYD fiksering buffer til at forhindre oocyter klistrer til hinanden. Vi fandt, at det er nødvendigt at udføre eksperimentet umiddelbart efter indsamling og fiksering af oocyter. Enhver forsinkelse resulterer i en meget lavere fluorescens signal, som ville resultere i undercounting af ud…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi takker Dr. Daniel R. Larson for hans generøse hjælp med installation og brug af i stedet at finde og programmet til sporing af 13 og teknisk støtte fra University of Nebraska Lincoln mikroskopi kerne for konfokalmikroskopi billeddannelse. Denne undersøgelse er et bidrag af University of Nebraska landbrugs forskning Division, Lincoln, Nebraska og blev støttet af UNL Hatch midler (NEB-26-206/tiltrædelse af-232435 og NEB-26-231/tiltrædelse nummer-1013511).
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

