Gebruik van één molecuul fluorescentie In Situ hybridisatie (SM-vis) om Quantify en lokaliseer mRNAs in lymfkliertest eicellen
Summary
Reproducibly het om getallen te tellen van mRNAs in individuele eicellen, enkel molecuul RNA fluorescentie in situ is hybridisatie (RNA-vis) geoptimaliseerd voor niet-aanhanger cellen. Eicellen werden verzameld, gekruist met het transcript specifieke sondes en gekwantificeerd aan de hand van een afbeelding kwantificering software.
Abstract
Huidige methoden routinematig gebruikt om mRNA te kwantificeren in eicellen en embryo’s omvatten digitale omgekeerde-transcriptie polymerase-kettingreactie (dPCR), kwantitatieve, real-time RT-PCR (RT-qPCR) en RNA sequencing. Wanneer deze technieken worden uitgevoerd met behulp van een enkele oöcyt of embryo, zijn laag-kopie mRNAs niet betrouwbaar gedetecteerd. Om dit probleem te verhelpen, kunnen eicellen of embryo’s worden gepoold samen voor analyse; Dit leidt echter vaak tot hoge veranderlijkheid onder de monsters. In dit protocol beschrijven we het gebruik van fluorescentie in situ hybridisatie (FISH) met behulp van vertakte DNA chemie. Deze techniek geeft het ruimtelijk patroon van mRNAs in afzonderlijke cellen. Wanneer de techniek is gekoppeld aan het vinden van de plek en de opsporingssoftware van de computer, kan de overvloed van mRNAs in de cel ook worden gekwantificeerd. Met behulp van deze techniek, er is verminderde variabiliteit binnen een experimentele groep en minder eicellen en embryo’s zijn nodig voor de detectie van verschillen tussen experimentele groepen. Verkrijgbare vertakt-DNA SM-vis kits zijn geoptimaliseerd om te sporen mRNAs in verdeelde weefsels of Adherente cellen op dia’s. Echter eicellen niet effectief voldoen aan dia’s en sommige reagentia in de kit waren te hard resulterend in lysis van de oöcyt. Om te voorkomen dat deze lysis, verschillende wijzigingen aangebracht aan de vis-kit. In het bijzonder vervangen oöcyt permeabilization en wastafel buffers ontworpen voor de immunofluorescentie van eicellen en embryo’s de merkgebonden buffers. De permeabilization, wast en incubations met sondes en versterker werden uitgevoerd in 6-Wells-platen en eicellen werden op dia’s aan het einde van het protocol gebruik van montage media geplaatst. Deze wijzigingen waren in staat om te overwinnen van de beperkingen van de verkrijgbare kit, in het bijzonder de lysis van de oöcyt. Als u reproducibly en nauwkeurig tellen het aantal mRNAs in individuele eicellen, werd computersoftware gebruikt. Samen, vertegenwoordigt dit protocol een alternatief voor PCR en rangschikken om te vergelijken van de expressie van specifieke afschriften in afzonderlijke cellen.
Introduction
Reverse-transcriptase polymerase-kettingreactie (PCR) is de gouden standaard voor mRNA kwantificatie. Twee tests, digitale PCR (dPCR)1 en kwantitatieve, real time PCR (qPCR)2 worden momenteel gebruikt. Van de twee PCR technieken heeft dPCR grotere gevoeligheid dan qPCR suggereren dat het kan worden gebruikt voor het meten van de mRNA overvloed in afzonderlijke cellen. Echter, in onze handen, dPCR analyse van lage overvloed mRNAs in poules van 5 tot en met 10 eicellen per elk experimentele monster heeft gegevens met lage reproduceerbaarheid en hoge variatie3. Dit is waarschijnlijk te wijten aan de experimentele fout RNA extractie en omgekeerde transcriptie efficiëntie is gekoppeld. RNA sequencing is ook uitgevoerd met behulp van een enkele muis en menselijke eicellen4,5. Deze techniek vereist cDNA amplificatie stappen die nodig zijn voor de bibliotheek-generatie waardoor waarschijnlijk variabiliteit binnen een experimentele groep. Bovendien, lage overvloed afschriften mogelijk niet aantoonbaar. Hoewel sequencing prijzen zijn gedaald in de afgelopen jaren, kan het nog steeds kosten onbetaalbaar vanwege de hoge kosten van bioinformatica analyses. Ten slotte, mRNA lokalisatie is een dynamisch proces met ruimtelijke veranderingen bij te dragen aan eiwit functie6. Daarom zetten we uit te nemen van een techniek die nauwkeurige en reproduceerbare kwantitatieve maatregelen en lokalisatie van individuele mRNAs in enkele eicellen produceren zou.
Vertakte DNA gekoppeld aan fluorescentie in situ hybridisatie versterkt fluorescentie signaal in plaats van uitdeinende RNA/cDNA inschakelen detectie van één mRNAs in afzonderlijke cellen 7,8,9. De test wordt uitgevoerd door middel van een reeks van hybridisatie, versterking (met behulp van vertakte DNA) en fluorescentie labeling stappen om het versterken van de fluorescentie signaal7. De techniek begint met bindende 18 – tot 25-base oligonucleotide sonde-paren die complementair aan een specifieke mRNA3,8,10 zijn. Vijftien tot twintig sonde paren zijn ontworpen voor elke specificiteit transcript te zorgen voor het transcript van de doelgroep. De mRNA specifieke hybridisatie wordt gevolgd door voorversterker en versterker sondes die een vertakte configuratie vormen. Ongeveer binden 400 label fluorophores aan elke versterker, resulterend in een verhoging van de 8000-fold in fluorescentie waardoor detectie van individuele mRNAs (Figuur 1)11.
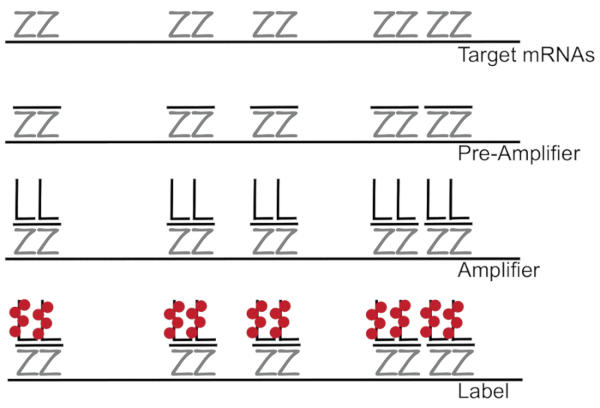
Figuur 1: schematische van de SM-vis protocol. Sequentiële kruising van transcript specifieke sonde, vertakt DNA versterker en fluorophore naar een doel dat mRNA wordt weergegeven. Klik hier voor een grotere versie van dit cijfer.
Eerdere studies met behulp van één molecuul fluorescentie in situ hybridisatie (SM-vis) gelokaliseerd β-actine mRNAs in individuele neuronen12 en humaan papillomavirus DNA in cervicale kanker cel lijnen7. De computersoftware plek vinden en Tracking programma identificeert individuele punctate fluorescent signaal en is met succes gebruikt om het aantal mRNAs in elke cel3,13te kwantificeren.
Op basis van de resultaten van mRNA detectie in neuronen12, veronderstelde wij dat SM-vis zou blijken een nuttig hulpmiddel om te kwantificeren transcript niveaus in lymfkliertest eicellen en embryo’s met inbegrip van lage overvloed mRNAs. Echter, de techniek is geoptimaliseerd voor het gebruik met aanhangend vaste cellen en formaldehyde vaste paraffine ingesloten weefselsecties (FFPE). Eicellen niet kunnen voldoen aan een dia, zelfs wanneer ze zijn bekleed met een Poly-L-lysine. Bovendien zijn ze kwetsbaarder dan somatische cellen en weefselsecties resulterend in lysis van de cel wanneer onderworpen aan sommige van de merkgebonden buffers in commercieel beschikbare kits3. Om deze uitdagingen te overwinnen, waren de eicellen vast en handmatig worden overgedragen tussen de buffers druppels. Bovendien, permeabilization en wastafel buffers in de kits werden vervangen ter vermindering van de lysis van de cel. Vooraf ontworpen sondes worden gekocht naast de vis kit of specifieke afschriften kunnen aangevraagd worden. Elke set merkgebonden sonde is beschikbaar in een van de drie kanalen van de fluorescentie (C1, C2 en C3) om multiplexing mogelijk te maken. In het huidige experiment waren lymfkliertest eicellen dual-gekleurd en gekwantificeerde met behulp van een sonde C2 Nanog en een C3 Pou5f1 -sonde. Deze sondes werden geselecteerd op basis van de gerapporteerde uitdrukking van Nanog en Pou5f1 in eicellen en embryo’s. Aan het einde van de kruising stappen, werden de eicellen in druppels anti-fade montage media voor toepassing op histologische dia’s geplaatst. Confocale afbeeldingen werden gebruikt om het kwantificeren van het aantal punctate fluorescerende signalen die individuele mRNAs vertegenwoordigen. Naast het kwantificeren van de mRNAs, imaging bleek ook de ruimtelijke spreiding van de specifieke mRNA in de cel, zijn welke andere RNA kwantificering methoden niet in staat om te bereiken. Deze techniek bleek te hebben lage variabiliteit binnen een experimentele groep toestaan van het gebruik van kleinere aantallen van eicellen in elke experimentele groep te identificeren van significante verschillen tussen experimentele groepen3.
Protocol
Representative Results
Discussion
Een aantal kleine stappen tijdens het protocol zorgt voor succesvolle fluorescentie en nauwkeurige graven van mRNAs. Ten eerste moet het protocol worden uitgevoerd onmiddellijk na het verzamelen en fixatie van de eicellen. Merk op dat de PVP is toegevoegd aan de 4% paraformaldehyde fixatie buffer om te voorkomen dat eicellen kleven aan elkaar. We vonden dat het noodzakelijk is voor het uitvoeren van het experiment onmiddellijk na het verzamelen en fixatie van de eicellen is. Elke vertraging resulteert in een veel lagere …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wij Dr. Daniel R. Larson bedanken voor zijn genereuze hulp met de installatie en het gebruik van het vinden van de plek en Tracking programma 13 en de technische ondersteuning van de Universiteit van Nebraska-Lincoln microscopie kern voor de beeldvorming van de confocal microscopie. Deze studie vormt een bijdrage van de Universiteit van Nebraska Agricultural Research Division, Lincoln, Nebraska en werd gesteund door UNL Hatch fondsen (NEB-26-206/toetreding nummer-232435 en NEB-26-231/toetreding nummer-1013511).
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

