Tek molekül floresan ın Situ hibridizasyon (SM-balık) kullanmak için fare yumurta miktarını ve Localize mRNA
Summary
Tekrarlanarak mRNA’ların içinde bireysel yumurtalar, tek molekül RNA Floresans in situ olarak sayıları saymak için hibridizasyon (RNA-balık) yapışık olmayan hücreler için optimize edildi. Yumurtalar toplanmıştır, transkript belirli probları ile melezleşmiştir ve bir görüntü miktar yazılımı kullanarak sayılabilir.
Abstract
Geçerli yöntemleri düzenli olarak yumurta ve embriyo mRNA ölçmek için kullanılan dijital ters transkripsiyon Polimeraz zincir reaksiyonu (dPCR), nicel, gerçek zamanlı RT-PCR (RT-qPCR) ve RNA sıralama içerir. Bu tekniklerin bir tek yumurta veya embriyo kullanılarak gerçekleştirildiğinde, düşük-kopya mRNA’ların güvenilir bir şekilde tespit edilmedi. Bu sorunu çözmek için yumurta veya embriyo birlikte analiz için havuza; Ancak, bu kez yüksek değişkenlik örnekleri arasında yol açar. Bu protokol için Floresans in situ hibridizasyon (balık) dallı DNA kimya kullanarak içinde nasıl kullanılacağını açıklar. Bu teknik mRNA’ların tek tek hücreleri içinde kayma desenini tanımlar. Teknik nokta bulma ve izleme bilgisayar yazılım ile birleştiğinde, mRNA’ların Hücre bolluğu da sayısal. Bu tekniği kullanarak, bir deney grubu içinde azaltılmış değişkenlik var ve daha az sayıda yumurta ve embriyo deneysel grupları arasında önemli farklılıklar tespit etmek gerekir. Piyasada bulunan dallı DNA SM-kesitli dokularda veya slaytlardaki yapışık hücreleri mRNA’ların algılamak için optimize balık kitleri. Ancak, yumurta etkili slaytlara uymayan ve bazı reaktif kitinde oosit lizis kaynaklanan çok sert. Bu lizis önlemek için birçok değişikliği balık seti için yapılmıştır. Özellikle, yumurta permeabilization ve yıkama arabellekleri yumurta ve embriyo ayirt için tasarlanmış özel arabellekleri yerine. 6-iyi levha permeabilization, yıkama ve incubations sonda ve amplifikatör ile gerçekleştirilen ve yumurta montaj medya iletişim kuralı’nı sonundaki slaytlarda yerleştirildi. Bu değişiklikler ticari olarak mevcut kit özellikle, oosit lizis sınırlamaları aşmak başardık. Doğru ve tekrarlanarak mRNA’ların tek tek yumurta içinde saymak için bilgisayar yazılımı kullanılmıştır. Birlikte, bu iletişim kuralını PCR ve sıralamanın Tek Kişilik hücrelerde belirli transkript ifadesi karşılaştırmak için bir alternatif temsil eder.
Introduction
Ters transkriptaz Polimeraz zincir tepkimesi (PCR) mRNA Nefelometri için altın standart olmuştur. İki deneyleri, dijital PCR (dPCR)1 ve nicel, real time PCR (qPCR)2 şu anda kullanılır. İki PCR teknikleri, dPCR qPCR tek hücrelerinde mRNA bereket ölçmek için kullanılabilir olduğu ima daha büyük duyarlılığa sahiptir. Ancak, bizim elimizde, deneysel her örnek başına 5-10 yumurta havuzlarda düşük bereket mRNA’ların analizini dPCR veri düşük tekrarlanabilirlik ve yüksek varyasyon3ile üretti. Bu RNA ayıklama ve ters transkripsiyon verimliliği ile ilgili deneysel hata nedeniyle muhtemeldir. RNA sıralama aynı zamanda tek bir fare ve insan yumurta4,5kullanılarak yapılmıştır. Bu teknik büyük olasılıkla bir deney grubu içinde değişkenlik artar Kütüphane üretimi için gerekli cDNA amplifikasyon adımları gerektirir. Ayrıca, düşük bereket transkript tespit olmayabilir. Her ne kadar sıralama fiyatlar son birkaç yıldır gitti, hala maliyet engelleyici Biyoinformatik analizi yüksek maliyet nedeniyle olabilir. Son olarak, mRNA yerelleştirme protein işlevi6için katkıda kayma değişikliklerle dinamik bir süreçtir. Bu nedenle, doğru ve tekrarlanabilir nicel ölçüler ve tek yumurta içinde bireysel mRNA’ların yerelleştirilmesini üretecektir bir teknik benimsemeye Vadisi’ni gezmeye gidiyoruz.
Dallı DNA Floresans in situ hibridizasyon için birleştiğinde yükselterek RNA/cDNA tek tek hücreler 7,8,9tek mRNA’ların etkinleştirme tespiti yerine Floresans sinyali güçlendirir. Tahlil hibridizasyon, amplifikasyon (dallı DNA’yı kullanarak) ve floresan sinyal7yükseltmek için adımları etiketleme floresan bir dizi aracılığıyla gerçekleştirilir. Teknik bir belirli mRNA3,8,10‘ a tamamlayıcı 18 – 25 Bankası Oligonükleotid sonda çiftlerinin bağlama ile başlar. On beş-yirmi sonda çiftleri her transkript sağlanması özgüllük hedef transkript için tasarlanmıştır. MRNA özgü hibridizasyon dallı bir yapılandırma oluşturan ön amfi ve amplifikatör probları tarafından takip edilir. Yaklaşık olarak, 400 etiket fluorophores 8000-fold artış bireysel mRNA (şekil 1)11olarak algılanmasını sağlayan Floresans sonuçlanan her amplifikatör bağlamak.
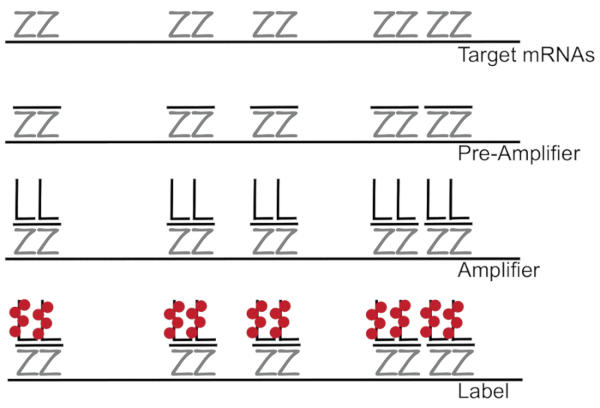
Şekil 1: SM-balık protokolünün şematik. Transkript belirli sonda, sıralı hibridizasyon dallı DNA amplifikatör ve mRNA gösterilir bir hedefe fluorophore. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Önceki çalışmalarda in situ hibridizasyon (SM-balık) Lokalize β-aktin mRNA bireysel nöronlar12 ve servikal kanser HPV DNA molekülünü Floresans kullanarak satırları7hücre. Bilgisayar yazılımı nokta bulma ve izleme programı bireysel punctate floresan sinyal tanımlar ve başarıyla mRNA’ların her hücre3,13sayısını ölçmek için kullanılır.
SM-balık transkript düzeyleri fare yumurta ve embriyo düşük bereket mRNA’ların da dahil olmak üzere quantitate için yararlı bir araç olduğunu kanıtlayacak ki nöronlar12mRNA tespiti sonuçlarına dayanarak, biz onaylanmadığına karar. Ancak, teknik yapisan sabit hücreleri ile kullanım için optimize edilmiş ve parafin sabit formaldehit (FFPE) doku bölümleri gömülü. Bile Poly-L-lizin ile kaplı olan yumurtalar bir slayt için uygun değil. Ayrıca, onlar somatik hücre ve doku bölümleri ne zaman piyasada kitleri3özel arabellekleri bazıları tabi hücre lizis sonuçlanan daha kırılgan. Bu zorlukları aşmak için yumurta sabit ve el ile arabellekleri damla arasında transfer. Ayrıca, permeabilization ve yıkama arabellekleri kitleri hücre lizis azaltmak için değiştirildi. Önceden tasarlanmış probları balık seti ile birlikte satın alınan veya belirli transkript talep edilebilir. Her özel yoklama kümesi birinde üç Floresans kanal (C1, C2 ve C3) çoğullama için izin kullanılabilir. Geçerli deneyde, çift lekeli ve quantified C2 MicroRNAs sonda ve C3 Pou5f1 sonda kullanarak fare yumurta vardı. Bu sonda bildirilen ifadede yumurta ve embriyo MicroRNAs ve Pou5f1 göre seçildi. Hibridizasyon adımlar sonucunda, yumurta damla uygulama histolojik slaytlar için anti-fade montaj ortamının yerleştirildi. Confocal görüntüleri bireysel mRNA’ların temsil punctate floresan sinyallerini sayısını ölçmek için kullanılmıştır. MRNA’ların, görüntüleme Ayrıca belirli mRNA kayma dağılımını hücre içinde gösterdi. miktarının yanı sıra hangi diğer RNA miktar yöntemleri elde edemiyoruz. Bu teknik deneysel grupları3arasındaki önemli farkları belirlemek için her deney grubu içinde yumurta daha küçük sayıda kullanımına izin veren bir deney grubu içinde düşük değişkenlik var kanıtladı.
Protocol
Representative Results
Discussion
Protokol sırasında küçük adımlar bir dizi başarılı Floresans ve mRNA’ların doğru sayar sağlayacaktır. İlk olarak, protokol koleksiyonu ve yumurta fiksasyonu hemen sonra gerçekleştirilmelidir. PVP yumurta birbirine yapışmasını önlemek için %4 paraformaldehyde fiksaj arabelleğin eklenir unutmayın. Biz deneme koleksiyonu ve yumurta fiksasyonu hemen sonra gerçekleştirmek gerekli bulundu. Herhangi bir gecikme transkript undercounting neden olacak bir çok daha düşük Floresans sinyal sonuçlanır. …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dr Daniel R. Larson yükleme ve 13 nokta bulma ve izleme programı kullanımı ve Nebraska Üniversitesi Lincoln mikroskobu çekirdek confocal mikroskobu görüntüleme için teknik destek ile cömert onun yardım için teşekkür. Bu çalışmada bir katkı Nebraska tarımsal araştırma bölümü, Lincoln, Nebraska Üniversitesi’nin temsil eder ve UNL Hatch fonlar (NF’leri-26-206/katılım numarası-232435 ve NF’leri-26-231/katılım numarası-1013511) tarafından desteklenmiştir.
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

