Использование одной молекулы флуоресцентные в гибридизации Situ (SM-рыбы) для Quantify и Localize mRNAs в мышиных ооцитов
Summary
Можно воспроизвести подсчитать количество мРНК в отдельных ооцитов, одной молекулы РНК флуоресцирования на месте гибридизации (РНК-рыбы) был оптимизирован для non сторонник клеток. Ооциты были собраны, гибридизированных конкретных датчиками Стенограмма и количественно с помощью программного обеспечения количественной оценки изображения.
Abstract
Нынешние методы, обычно используемые для количественного определения мРНК в яйцеклеток и эмбрионов включают цифровые реверс транскрипция полимеразной цепной реакции (dPCR), количественные, реальном времени RT-PCR (RT-ПЦР) и РНК последовательности. Когда эти методы выполняются с помощью одной яйцеклетки или эмбриона, низкий копия mRNAs надежно не обнаруживаются. Для преодоления этой проблемы, яйцеклеток или эмбрионов могут быть объединены вместе для анализа; Однако это часто приводит к высокой изменчивости среди образцов. В этом протоколе мы описывают использование флюоресценции в гибридизации situ (рыба) с помощью разветвленной ДНК химия. Этот метод определяет пространственное распределение mRNAs в отдельных клетках. Когда техника связан с местом нахождения и отслеживания программного обеспечения, обилие mRNAs в ячейке также может быть определена количественно. Используя эту технику, есть снижение изменчивости в экспериментальной группе и меньше яйцеклеток и эмбрионов обязаны обнаружить существенные различия между экспериментальной группы. Коммерчески доступные разветвленной ДНК SM-рыба наборы были оптимизированы для выявления mRNAs в секционного тканей или сторонником клетки на слайдах. Однако ооциты не эффективно придерживаться слайды и некоторые реагенты из комплекта были слишком тяжелыми, результате лизис ооцитов. Для предотвращения этого лизиса, некоторые изменения были внесены комплектом рыбу. В частности ооцитов permeabilization и мыть буферы предназначены для иммунофлюоресценции яйцеклеток и эмбрионов вместо несвободных буферов. Permeabilization, моет и инкубаций с датчики и усилители выполнены в 6-ну пластины и ооциты были размещены на слайды в конце протокола, с помощью установки средств массовой информации. Эти изменения были в состоянии преодолеть ограничения, коммерчески доступных комплект, в частности, лизис ооцитов. Для герметизации и точно подсчитать количество мРНК в отдельных ооцитов, компьютерного программного обеспечения был использован. Вместе этот протокол представляет собой альтернативу ПЦР и последовательности для сравнения выражения конкретных протоколов в одиночных клетках.
Introduction
Обратной транскриптазы полимеразной цепной реакции (ПЦР) был золотым стандартом для мРНК quantitation. В настоящее время используются два анализов, цифровой ПЦР (dPCR)1 и количественных, реальное время ПЦР (ПЦР)2 . Из двух методов ПЦР dPCR имеет большую чувствительность, чем ПЦР предполагая, что он может использоваться для измерения mRNA изобилия в одиночных клетках. Однако в наших руках, dPCR анализ низкого залегания mRNAs в бассейнах 5 до 10 яйцеклеток за каждый экспериментальный образец выпустила данных с низкой воспроизводимостью и высокий вариант3. Это, вероятно, из-за экспериментальной ошибки, связанной с РНК добыча и эффективности обратной транскрипции. РНК последовательности также была выполнена с помощью мыши и человеческих яйцеклеток4,5. Этот метод требует cDNA амплификации шаги, необходимые для библиотеки поколения, которое вероятно увеличивает изменчивость в экспериментальной группе. Кроме того низкие изобилии стенограммы не может быть обнаружено. Хотя последовательности цены снизились за последние несколько лет, это может быть дорого из-за высокой стоимости биоинформатики анализов. Наконец мРНК Локализация — это динамичный процесс с пространственных изменений, способствующих белков функции6. Таким образом мы намереваемся принять метод, который будет производить точные и воспроизводимые количественные показатели и локализации индивидуальных mRNAs в одно ооцитов.
Разветвленный ДНК, в сочетании с флуоресценции в гибридизации situ усиливает сигнал флуоресценции, вместо того, чтобы усилительных благоприятных обнаружение РНК/кДНК одной мРНК в отдельных клетках 7,8,9. Генотипирования через серию гибридизации, амплификация (с помощью разветвленной ДНК) и флуоресценции маркировки шаги для того, чтобы усилить флуоресценции сигнала7. Этот метод начинается с привязки 18 – 25-base олигонуклеотида зонд пар, которые дополняют конкретные мРНК3,8,10. Пятнадцати до двадцати зонд пары предназначены для обеспечение специфику каждого Стенограмма для целевого транскрипт. МРНК конкретных гибридизации следуют предварительный усилитель и усилитель зондами, которые формируют разветвленную конфигурации. Приблизительно 400 лейбл флуорофоров привязку каждого усилителя, что привело к увеличению 8000-fold в флуоресцировании, позволяя обнаружения индивидуальных mRNAs (рис. 1)11.
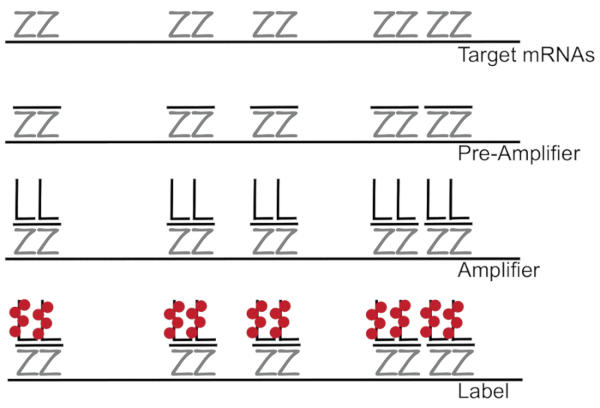
Рисунок 1: Схема протокола SM-рыба. Последовательные гибридизации Стенограмма конкретного ПЭП, разветвленной ДНК усилитель и Флюорофор к цели, которую показана mRNA. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Предыдущие исследования с использованием одной молекулы флуоресценции в situ гибридизация (SM-рыбы) локализованные β-актина mRNAs в12 отдельных нейронов и ДНК вируса папилломы человека в рак шейки матки клеток линии7. Компьютерное программное обеспечение, месте нахождения и отслеживания Программа идентифицирует отдельных пунктата флуоресцентного сигнала и успешно используется для количественного определения количество мРНК в каждой ячейке3,13.
Основываясь на результаты обнаружения мРНК в нейроны12, мы предположили, что SM-рыб окажется полезным инструментом для quantitate Стенограмма уровней в мышиных яйцеклеток и эмбрионов, включая низкие изобилии мРНК. Однако техника оптимизирована для использования с фиксированной адэрентных клеток и формальдегида фиксированной парафин встроенных разделов ткани (FFPE). Ооциты не может присоединиться к слайду, даже когда они покрыты с поли-L-лизин. Кроме того они являются более хрупкие, чем соматических клетках и результате лизис клеток когда подвергаются некоторые несвободные буферов в коммерчески доступные комплекты3разделах ткани. Для преодоления этих проблем, ооциты фиксировали и вручную переведен между капли буферов. Кроме того permeabilization и мыть буферов в наборы были заменены уменьшить lysis клетки. Встроенные датчики приобретаются вместе с комплектом рыбу или конкретных протоколов могут запрашиваться. Каждого собственности зонда набор доступен в одном из трех каналов флуоресцирования (C1, C2 и C3) для мультиплексирования. В ходе текущего эксперимента мышиных ооциты были двойной окрашенных и количественных, используя зонд Nanog C2 и C3 Pou5f1 зонда. Эти зонды были отобраны на основе сообщенных выражения Nanog и Pou5f1 яйцеклеток и эмбрионов. В заключение гибридизации шаги ооциты были помещены в капли анти затухания монтажа СМИ для приложения гистологические слайды. Конфокальный изображения были использованы для количественного определения количество пунктата флуоресцентные сигналов, которые представляют индивидуальных mRNAs. Помимо количественного мРНК, изображений также показал пространственное распределение конкретные мРНК в ячейке, какие другие методы количественного определения РНК не в состоянии достичь. Этот метод оказался низким изменчивости в пределах экспериментальной группы, что позволяет использовать меньшее количество яйцеклеток в каждой экспериментальной группе выявить существенные различия между экспериментальной группы3.
Protocol
Representative Results
Discussion
Ряд мелких шагов во время протокол обеспечит успешное флуоресценции и точное количество мРНК. Во-первых протокол должны быть выполнены сразу после сбора и фиксации яйцеклеток. Обратите внимание, что PVP добавляется в буфер фиксации параформальдегида 4% для предотвращения ооциты от прил?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы благодарим д-р Даниэль р. Ларсон за его щедрую помощь с установки и использования местом поиска и отслеживания программы 13 и технической поддержке Университета Небраска Линкольн микроскопии Core для воображения confocal микроскопии. Это исследование представляет собой вклад Университета Небраска сельскохозяйственных исследований отдела, Линкольн, штат Небраска и была поддержана UNL Люк фондов (NEB-26-206/присоединения номер-232435 и НЭБ-26-231/присоединения номер-1013511).
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

