Bruk av ett molekyl fluorescerende i Situ hybridisering (SM-fisk) å kvantifisere og Localize mRNAs i Murine Oocytes
Summary
Du reproduserbar teller antallet mRNAs i individuelle oocytes, ett molekyl RNA fluorescens i situ var hybridisering (RNA-fisk) optimalisert for ikke-tilhenger celler. Oocytes var samlet, hybridiserte med transkripsjon bestemt sonder og kvantifisert bruker en kvantifisering programvare.
Abstract
Gjeldende metoder rutinemessig brukes å kvantifisere mRNA i oocytes og embryo inkluderer digitale omvendt-transkripsjon polymerasekjedereaksjons (dPCR), kvantitativ, real-time RT-PCR (RT-qPCR) og RNA sekvensering. Når disse teknikkene utføres ved hjelp av en enkelt oocytter og embryo, gjenkjennes lav-kopi mRNAs pålitelig ikke. Du løser dette problemet, kan oocytes eller embryo samordnes sammen for analyse; men fører dette ofte til høy variasjon blant prøver. I denne protokollen beskriver vi bruk av fluorescens i situ hybridisering (fisk) bruker forgrenet DNA kjemi. Denne teknikken identifiserer romlige mønster av mRNAs i enkeltceller. Når teknikken er kombinert med sted å finne og sporingsprogramvare, kan også overflod av mRNAs i cellen kvantifiseres. Bruker denne teknikken, det er redusert variasjon innen en forsøksgruppen og færre oocytes og embryo kreves for å gjenkjenne betydelige forskjeller mellom eksperimentelle grupper. Kommersielt tilgjengelige forgrenet-DNA SM-fisk kits er optimalisert for å oppdage mRNAs i valgte vev eller tilhenger celler i lysbilder. Men oocytes effektivt overholder ikke lysbilder og noen reagenser i kit var for harde resulterer i oocyte lysis. For å forhindre denne lyse, ble flere endringer gjort fisk Kit. Spesielt erstattet oocyte permeabilization og vask buffere designet for immunofluorescence av oocytes og embryo de proprietære bufferne. Den permeabilization, vasker og incubations med sonder og forsterker ble utført i 6-vel plater og oocytes ble plassert på lysbilder på slutten av protokollen bruker monterer medier. Disse endringene kunne overvinne begrensningene til kommersielt tilgjengelige settet, spesielt oocyte lysis. For å nøyaktig og reproduserbar telle antall mRNAs i individuelle oocytes, ble programvare brukt. Sammen, representerer denne protokollen et alternativ til PCR og sekvensering sammenligne uttrykk for bestemte utskrifter i enkeltceller.
Introduction
Revers transkriptase polymerasekjedereaksjons (PCR) har vært gullstandarden for mRNA kvantifisering. To analyser, digital PCR (dPCR)1 og kvantitativ, virkelig tid PCR (qPCR)2 brukes for øyeblikket. Av de to PCR teknikkene har dPCR større følsomhet enn qPCR antyder at det kan brukes til å måle mRNA overflod i enkeltceller. Men i våre hender, har dPCR analyse av lav overflod mRNAs i av 5 til 10 oocytes per hvert eksperimentelle utvalg produsert data med lav reproduserbarhet og høy variant3. Dette er sannsynligvis på grunn av eksperimentelle feil forbundet med RNA utvinning og omvendt transkripsjon effektivitet. RNA sekvensering er også utført med et enkelt museklikk og menneskelig oocytes4,5. Denne teknikken krever cDNA forsterkning fremgangsmåtene for biblioteket generasjon som sikkert øker variasjonen i en forsøksgruppen. Videre kan lav overflod utskrifter ikke være synlig. Selv om sekvensering prisene har gått ned de siste årene, kan det likevel være kostnadseffektivt uoverkommelige på grunn av de høye kostnadene av bioinformatikk analyser. Endelig er mRNA lokalisering en dynamisk prosess med romlig endringer bidra til protein funksjon6. Derfor setter vi opp for vedta en teknikk som vil produsere nøyaktig og reproduserbar kvantitative tiltak og lokalisering av personlige mRNAs i én oocytes.
Forgrenede DNA koblet til fluorescens i situ hybridisering forsterker fluorescens signal i stedet for å forsterke RNA/cDNA aktivere gjenkjenning av ett mRNAs i enkeltceller 7,8,9. Analysen utføres gjennom en rekke hybridisering, forsterkning (med forgrenet DNA) og fluorescens merking skritt for å forsterke fluorescens signal7. Teknikken begynner med binding av 18 – til 25-base oligonucleotide sonde par som er komplementære til en bestemt mRNA3,8,10. Femten til tjue sonde parene er designet for hver utskrift sikre spesifisitet for målet transkripsjon. MRNA-spesifikke hybridization etterfølges av pre forsterker og forsterker sonder som danner en forgrenet konfigurasjon. Ca, 400 etiketten fluorophores binder seg til hver forsterker, resulterer i en 8000-fold økning i fluorescens tillater påvisning av individuelle mRNAs (figur 1)11.
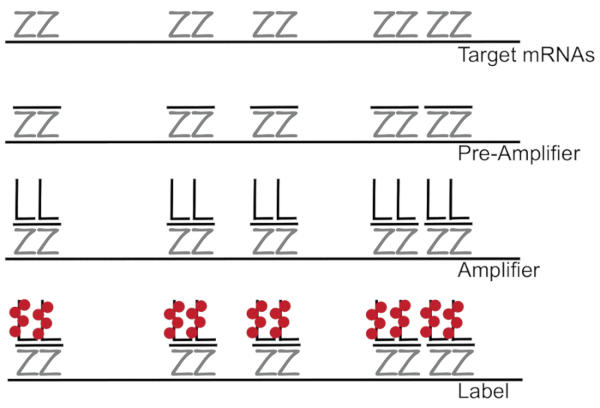
Figur 1: skjematisk av SM-fisk protokollen. Sekvensiell blanding av transkripsjon bestemt sonde, forgrenet DNA forsterker og fluorophore til et mål mRNA vises. Klikk her for å se en større versjon av dette tallet.
Tidligere studier med ett molekyl fluorescens i situ hybridisering (SM-fisk) lokalisert β-utgangen mRNAs i individuelle neurons12 og humant papillomavirus DNA i livmorhalskreft celle linjer7. Dataprogramvaren sted å finne og sporing programmet identifiserer individuelle vises punctate fluorescerende signal og har blitt brukt å tallfeste antallet mRNAs i hver celle3,13.
Basert på resultatene av mRNA gjenkjenning i neurons12, hypotese vi at SM-fisk ville bevise en nyttig verktøyet å quantitate transkripsjon nivåer i murine oocytes og embryo inkludert lav overflod mRNAs. Men teknikken er optimalisert for bruk med tilhenger fast celler og formaldehyd fast parafin innebygd (FFPE) vev deler. Oocytes kan ikke følge et lysbilde, selv når de er belagt med Poly-L-lysine. Videre, de er mer sårbar enn somatiske celler og vev inndelinger som resulterer i cellen lysis når utsatt for noen av proprietære bufferne i kommersielt tilgjengelig kits3. For å overvinne disse utfordringene, oocytes fast og manuelt overføres mellom dråper bufferne. Videre erstattet permeabilization og vask buffere i settene for å redusere celle lysis. Forhåndsutformede sonder kjøpes sammen med fisk kit eller bestemte utskrifter kan forespørres. Hver proprietære sonde er tilgjengelig i en av tre fluorescens kanaler (C1, C2 og C3) for å tillate multipleksing. I gjeldende eksperimentet var murine oocytes dual-farget og kvantifisert ved hjelp av en C2 Nanog sonde og en C3 Pou5f1 sonde. Disse sonder ble valgt basert på rapporterte uttrykk for Nanog og Pou5f1 i oocytes og embryo. Ved avslutningen av hybridisering trinnene plassert oocytes i dråper av anti-fade monterer medier skal histologiske lysbilder. AC confocal bilder ble brukt om å kvantifisere antall vises punctate fluorescerende signaler som representerer personlige mRNAs. Kvantifisere mRNAs, bildebehandling viste også den romlige fordelingen av den spesifikke mRNA i cellen, er hvilke andre RNA kvantifisering metoder ikke å oppnå. Denne teknikken vist seg for å ha lav variasjon innen en forsøksgruppen tillater bruk av mindre antall oocytes i hver forsøksgruppen å identifisere betydelige forskjeller mellom eksperimentelle grupper3.
Protocol
Representative Results
Discussion
En rekke mindre trinn under protokollen sikrer vellykket fluorescens og nøyaktig antall mRNAs. Protokollen må først utføres umiddelbart etter innsamling og fiksering av oocytes. Merk at PVP legges til 4% paraformaldehyde fiksering bufferen slik at oocytes henger sammen. Vi fant at det er nødvendig å utføre eksperimentet umiddelbart etter innsamling og fiksering av oocytes. Forsinkelser resulterer i en mye lavere fluorescens signal som vil føre til undercounting av transkripsjoner. Dette skyldes delvis RNA degrade…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi takker Dr. Daniel R. Larson for hans sjenerøs hjelp med installasjon og bruk av sted å finne og sporing Program 13 og teknisk støtte fra University of Nebraska Lincoln mikroskopi kjernen for AC confocal mikroskopi avbilding. Denne studien representerer et bidrag på University of Nebraska Agricultural Research Division, Lincoln, Nebraska og ble støttet av UNL Luke midler (NB-26-206/tiltredelse-232435 og NB-26-231/tiltredelse-1013511).
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

