Bereiding van DNA verknoopt polyacrylamide hydrogels
Summary
Ons laboratorium heeft DNA verknoopt polyacrylamide hydrogels, een dynamisch hydrogel systeem ontwikkeld om de effecten van modulerende weefsels stijfheid op celfunctie beter te begrijpen. Hier bieden we schema's, beschrijvingen en protocollen om deze hydrogels bereiden.
Abstract
Mechanobiology is een opkomende wetenschappelijk gebied dat de kritische rol van fysieke signalen in regisseren cel morfologie en functie-adressen. Bijvoorbeeld, het effect weefsel elasticiteit celfunctie is een belangrijk gebied van Mechanobiology onderzoek omdat weefsel stijfheid moduleert met de ziekte, ontwikkeling, en schade. Static weefsel nabootsen materialen, of materialen die niet kan wijzigen stijfheid wanneer cellen worden uitgeplaat, worden voornamelijk gebruikt om de effecten weefsel stijfheid op celfuncties te onderzoeken. Terwijl verzameld van statische studies informatie waardevol is, deze studies zijn niet indicatief voor het dynamische karakter van de cellulaire micro-omgeving in vivo. Om de effecten van dynamische stijfheid op celfunctie beter tegemoet ontwikkelden we een DNA verknoopt polyacrylamide hydrogel systeem (DNA gels). In tegenstelling tot andere dynamische substraten, DNA gels hebben de mogelijkheid om te verlagen of te verhogen van de stijfheid na fabricage zonder stimuli. DNA gels bestaan uit DNA crosslinkten die worden gepolymeriseerd tot een polyacrylamide hoofdketen. Toevoegen en verwijderen verknopingen via levering van enkelstrengs DNA mogelijk maakt temporele, ruimtelijke en reversibele controle gel elasticiteit. We hebben laten zien in de vorige verslagen die de dynamische modulatie van DNA-gel elasticiteit beïnvloedt fibroblast en neuron gedrag. In dit verslag en video, bieden we een schema dat de DNA-gel verknoping mechanismen en stap-voor-stap instructies beschrijft over het preparaat DNA gels.
Introduction
Statische en dynamische substraten twee categorieën biomaterialen die zijn ontwikkeld om de effecten van weefsel elasticiteit of stijfheid op celfunctie bestuderen. Statische substraten kunnen hun fysische eigenschappen veranderen nadat ze zijn vervaardigd en / of wanneer cellen worden uitgeplaat. Polyacrylamide (PA) gels waren de eerste tweedimensionale, statische substraten die werden gesynthetiseerd voor Mechanobiology onderzoeken 5,17. PA gels zijn gemakkelijk te bereiden, goedkoop, veelzijdig en kan worden vervaardigd met een groot aantal elastische moduli. Hoewel deze technische voordelen maken PA gels een algemeen toegepaste substraat, statische substraten zijn niet indicatief voor het dynamische karakter van de extracellulaire matrix (ECM) en de omliggende cellulaire omgeving in vivo. Bijvoorbeeld, de ECM stijfheid veranderingen ondergaat als gevolg van een verwonding, ontwikkeling of ziekte. Dynamische substraten zijn daarom de voorkeur als weefsel nabootsen substraat modellen in Mechanobiology studies <sup> 22,24,25.
Talrijke synthetisch zijn natuurlijke, tweedimensionale, driedimensionaal, statische en dynamische biomaterialen ontwikkeld om weefsel stijfheid 1,3,6,16,23,26 nabootsen. Sommige dynamische substraten vereisen hitte, UV-, elektrische stroom, ionen en de pH veranderingen in hun mechanische eigenschappen 2,4,7,8,12,15,16 veranderen, maar deze prikkels kunnen bio-applicatie van de hydrogel te beperken. DNA verknoopt polyacrylamide hydrogels (DNA gels) dynamisch tweedimensionale elastische substraten. DNA crosslinks zorgen voor temporele, ruimtelijke en reversibele modulatie van DNA gel stijfheid door toevoeging van enkelstrengs DNA (ssDNA) media of buffer 9-11,13,14,18,21. Anders dan de bovengenoemde dynamische gels waar stimuli worden toegepast modulatie van elasticiteit, de DNA gels vertrouwen op de diffusie van toegepaste ssDNA voor de wijziging van elasticiteit. Daarom is de bovenste gel oppervlak, waar de cellen worden gekweekt, is het eerste gebied gemoduleerde omdat het tarief of elasticiteit modulatie afhankelijk van de gel dikte.
DNA gels zijn vergelijkbaar met hun tegenhangers PA gel in dat ze een polyacrylamide hoofdketen, maar de bis-acrylamide verknopingen vervangen verknopingen samengesteld DNA (figuur 1). Twee ssDNAs (SA1 en SA2) hybridiseren met een verharder streng (L2) te maken van de DNA-verknopingen van de gel. SA1 en SA2 hebben verschillende sequenties die beide bevatten een acrydite modificatie aan het 5'-end voor een effectieve integratie in de PA-netwerk. Voor de bereiding van de gels, SA1 en SA2 worden afzonderlijk gepolymeriseerd tot een PA hoofdketen en vervolgens het gepolymeriseerde SA1 en SA2 samen gemengd. L2, de crosslinker, wordt toegevoegd aan de SA1 en SA2 mengsel. De L2 basenvolgorde is complementair aan zowel SA1 en SA2 sequenties en L2 hybridiseert met SA1 plus SA2 aan het DNA crosslinks vormen. Eerste, DNA gel elasticiteit wordt bepaald door zowel L2 concentraties en verknoping (tabellen 1 </strong> en 2). DNA gels die gelijk stoichiometrische hoeveelheden L2, SA1 en SA2 zijn de stijfste gels omdat SA1 en SA2 zijn 100% verknoopt door L2 (aangeduid als 100% gels). Lagere concentraties L2 resulteren in een lager percentage DNA verknoping en derhalve zachter DNA gels. Gels zo laag als 50% verknoopt (aangeduid als 50% gels) zijn geconstrueerd 9-11.
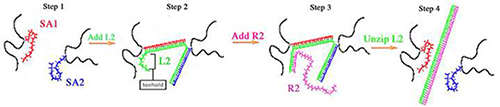
Figuur 1 DNA gel verknoping en uncrosslinking schematische 9-11,13,14,18,21 Stap 1:. SA1 (rood) en SA2 (blauw) zijn individueel gepolymeriseerd tot een polyacrylamide backbone (zwart). Na polymerisatie worden SA1 en SA2 gepolymeriseerde oplossingen met elkaar vermengd. Stap 2: L2 (groen) wordt toegevoegd en hybridiseert met SA1 plus SA2 de verknopingen van de gel. Stap 3: R2 hybridiseert met thij steunpunt van L2. Stap 4: steunpunt hybridisatie van R2 stuwt het openritsen van L2 uit SA1 en SA2.
In tegenstelling tot de PA gels, kan DNA-gels verstijven en te verzachten na de synthese. Daarom kunnen cellen gekweekt op DNA gels onderworpen aan dynamische stijfheid veranderingen. Om cel-hechtende gels verstevigen, L2 kunnen worden toegevoegd aan het kweekmedium van laag percentage gels het percentage verknopingen verhogen. Om cel-hechtende gels verzachten, L2 kan worden verwijderd om het percentage verknopingen 10,13,21 verminderen. L2 heeft een extra steunpunt sequentie aan het 3'einde om L2 te uncrosslink van SA1 en SA2 (tabel 1). Verwijdering van L2 wordt bereikt door hybridisatie van een omkering streng genoemd R2. R2 is een aanvulling op de volledige lengte van L2 en hybridiseert eerst met de L2 houvast. Toehold hybridisatie stuwt het openritsen van L2 uit SA1 en SA2, die de crosslink elimineert en vermindert de gel stijfheid.
In dit rapport envideo, stap-voor-stap instructies worden verstrekt voor het opstellen van verstijving en verzachtende DNA gels. Terwijl 100% en 80% gelpreparaten worden beschreven, kan dit protocol worden aangepast aan DNA gels van andere eerste en laatste verknoopt percentages creëren. In het algemeen zijn 100% en 80% gels bereid, geïmmobiliseerd op glazen dekglaasjes, gefunctionaliseerde en geënt met cellen. L2 wordt toegevoegd aan het medium van 80% gels en R2 wordt toegevoegd aan het medium 100% gels, 48 uur na uitplaten. De toevoeging van L2 tot media verstijft 80% gels tot 100% verknoopt, terwijl de toevoeging van R2 naar media verzacht 100% gels tot 80% verknoopt. Verstijfde gels worden aangeduid als 80 → 100% gels en zacht gels worden aangeduid als 100 → 80% gelen in de tekst. Voor control of statische gels, ssDNA bestaande uit Ts of As is afgeleverd andere set 100% en 80% gels. Na minimaal twee dagen na elasticiteit modulatie, kunnen cellen worden verwerkt en geanalyseerd.
<table border="0" cellpadding="0" cellspacing="0" fo:keep-together.within-page = "always">Tabel 1 Basis sequenties voor ssDNA 9-11,13,14,18,21. Cellulaire en mechanische studies hebben verscheidene d benutifferent crosslink ontwerpen DNA gels met verschillende statische en dynamische mechanische eigenschappen te genereren. De gemoduleerd verknoping ontwerpparameters zijn basensequentie sequentie en lengte of verknopen lengte. Vet en cursief lettertype illustreren baseparing tussen SA1 en L2 en tussen respectievelijk SA2 en L2.
| Ontwerp | ||||||||
| 1 | 2 | 3 | ||||||
| Acrylamide Concentratie (%) | 10 | 10 | 10 | 4 | ||||
| SA1 plus SA2 gehybridiseerd met L2 (% verknoopt) | 50 | 80 | 100 | 50 | 80 | 100 | 100 | 100 |
| <strong> Elasticiteit (kPa, gemiddelde ± SEM) | 6,6 ± 0,6 | 17,1 ± 0,8 | 29.8 ± 2.5 | 5.85 ± 0.62 | 12.67 ± 1.33 | 22.88 ± 2.77 | 25.2 ± 0.5 | 10,4 ± 0,6 |
Tabel 2 Young's modulus (E) van DNA gels 9-11,13,14,18,21. Acrylamide concentratie crosslink percentage en verknoping lengte kan worden gemoduleerd DNA gels. Designs 1, 2 en 3 hebben 20, 28 en 40 bp verknopen lengten respectievelijk. 100% gels voor alle ontwerpen hebben vergelijkbare moduli aangeeft crosslink lengte heeft geen invloed gel elasticiteit. Echter, variaties in concentratie acrylamide veranderen DNA gel elasticiteit.
Protocol
Representative Results
Discussion
Het vermogen van DNA gels zacht of verstijven voor en na celadhesie zijn ze ideaal model voor de rol van dynamische stijfheid weefsels op celfunctie bestuderen. Alle drie ontwerpen zijn gebruikt in mechanische en biologische studies. Echter, alle drie ontwerpen hebben vergelijkbare elasticiteit bij verschillende crosslink percentages vermelding verknoping lengte ofwel geen invloed DNA gel elasticiteit (tabel 2). Daarentegen acrylamide concentratie beïnvloedt elasticiteit. Deze ontwerpen kunnen verschil…
Divulgations
The authors have nothing to disclose.
Acknowledgements
De auteurs willen graag bedanken: Dr Frank Jiang, Dr David Lin, Dr Bernard Yurke en Dr Uday Chippada voor hun bijdrage aan de ontwikkeling van de DNA-gel technologie; Dr Norell Hadzimichalis, Smit Shah, Kimberly Peterman, Robert Arter voor hun opmerkingen en bewerkingen van dit manuscript; financieringsbronnen, waaronder de New Jersey Commissie over Spinal Cord Research (Grant # 07A-019-SCR1, NAL) en New Jersey Instituut voor Neurowetenschappen (MLP); en uitgevers van Tissue Engineering, deel A, voor de toestemming om de figuren 2 en 4 en Biomaterialen herdrukken voor toestemming om Figuur 3 herdrukken.
Materials
| ssDNA | Integrated DNA Technologies (Coralville, Iowa) idtdna.com |
Do not vortex ssDNA. Gentle invert the vial and/or pipette solution to mix. | |
| PBS with calcium and magnesium | Any brand. | ||
| 100X Tris-EDTA buffer (TE buffer) | Sigma-Aldrich (St. Loius, MO) sigmaldrich.com |
T9285 | |
| 10X Tris-Borate-EDTA buffer (TBE buffer) | Sigma-Aldrich (St. Loius, MO) | 93290 | TBE is a reproductive toxin. |
| 40% Acrylamide solution | Fisher Scientific (Pittsburg, PA) | BP14021 | Acrylamide is a toxin. |
| Ammonium persulfate (APS) | Sigma-Aldrich (St. Loius, MO) | A3678 | Prepare a 2% solution in TE buffer. APS is a toxin and irratant. |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich (St. Loius, MO) | T9281 | Prepare a 20% solution in TE buffer. TEMED is flammable, a corrosive, and a toxin. |
| 12-mm diameter round coverglass | Fisher Scientific (Pittsburg, PA) fishersci.com |
12-545-82 | |
| Norland optical adhesive 72 | Norland Products (Cranbury, NJ) norlandprod.com |
NOA72 | |
| 24-well tissue culture plate | Any brand. | ||
| Microcentrifuge tubes | Any brand. | ||
| Sulfo-SANPAH | ProteoChem or Thermo Fisher, (Rockland, IL) proteochem.com or thermofisher.com |
C111 or 22589 | Prepare a 0.315 mg/ml solution in water immediately before use. Dissolve at 37°C and filter sterilze. It is normal to observe undisolved sulfo-SANPAH in the filter. Sulfo-SANPAH is light sensitive and, therefore, the solution should be protect from light until UV exposure. |
| Poly-D-Lysine (PDL) | Sigma-Aldrich (St. Loius, MO) | P6407 | Prepare a 0.2 mg/ml solution in water and filter sterilize. |
| Collagen Type I | Affymetrix (Santa Clara, CA) affymetrix.com |
13813 | Prepare a 0.2 mg/ml solution in 0.2 N acetic acid. Solution needs to remain cold at all times to avoid polymerization. Acetic acid is a flammable, toxic, and corrosive. |
| 22 X 60 cover glass | Fisher Scientific (Pittsburg, PA) | 12-544-G | |
| Positive-displacement pipette | Gilson, Inc (Middletown, WI) gilson.com |
F148504 | |
| Heat block | Fisher Scientific (Pittsburg, PA) | 11-718 | |
| UV light source | Place gels as close as possible to the UV light. UV light can cause skin or eye injury. | ||
| Thermometer | Any brand. | ||
| Nitrogen gas | GTS-Welco (Flemington, NJ) www.praxairmidatlantic.com/ |
NI 5.0UH-R |
References
- Balaban, N. Q., et al. Force and focal adhesion assembly A close relationship studied using elastic micropatterned substrates. Nat Cell Biol. 3 (5), 466-472 (2001).
- Peppas Brannon-Peppas, L., Peppas, N. A. Dynamic and equilibrium swelling behaviour of ph-sensitive hydrogels containing 2-hydroxyethyl methacrylate. Biomaterials. 11 (9), 635-644 (1990).
- Charati, M. B., Ifkovits, J. L., Burdick, J. A., Linhardt, J. G., Kiick, K. L. Hydrophilic elastomeric biomaterials based on resilin-like polypeptides. Soft Matter. 5 (18), 3412-3416 (2009).
- Davis, K. A., Burke, K. A., Mather, P. T., Henderson, J. H. Dynamic cell behavior on shape memory polymer substrates. Biomaterials. 32 (9), 2285-2293 (2011).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophys J. 76 (4), 2307-2316 (1999).
- Gray, D. S., Tien, J., Chen, C. S. Repositioning of cells by mechanotaxis on surfaces with micropatterned youngs modulus. J Biomed Mater Res A. 66 (3), 605-614 (2003).
- Homma, M., Seida, Y., Nakano, Y. Effect of ions on the dynamic behavior of an electrodriven ionic polymer hydrogel membrane. Journal of applied Polymer Science. 82 (1), 76-80 (2001).
- Horkay, F., Tasaki, I., Basser, P. J. Osmotic swelling of polyacrylate hydrogels in physiological salt solutions. Biomacromolecules. 1 (1), 84-90 (2000).
- Jiang, F. X., Yurke, B., Firestein, B. L., Langrana, N. A. Neurite outgrowth on a DNA crosslinked hydrogel with tunable stiffnesses. Ann Biomed Eng. 36 (9), 1565-1579 (2008).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. Effect of dynamic stiffness of the substrates on neurite outgrowth by using a DNA-crosslinked hydrogel. Tissue Engineering Part A. 16 (6), 1873-1889 (2010).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. The relationship between fibroblast growth and the dynamic stiffnesses of a DNA crosslinked hydrogel. Biomaterials. 31 (6), 1199-1212 (2010).
- Kloxin, A. M., Tibbitt, M. W., Anseth, K. S. Synthesis of photodegradable hydrogels as dynamically tunable cell culture platforms. Nat Protoc. 5 (12), 1867-1887 (2010).
- Lin, D. C., Yurke, B., Langrana, N. A. Mechanical properties of a reversible, DNA-crosslinked polyacrylamide hydrogel. J Biomech Eng. 126 (1), 104-110 (2004).
- Lin, D. C., Yurke, B., Langrana, N. A. Use of rigid spherical inclusions in young’s moduli determination: Application to DNA-crosslinked gels. J Biomech Eng. 127 (4), 571-579 (2005).
- Luo, Y., Shoichet, M. S. Light-activated immobilization of biomolecules to agarose hydrogels for controlled cellular response. Biomacromolecules. 5 (6), 2315-2323 (2004).
- Marklein, R. A., Burdick, J. A. Spatially controlled hydrogel mechanics to modulate stem cell interactions. Soft Matter. 6 (1), 136-143 (2010).
- Pelham Jr, R. J., Wang, Y. Cell locomotion and focal adhesions are regulated by substrate flexibility. Proc Natl Acad Sci U S A. 94 (25), 13661-13665 (1997).
- Previtera, M. L., Chippada, U., Schloss, R. S., Yurke, B., Langrana, N. A. Mechanical properties of DNA-crosslinked polyacrylamide hydrogels with increasing crosslinker density. BioResearch Open Access. 1 (5), 256-259 (2012).
- Previtera, M. L., Langhammer, C. G., Firestein, B. L. Effects of substrate stiffness and cell density on primary hippocampal cultures. J Biosci Bioeng. 110 (4), 459-470 (2010).
- Previtera, M. L., Langhammer, C. G., Langrana, N. A., Firestein, B. L. Regulation of dendrite arborization by substrate stiffness is mediated by glutamate receptors. Ann Biomed Eng. 38 (12), 3733-3743 (2010).
- Previtera, M. L., Trout, K. L., Verma, D., Chippada, U., Schloss, R. S., Langrana, N. A. Fibroblast morphology on dynamic softening of hydrogels. Ann Biomed Eng. 40 (5), 1061-1072 (2012).
- Saxena, T., Gilbert, J., Stelzner, D., Hasenwinkel, J. Mechanical characterization of the injured spinal cord after lateral spinal hemisection injury in the rat. J Neurotrauma. 29 (9), 1747-1757 (2012).
- Sundararaghavan, H. G., Monteiro, G. A., Firestein, B. L., Shreiber, D. I. Neurite growth in 3d collagen gels with gradients of mechanical properties. Biotechnol Bioeng. 102 (2), 632-643 (2009).
- Wozniak, M. A., Chen, C. S. Mechanotransduction in development A growing role for contractility. Nat Rev Mol Cell Biol. 10 (1), 34-43 (2009).
- Wuerfel, J., et al. Mr-elastography reveals degradation of tissue integrity in multiple sclerosis. Neuroimage. 49 (3), 2520-2525 (2012).
- Zaari, N., Rajagopalan, P., Kim, S. K., Engler, A. J., Wong, J. Y. Photopolymerization in microfluidic gradient generators Microscale control of substrate compliance to manipulate cell response. Adv Mater. 16 (23-24), 2133-2137 (2004).

