Preparazione di DNA-reticolato poliacrilammide idrogel
Summary
Il nostro laboratorio ha sviluppato idrogel di poliacrilamide DNA-reticolato, un sistema dinamico di idrogel, per comprendere meglio gli effetti di modulazione rigidità dei tessuti sulla funzione delle cellule. Qui, forniamo schemi, descrizioni e protocolli per preparare questi idrogel.
Abstract
Mechanobiology è un settore scientifico emergente che affronta il ruolo critico di segnali fisici nel dirigere la morfologia e la funzione delle cellule. Ad esempio, l'effetto di elasticità dei tessuti sulla funzione delle cellule è un importante area di ricerca mechanobiology perché la rigidità del tessuto modula con la malattia, lo sviluppo, e lesioni. Materiali tessuto-mimando statiche, o materiali che non possono alterare la rigidezza volta che le cellule sono placcati, vengono prevalentemente utilizzati per studiare gli effetti della rigidità dei tessuti sulle funzioni cellulari. Mentre le informazioni raccolte da studi statici è prezioso, questi studi non sono indicativi della natura dinamica del microambiente cellulare in vivo. Per affrontare meglio gli effetti della rigidità dinamica sulla funzione delle cellule, abbiamo sviluppato un sistema di poliacrilammide idrogel DNA-reticolato (gel di DNA). A differenza di altri supporti dinamici, gel di DNA hanno la capacità di aumentare o diminuire la rigidità dopo la fabbricazione, senza stimoli. Gel di DNA sono costituiti da DNA crosslinchiostri che vengono polimerizzate in una spina dorsale di poliacrilamide. Aggiunta e rimozione di legami crociati con la consegna di DNA a singolo filamento permette temporale, spaziale, e il controllo reversibile di elasticità del gel. Abbiamo dimostrato in precedenti relazioni che la modulazione dinamica di elasticità del gel DNA influenza dei fibroblasti e neuroni comportamento. In questo rapporto e video, forniamo uno schema che descrive i meccanismi di reticolazione gel del DNA e passo-passo le istruzioni su gel di preparazione del DNA.
Introduction
Supporti statici e dinamici sono due categorie di biomateriali che sono stati sviluppati per studiare gli effetti di elasticità dei tessuti o di rigidità sulla funzione cellulare. Supporti statici sono in grado di cambiare le loro proprietà fisiche dopo che sono stati fabbricati e / o una volta che le cellule sono placcati. Poliacrilammide (PA) gel sono stati i primi bidimensionali, supporti statici che sono stati sintetizzati per le indagini mechanobiology 5,17. Gel PA sono facili da preparare, economico, versatile e può essere fabbricato con una vasta gamma di moduli elastici. Anche se questi vantaggi tecnici rendono PA gelifica un substrato comunemente applicato, supporti statici non sono indicativi della natura dinamica della matrice extracellulare (ECM) e di ambiente cellulare circostante in vivo. Ad esempio, l'ECM subisce alterazioni rigidità come risultato di lesioni, sviluppo, o malattia. Supporti dinamici sono quindi favoriti come modelli substrato di tessuto-mimando negli studi mechanobiology <sup> 22,24,25.
Numerosi sintetico, bidimensionale biomateriali, tridimensionali, statiche e dinamiche naturali sono stati sviluppati per imitare il tessuto rigidità 1,3,6,16,23,26. Alcuni supporti dinamici richiedono calore, ai raggi UV, corrente elettrica, gli ioni, e variazioni di pH per alterare le loro proprietà meccaniche 2,4,7,8,12,15,16, ma questi stimoli possono limitare bio-applicazione del idrogel. Idrogel di poliacrilamide DNA-reticolato (gel di DNA) sono substrati elastici bidimensionali dinamici. Legami crociati DNA permettono la modulazione temporale, spaziale, e reversibile di gel DNA rigidità con l'aggiunta di DNA a singola elica (ssDNA) a supporto o tampone 9-11,13,14,18,21. A differenza dei gel dinamici suddetti in cui si applicano stimoli per la modulazione di elasticità, i gel di DNA basano sulla diffusione di ssDNA applicata per la modifica di elasticità. Pertanto, la superficie del gel superiore, dove si coltivano cellule, è la prima zona modulato perché il tasso omodulazione elasticità f dipende dallo spessore del gel.
Gel di DNA sono simili alle loro controparti gel PA dal fatto che essi hanno un backbone poliacrilammide, tuttavia i legami crociati bis-acrilamide vengono sostituiti con legami crociati costituite da DNA (Figura 1). Due ssDNAs (SA1 e SA2) ibridano con un filamento reticolante (L2) per compensare i legami crociati DNA del gel. SA1 e SA2 hanno sequenze distinte che contengono sia una modifica Acrydite alla fine 5'per incorporazione efficace nella rete PA. Per la preparazione del gel, SA1 e SA2 sono individualmente polimerizzati in una spina dorsale PA e, in seguito, la SA1 e SA2 polimerizzato sono mescolati insieme. L2, il reticolante, viene aggiunto alla miscela SA1 e SA2. La sequenza di basi L2 è complementare ad entrambe le sequenze SA1 e SA2 e L2 si ibrida con SA1 SA2 più per formare i legami crociati DNA. , Elasticità del gel DNA iniziale è determinato da entrambe le concentrazioni L2 e reticolazione (tabelle 1 </strong> e 2). Gel di DNA contenenti uguali quantità stechiometriche di L2, SA1, SA2 e sono i gel più rigide perché SA1 e SA2 sono reticolati al 100% da L2 (designato come 100% gel). Concentrazioni minori di risulta L2 in una percentuale inferiore di reticolazione DNA e, quindi, più morbide gel di DNA. Gel a partire da 50% reticolato (designato come il 50% gel) sono stati costruiti 9-11.
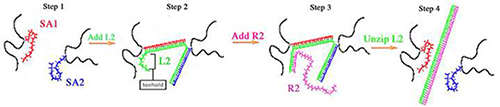
Figura 1 gel DNA reticolazione e uncrosslinking schematica 9-11,13,14,18,21 Passo 1:. SA1 (rosso) e SA2 (blu) sono individualmente polimerizzati in una spina dorsale poliacrilammide (nero). Dopo la polimerizzazione, soluzioni polimerizzati SA1 e SA2 sono mescolati insieme. Fase 2: L2 (verde) si aggiunge e si ibrida con SA1 SA2 più per formare i legami crociati del gel. Fase 3: R2 ibrida con tegli toehold di L2. Fase 4: ibridazione Toehold R2 spinge la decompressione di L2 da SA1 e SA2.
A differenza dei gel PA, gel DNA possono irrigidirsi e ammorbidire dopo la sintesi. Per questo motivo, le cellule coltivate su gel di DNA possono essere sottoposti a modifiche di rigidità dinamica. Per irrigidire gel cellule aderenti, L2 può essere aggiunto al mezzo di coltura di gel basse percentuali di aumentare la percentuale di legami crociati. Per ammorbidire gel cellule aderenti, L2 può essere rimosso per diminuire la percentuale di legami crociati 10,13,21. L2 ha una sequenza appiglio supplementare al fine di consentire 3'L2 a uncrosslink da SA1 e SA2 (Tabella 1). Rimozione di L2 è compiuta mediante ibridazione di un filamento inversione chiamato R2. R2 è complementare alla lunghezza della L2 e si ibrida con il primo appiglio L2. Toehold ibridazione spinge la decompressione di L2 da SA1 e SA2, che elimina la reticolazione e riduce la rigidità gel.
In questa relazione evideo, passo-passo le istruzioni sono fornite per la preparazione di irrigidimento e ammorbidire gel di DNA. Mentre 100% e 80% preparazioni gel sono descritti, questo protocollo può essere adattato per creare gel di DNA di altre percentuali reticolato iniziali e finali. In generale, 100% e 80% gel vengono preparati, immobilizzati su vetrini di vetro, funzionalizzati, e seminati con cellule. L2 viene aggiunto ai media di 80% gel e R2 si aggiunge ai media di 100% gel, 48 ore dopo la placcatura. L'aggiunta di L2 ai media irrigidisce 80% gel 100% reticolato, mentre l'aggiunta di R2 ai media ammorbidisce 100% gel al 80% reticolato. Gel irrigiditi sono designati come 80 → 100% gel e gel ammorbiditi sono designati come 100 → 80% gel nel testo. Per il controllo o gel statici, ssDNA composto da Ts o AS è consegnato ad un altro gruppo di 100% e 80% gel. Dopo un minimo di due giorni modulazione elasticità, le cellule possono essere elaborati ed analizzati.
<table border="0" cellpadding="0" cellspacing="0" fo:keep-together.within-page = "always">Tabella 1. sequenze di base per ssDNA 9-11,13,14,18,21. Cellulare e studi meccanici hanno utilizzato diversi ddisegni di reticolazione ifferent per generare gel di DNA con una gamma di statici e dinamici proprietà meccaniche. I parametri modulati nel design di reticolazione sono sequenza di basi e lunghezza della sequenza o la lunghezza di reticolazione. Caratteri in grassetto e in corsivo illustrano appaiamento delle basi tra SA1 e L2 e tra SA2 e L2 rispettivamente.
| Design | ||||||||
| 1 | 2 | 3 | ||||||
| Acrilamide Concentrazione (%) | 10 | 10 | 10 | 4 | ||||
| SA1 SA2 più ibridato a L2 (% reticolato) | 50 | 80 | 100 | 50 | 80 | 100 | 100 | 100 |
| <strong> Elasticità (kPa, media ± SEM) | 6.6 ± 0.6 | 17.1 ± 0.8 | 29.8 ± 2.5 | 5.85 ± 0.62 | 12.67 ± 1.33 | 22.88 ± 2.77 | 25.2 ± 0.5 | 10.4 ± 0.6 |
Tabella 2 Modulo di Young (E) del gel del DNA 9-11,13,14,18,21. Concentrazione di acrilamide, percentuale di reticolazione, e la lunghezza di reticolazione può essere modulata in gel di DNA. Designs 1, 2, e 3 hanno 20, 28, e 40 bp lunghezze di reticolazione, rispettivamente. 100% gel per tutti i disegni hanno moduli simili indicante la lunghezza di reticolazione non influenza l'elasticità del gel. Tuttavia, le variazioni della concentrazione di acrilamide alterano l'elasticità del gel del DNA.
Protocol
Representative Results
Discussion
La capacità del gel di DNA per ammorbidire o irrigidire prima e dopo l'adesione cellulare li rende un modello ideale per studiare il ruolo della rigidità dei tessuti dinamica sulla funzione delle cellule. Tutti e tre i disegni sono stati utilizzati in studi meccanici e biologici. Tuttavia, tutti e tre i disegni hanno elasticità simili in varie percentuali di reticolazione, indicante la lunghezza di reticolazione non influenza l'elasticità del gel del DNA (Tabella 2). Al contrario, la concent…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Gli autori desiderano ringraziare: Dr. Frank Jiang, il dottor David Lin, il Dr. Bernard Yurke e il Dr. Uday Chippada per i loro contributi sullo sviluppo della tecnologia del gel del DNA; Dr. Norell Hadzimichalis, Smit Shah, Kimberly Peterman, Robert Arter per i loro commenti e le modifiche di questo manoscritto; fonti di finanziamento, tra cui il New Jersey Commissione su Spinal Cord Research (Grant # 07A-019-SCR1, NAL) e New Jersey Neuroscience Institute (MLP); e editori di Ingegneria Tissutale, parte A, per il permesso di ristampare figure 2 e 4 e Biomateriali per il permesso di ristampare figura 3.
Materials
| ssDNA | Integrated DNA Technologies (Coralville, Iowa) idtdna.com |
Do not vortex ssDNA. Gentle invert the vial and/or pipette solution to mix. | |
| PBS with calcium and magnesium | Any brand. | ||
| 100X Tris-EDTA buffer (TE buffer) | Sigma-Aldrich (St. Loius, MO) sigmaldrich.com |
T9285 | |
| 10X Tris-Borate-EDTA buffer (TBE buffer) | Sigma-Aldrich (St. Loius, MO) | 93290 | TBE is a reproductive toxin. |
| 40% Acrylamide solution | Fisher Scientific (Pittsburg, PA) | BP14021 | Acrylamide is a toxin. |
| Ammonium persulfate (APS) | Sigma-Aldrich (St. Loius, MO) | A3678 | Prepare a 2% solution in TE buffer. APS is a toxin and irratant. |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich (St. Loius, MO) | T9281 | Prepare a 20% solution in TE buffer. TEMED is flammable, a corrosive, and a toxin. |
| 12-mm diameter round coverglass | Fisher Scientific (Pittsburg, PA) fishersci.com |
12-545-82 | |
| Norland optical adhesive 72 | Norland Products (Cranbury, NJ) norlandprod.com |
NOA72 | |
| 24-well tissue culture plate | Any brand. | ||
| Microcentrifuge tubes | Any brand. | ||
| Sulfo-SANPAH | ProteoChem or Thermo Fisher, (Rockland, IL) proteochem.com or thermofisher.com |
C111 or 22589 | Prepare a 0.315 mg/ml solution in water immediately before use. Dissolve at 37°C and filter sterilze. It is normal to observe undisolved sulfo-SANPAH in the filter. Sulfo-SANPAH is light sensitive and, therefore, the solution should be protect from light until UV exposure. |
| Poly-D-Lysine (PDL) | Sigma-Aldrich (St. Loius, MO) | P6407 | Prepare a 0.2 mg/ml solution in water and filter sterilize. |
| Collagen Type I | Affymetrix (Santa Clara, CA) affymetrix.com |
13813 | Prepare a 0.2 mg/ml solution in 0.2 N acetic acid. Solution needs to remain cold at all times to avoid polymerization. Acetic acid is a flammable, toxic, and corrosive. |
| 22 X 60 cover glass | Fisher Scientific (Pittsburg, PA) | 12-544-G | |
| Positive-displacement pipette | Gilson, Inc (Middletown, WI) gilson.com |
F148504 | |
| Heat block | Fisher Scientific (Pittsburg, PA) | 11-718 | |
| UV light source | Place gels as close as possible to the UV light. UV light can cause skin or eye injury. | ||
| Thermometer | Any brand. | ||
| Nitrogen gas | GTS-Welco (Flemington, NJ) www.praxairmidatlantic.com/ |
NI 5.0UH-R |
References
- Balaban, N. Q., et al. Force and focal adhesion assembly A close relationship studied using elastic micropatterned substrates. Nat Cell Biol. 3 (5), 466-472 (2001).
- Peppas Brannon-Peppas, L., Peppas, N. A. Dynamic and equilibrium swelling behaviour of ph-sensitive hydrogels containing 2-hydroxyethyl methacrylate. Biomaterials. 11 (9), 635-644 (1990).
- Charati, M. B., Ifkovits, J. L., Burdick, J. A., Linhardt, J. G., Kiick, K. L. Hydrophilic elastomeric biomaterials based on resilin-like polypeptides. Soft Matter. 5 (18), 3412-3416 (2009).
- Davis, K. A., Burke, K. A., Mather, P. T., Henderson, J. H. Dynamic cell behavior on shape memory polymer substrates. Biomaterials. 32 (9), 2285-2293 (2011).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophys J. 76 (4), 2307-2316 (1999).
- Gray, D. S., Tien, J., Chen, C. S. Repositioning of cells by mechanotaxis on surfaces with micropatterned youngs modulus. J Biomed Mater Res A. 66 (3), 605-614 (2003).
- Homma, M., Seida, Y., Nakano, Y. Effect of ions on the dynamic behavior of an electrodriven ionic polymer hydrogel membrane. Journal of applied Polymer Science. 82 (1), 76-80 (2001).
- Horkay, F., Tasaki, I., Basser, P. J. Osmotic swelling of polyacrylate hydrogels in physiological salt solutions. Biomacromolecules. 1 (1), 84-90 (2000).
- Jiang, F. X., Yurke, B., Firestein, B. L., Langrana, N. A. Neurite outgrowth on a DNA crosslinked hydrogel with tunable stiffnesses. Ann Biomed Eng. 36 (9), 1565-1579 (2008).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. Effect of dynamic stiffness of the substrates on neurite outgrowth by using a DNA-crosslinked hydrogel. Tissue Engineering Part A. 16 (6), 1873-1889 (2010).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. The relationship between fibroblast growth and the dynamic stiffnesses of a DNA crosslinked hydrogel. Biomaterials. 31 (6), 1199-1212 (2010).
- Kloxin, A. M., Tibbitt, M. W., Anseth, K. S. Synthesis of photodegradable hydrogels as dynamically tunable cell culture platforms. Nat Protoc. 5 (12), 1867-1887 (2010).
- Lin, D. C., Yurke, B., Langrana, N. A. Mechanical properties of a reversible, DNA-crosslinked polyacrylamide hydrogel. J Biomech Eng. 126 (1), 104-110 (2004).
- Lin, D. C., Yurke, B., Langrana, N. A. Use of rigid spherical inclusions in young’s moduli determination: Application to DNA-crosslinked gels. J Biomech Eng. 127 (4), 571-579 (2005).
- Luo, Y., Shoichet, M. S. Light-activated immobilization of biomolecules to agarose hydrogels for controlled cellular response. Biomacromolecules. 5 (6), 2315-2323 (2004).
- Marklein, R. A., Burdick, J. A. Spatially controlled hydrogel mechanics to modulate stem cell interactions. Soft Matter. 6 (1), 136-143 (2010).
- Pelham Jr, R. J., Wang, Y. Cell locomotion and focal adhesions are regulated by substrate flexibility. Proc Natl Acad Sci U S A. 94 (25), 13661-13665 (1997).
- Previtera, M. L., Chippada, U., Schloss, R. S., Yurke, B., Langrana, N. A. Mechanical properties of DNA-crosslinked polyacrylamide hydrogels with increasing crosslinker density. BioResearch Open Access. 1 (5), 256-259 (2012).
- Previtera, M. L., Langhammer, C. G., Firestein, B. L. Effects of substrate stiffness and cell density on primary hippocampal cultures. J Biosci Bioeng. 110 (4), 459-470 (2010).
- Previtera, M. L., Langhammer, C. G., Langrana, N. A., Firestein, B. L. Regulation of dendrite arborization by substrate stiffness is mediated by glutamate receptors. Ann Biomed Eng. 38 (12), 3733-3743 (2010).
- Previtera, M. L., Trout, K. L., Verma, D., Chippada, U., Schloss, R. S., Langrana, N. A. Fibroblast morphology on dynamic softening of hydrogels. Ann Biomed Eng. 40 (5), 1061-1072 (2012).
- Saxena, T., Gilbert, J., Stelzner, D., Hasenwinkel, J. Mechanical characterization of the injured spinal cord after lateral spinal hemisection injury in the rat. J Neurotrauma. 29 (9), 1747-1757 (2012).
- Sundararaghavan, H. G., Monteiro, G. A., Firestein, B. L., Shreiber, D. I. Neurite growth in 3d collagen gels with gradients of mechanical properties. Biotechnol Bioeng. 102 (2), 632-643 (2009).
- Wozniak, M. A., Chen, C. S. Mechanotransduction in development A growing role for contractility. Nat Rev Mol Cell Biol. 10 (1), 34-43 (2009).
- Wuerfel, J., et al. Mr-elastography reveals degradation of tissue integrity in multiple sclerosis. Neuroimage. 49 (3), 2520-2525 (2012).
- Zaari, N., Rajagopalan, P., Kim, S. K., Engler, A. J., Wong, J. Y. Photopolymerization in microfluidic gradient generators Microscale control of substrate compliance to manipulate cell response. Adv Mater. 16 (23-24), 2133-2137 (2004).

