Herstellung von DNA-vernetztem Polyacrylamid, Hydrogele
Summary
Unser Labor hat DNA-vernetzte Polyacrylamid-Hydrogele, ein dynamisches System Hydrogel entwickelt, um die Effekte der Modulation Gewebesteifigkeit auf die Zellfunktion zu verstehen. Hier bieten wir Schaltpläne, Beschreibungen und Protokolle, um diese Hydrogele vorzubereiten.
Abstract
Mechanobiologie ist ein aufstrebendes Forschungsgebiet, das die kritische Rolle der körperliche Signale in der Leitung der Zellmorphologie und Funktion adressiert. Zum Beispiel ist die Wirkung der Elastizität des Gewebes auf die Zellfunktion ein wichtiger Bereich der Forschung, da Mechanobiologie Gewebesteifigkeit moduliert mit der Krankheit, Entwicklung und Verletzungen. Statische Gewebe-ähnlichen Materialien oder Materialien, die nicht ändern kann, wenn die Steifigkeit Zellen plattiert werden hauptsächlich verwendet, um die Effekte der Gewebesteifigkeit auf die Zellfunktionen zu untersuchen. Während Informationen aus statischen Studien gesammelt wertvoll ist, sind diese Studien kein Hinweis auf die dynamische Natur der zellulären Mikroumgebung in vivo. Um die Wirkungen der dynamischen Steifigkeit auf die Zellfunktion besser erfüllen eine DNA-vernetzte Polyacrylamid-Hydrogel-System (DNS-Gele) entwickelt. Im Gegensatz zu anderen dynamischen Substraten, haben DNA-Gelen die Fähigkeit zu verringern oder zu erhöhen in der Steifigkeit nach der Herstellung ohne Reize. DNA-Gele bestehen aus DNA crosslTinten, die in einem Polyacrylamid-Grundgerüst polymerisiert werden. Hinzufügen und Entfernen von Vernetzungen durch Lieferung von Einzelstrang-DNA kann zeitlicher, räumlicher und reversible Steuerung Gelelastizität. Wir haben in früheren Berichten dargestellt, dass die dynamische Modulation der DNA-Gel Elastizität beeinflusst Fibroblasten und Neuronen Verhalten. In diesem Bericht und Video bieten wir eine schematische, die die DNA-Gel Vernetzungsmechanismen und Schritt-für-Schritt-Anleitung beschreibt auf die Vorbereitung DNA-Gelen.
Introduction
Statische und dynamische Substrate gibt zwei Kategorien von Biomaterialien, die entwickelt wurden, um die Wirkungen einer Gewebe Elastizität oder Steifigkeit auf die Zellfunktion untersucht werden. Statische Substrate sind nicht in der Lage, ihre physikalischen Eigenschaften ändern, nachdem sie hergestellt sind, und / oder einmal Zellen plattiert. Polyacrylamid (PA)-Gelen wurden die ersten zweidimensionalen statischen Substraten für Untersuchungen Mechanobiologie 5,17 synthetisiert. PA Gele sind leicht herzustellen, preiswert, flexibel und kann mit einer Vielzahl von elastischen Moduli hergestellt werden. Obwohl diese technischen Vorteile machen PA-Gele häufig angewendet Substrat, sind statische Substrate kein Hinweis auf die Dynamik der extrazellulären Matrix (ECM) und die umliegenden Zellumgebung in vivo. Zum Beispiel kann die ECM-Steifigkeit erfährt Änderungen als Folge von Verletzungen, die Entwicklung oder Krankheit. Dynamische Substrate werden daher als gehirnähnlichen Substrat Modelle in Mechanobiologie Studien begünstigt <sup> 22,24,25.
Zahlreiche synthetische, natürliche, zweidimensionalen, dreidimensionalen, statischen und dynamischen Biomaterialien wurden entwickelt, um Gewebesteifigkeit 1,3,6,16,23,26 imitieren. Einige dynamische Substrate benötigen Wärme, UV, elektrischen Strom, Ionen und pH-Änderungen, um ihre mechanischen Eigenschaften zu verändern 2,4,7,8,12,15,16, aber diese Reize Bio-Anwendung des Hydrogels zu beschränken. DNA-Polyacrylamid vernetzt Hydrogele (DNA Gele) sind dynamisch zweidimensionalen elastischen Substraten. DNA-Vernetzungen ermöglichen zeitlichen, räumlichen und reversible Modulation der DNA-Gel-Steifigkeit durch die Zugabe von Einzelstrang-DNA (ssDNA), um Medien oder Puffer 9-11,13,14,18,21. Im Gegensatz zu den oben erwähnten dynamischen Gele wo Stimuli zur Modulation der Elastizität aufgebracht, stützen sich die DNA Gele auf der Diffusion von Applied ssDNA zur Veränderung der Elastizität. Deshalb ist die obere Geloberfläche, wo Zellen gezüchtet werden, ist der erste Bereich moduliert, da die Geschwindigkeit of Elastizität Modulation hängt von der Gel-Dicke.
DNA-Gele sind ähnlich wie ihre Gegenstücke in PA-Gel, dass sie eine Polyacrylamid-Rückgrat haben jedoch die Bis-Acrylamid Vernetzungen mit Vernetzungen von DNA (Figur 1) zusammengesetzt ersetzt. Zwei ssDNAs (SA1 und SA2) hybridisieren mit einem Vernetzer Strang (L2) bilden die DNA-Quervernetzungen des Gels. SA1 und SA2 haben verschiedene Sequenzen, die sowohl eine ACRYDIT Modifikation am 5'Ende für eine effektive Einbindung in die PA-Netzwerk enthalten. Zur Herstellung des Gels, SA1 und SA2 werden einzeln in einem PA Gerüst polymerisiert und anschließend die polymerisierten SA1 und SA2 werden miteinander vermischt. L2, das Vernetzungsmittel wird zu der SA1 und SA2 Mischung zugegeben. Der L2-Basensequenz komplementär zu beiden SA1 und SA2 und L2 Sequenzen hybridisiert SA1 und SA2, um die DNA-Vernetzungen zu bilden. Initial, DNA-Gel Elastizität wird von beiden L2-Konzentrationen und Vernetzung (Tabellen 1 bestimmt </strong> und 2). DNA-Gelen, die gleiche stöchiometrische Mengen L2, SA1, SA2 und sind die härtesten Gele, weil SA1 und SA2 sind 100% vernetzt durch L2 (als 100% Gele bezeichnet). Niedrigere Konzentrationen von L2 zu einem niedrigeren Prozentsatz an DNA-Vernetzung und damit weicher DNA Gele. Gele so niedrig wie 50% vernetzt (zu 50% Gele bezeichnet) wurden konstruiert, 9-11.
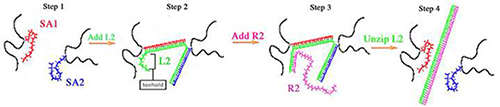
Abbildung 1. DNA-Gel Vernetzung und uncrosslinking schematische 9-11,13,14,18,21. Schritt 1: SA1 (rot) und SA2 (blau) werden einzeln in einem Polyacrylamid-Rückgrat (schwarz) polymerisiert. Nach der Polymerisation werden SA1 und SA2 polymerisiert Lösungen miteinander vermischt. Schritt 2: L2 (grün) zu und hybridisiert mit SA1 und SA2, die Vernetzungen des Gels. Schritt 3: R2 hybridisiert mit ter Ansatzpunkt von L2. Schritt 4: Brückenkopf-Hybridisierung von R2 treibt das Entpacken von L2 von SA1 und SA2.
Im Gegensatz zu PA-Gele können DNA-Gele zu versteifen und zu erweichen nach der Synthese. Aus diesem Grund können die Zellen auf DNA-Gelen gezüchtet, um dynamische Steifigkeit Änderungen unterzogen werden. Zellhaftenden Gelen erstarren kann L2 zu den Kulturmedien von geringen Prozentsatz Gels zugegeben werden, um den Prozentsatz von Vernetzungen zu erhöhen. Erweichen zellhaft Gele können L2 entfernt werden, um den Prozentsatz von Vernetzungen 10,13,21 verringern. L2 hat einen zusätzlichen Brückenkopf-Sequenz am 3'Ende zu ermöglichen L2 von SA1 und SA2 (Tabelle 1) uncrosslink. Entfernung von L2 durch Hybridisierung einer Umkehrstrang genannt R2 erreicht. R2 ist komplementär zu der vollen Länge von L2 und hybridisiert mit dem ersten Ansatzpunkt L2. Brückenkopf-Hybridisierung treibt das Entpacken von L2 von SA1 und SA2, die die Vernetzungs beseitigt und reduziert das Gel Steifigkeit.
In diesem Bericht undVideo, Schritt-für-Schritt-Anleitungen für die Herstellung von Versteifungs und Erweichung DNA-Gelen zur Verfügung gestellt. Während 100% und 80% Gel Präparate beschrieben, kann dieses Protokoll zugeschnitten werden, um DNA-Gelen anderer anfänglichen und endgültigen vernetzten Prozentsätze zu erstellen. Im allgemeinen sind 100% und 80% Gele hergestellt, auf Glas-Deckgläschen immobilisiert, funktionalisiert und mit Zellen besät. L2 ist mit den Medien 80% Gele gegeben und R2 ist mit dem Medium 100% Gelen, 48 h nach dem Plattieren zugegeben. Die Zugabe von L2 Medien versteift 80% bis 100% Gelen vernetzt, während die Zugabe von R2 Medien erweicht 100% Gele zu 80% vernetzt ist. Versteift Gele sind als 80 → 100% Gele bezeichnet und aufgeweicht Gele als 100 → 80% Gele im Text bezeichnet. Für die Kontrolle oder statische Gele, ssDNA aus Ts oder As ist, um einen weiteren Satz von 100% und 80% Gelen geliefert. Nach einem Minimum von zwei Tagen nach der Elastizität Modulations können Zellen verarbeitet und analysiert werden.
<table border="0" cellpadding="0" cellspacing="0" fo:keep-together.within-page = "always">Tabelle 1. Basissequenzen für ssDNA 9-11,13,14,18,21. Zelluläre und mechanischen Studien haben mehrere d verwendetifferent Vernetzungs Designs zur DNA-Gelen mit verschiedenen statischen und dynamischen mechanischen Eigenschaften zu erzeugen. Die in der Quer Design moduliert Parameter sind Basensequenz und Sequenzlänge oder vernetzen Länge. Bold und kursiv Schriften veranschaulichen Basenpaarung zwischen SA1 und L2 und zwischen SA2 und L2.
| Design | ||||||||
| 1 | 2 | 3 | ||||||
| Acrylamidkonzentration (%) | 10 | 10 | 10 | 4 | ||||
| SA1 und SA2 auf den L2-hybridisiert (% vernetzt) | 50 | 80 | 100 | 50 | 80 | 100 | 100 | 100 |
| <strong> Elastizität (kPa, Mittelwert ± SEM) | 6,6 ± 0,6 | 17,1 ± 0,8 | 29,8 ± 2,5 | 5,85 ± 0,62 | 12,67 ± 1,33 | 22.88 ± 2.77 | 25,2 ± 0,5 | 10,4 ± 0,6 |
Tabelle 2. Elastizitätsmodul (E) von DNA-Gelen 9-11,13,14,18,21. Acrylamidkonzentration, Vernetzungsprozent und Vernetzungs Länge kann in DNA-Gelen moduliert werden. Motive 1, 2 und 3 sind 20, 28 und 40 bp Vernetzungslängen sind. 100%-Gele für alle Ausführungen haben ähnliche Module anzeigt vernetzen Länge hat keinen Einfluss auf Gel-Elastizität. Variationen in Acrylamidkonzentration verändern jedoch DNA-Gel Elastizität.
Protocol
Representative Results
Discussion
Die Fähigkeit von DNA-Gelen zu erweichen oder zu versteifen, bevor und nachdem die Zelladhäsion macht sie zu einem idealen Modell, um die Rolle von Gewebesteifigkeit dynamisch auf die Zellfunktion untersucht werden. Alle drei Ausführungen sind in mechanischen und biologischen Untersuchungen verwendet. Allerdings haben alle drei Ausführungen ähnlich Elastizitäten bei verschiedenen Vernetzungsprozent anzeigt Vernetzungslänge nicht beeinflusst DNA Gel-Elastizität (Tabelle 2). Im Gegensatz dazu betr…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Die Autoren danken: Dr. Frank Jiang, Dr. David Lin, Dr. Bernard Yurke und Dr. Uday Chippada für ihre Beiträge zur Entwicklung der DNA-Gel-Technologie; Dr. Norell Hadzimichalis, Smit Shah, Kimberly Peterman, Robert Arter für ihre Kommentare und Bearbeitungen dieser Handschrift; Finanzierungsquellen, einschließlich der New Jersey Kommission auf Rückenmarksforschung (Grant # 07A-019-SCR1, NAL) und New Jersey Neuroscience Institute (MLP); und Herausgeber von Tissue Engineering, Teil A, für die Erlaubnis, 2 und 4 und Biomaterialien für die Erlaubnis, Abbildung 3 Reprint.
Materials
| ssDNA | Integrated DNA Technologies (Coralville, Iowa) idtdna.com |
Do not vortex ssDNA. Gentle invert the vial and/or pipette solution to mix. | |
| PBS with calcium and magnesium | Any brand. | ||
| 100X Tris-EDTA buffer (TE buffer) | Sigma-Aldrich (St. Loius, MO) sigmaldrich.com |
T9285 | |
| 10X Tris-Borate-EDTA buffer (TBE buffer) | Sigma-Aldrich (St. Loius, MO) | 93290 | TBE is a reproductive toxin. |
| 40% Acrylamide solution | Fisher Scientific (Pittsburg, PA) | BP14021 | Acrylamide is a toxin. |
| Ammonium persulfate (APS) | Sigma-Aldrich (St. Loius, MO) | A3678 | Prepare a 2% solution in TE buffer. APS is a toxin and irratant. |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich (St. Loius, MO) | T9281 | Prepare a 20% solution in TE buffer. TEMED is flammable, a corrosive, and a toxin. |
| 12-mm diameter round coverglass | Fisher Scientific (Pittsburg, PA) fishersci.com |
12-545-82 | |
| Norland optical adhesive 72 | Norland Products (Cranbury, NJ) norlandprod.com |
NOA72 | |
| 24-well tissue culture plate | Any brand. | ||
| Microcentrifuge tubes | Any brand. | ||
| Sulfo-SANPAH | ProteoChem or Thermo Fisher, (Rockland, IL) proteochem.com or thermofisher.com |
C111 or 22589 | Prepare a 0.315 mg/ml solution in water immediately before use. Dissolve at 37°C and filter sterilze. It is normal to observe undisolved sulfo-SANPAH in the filter. Sulfo-SANPAH is light sensitive and, therefore, the solution should be protect from light until UV exposure. |
| Poly-D-Lysine (PDL) | Sigma-Aldrich (St. Loius, MO) | P6407 | Prepare a 0.2 mg/ml solution in water and filter sterilize. |
| Collagen Type I | Affymetrix (Santa Clara, CA) affymetrix.com |
13813 | Prepare a 0.2 mg/ml solution in 0.2 N acetic acid. Solution needs to remain cold at all times to avoid polymerization. Acetic acid is a flammable, toxic, and corrosive. |
| 22 X 60 cover glass | Fisher Scientific (Pittsburg, PA) | 12-544-G | |
| Positive-displacement pipette | Gilson, Inc (Middletown, WI) gilson.com |
F148504 | |
| Heat block | Fisher Scientific (Pittsburg, PA) | 11-718 | |
| UV light source | Place gels as close as possible to the UV light. UV light can cause skin or eye injury. | ||
| Thermometer | Any brand. | ||
| Nitrogen gas | GTS-Welco (Flemington, NJ) www.praxairmidatlantic.com/ |
NI 5.0UH-R |
References
- Balaban, N. Q., et al. Force and focal adhesion assembly A close relationship studied using elastic micropatterned substrates. Nat Cell Biol. 3 (5), 466-472 (2001).
- Peppas Brannon-Peppas, L., Peppas, N. A. Dynamic and equilibrium swelling behaviour of ph-sensitive hydrogels containing 2-hydroxyethyl methacrylate. Biomaterials. 11 (9), 635-644 (1990).
- Charati, M. B., Ifkovits, J. L., Burdick, J. A., Linhardt, J. G., Kiick, K. L. Hydrophilic elastomeric biomaterials based on resilin-like polypeptides. Soft Matter. 5 (18), 3412-3416 (2009).
- Davis, K. A., Burke, K. A., Mather, P. T., Henderson, J. H. Dynamic cell behavior on shape memory polymer substrates. Biomaterials. 32 (9), 2285-2293 (2011).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophys J. 76 (4), 2307-2316 (1999).
- Gray, D. S., Tien, J., Chen, C. S. Repositioning of cells by mechanotaxis on surfaces with micropatterned youngs modulus. J Biomed Mater Res A. 66 (3), 605-614 (2003).
- Homma, M., Seida, Y., Nakano, Y. Effect of ions on the dynamic behavior of an electrodriven ionic polymer hydrogel membrane. Journal of applied Polymer Science. 82 (1), 76-80 (2001).
- Horkay, F., Tasaki, I., Basser, P. J. Osmotic swelling of polyacrylate hydrogels in physiological salt solutions. Biomacromolecules. 1 (1), 84-90 (2000).
- Jiang, F. X., Yurke, B., Firestein, B. L., Langrana, N. A. Neurite outgrowth on a DNA crosslinked hydrogel with tunable stiffnesses. Ann Biomed Eng. 36 (9), 1565-1579 (2008).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. Effect of dynamic stiffness of the substrates on neurite outgrowth by using a DNA-crosslinked hydrogel. Tissue Engineering Part A. 16 (6), 1873-1889 (2010).
- Jiang, F. X., Yurke, B., Schloss, R. S., Firestein, B. L., Langrana, N. A. The relationship between fibroblast growth and the dynamic stiffnesses of a DNA crosslinked hydrogel. Biomaterials. 31 (6), 1199-1212 (2010).
- Kloxin, A. M., Tibbitt, M. W., Anseth, K. S. Synthesis of photodegradable hydrogels as dynamically tunable cell culture platforms. Nat Protoc. 5 (12), 1867-1887 (2010).
- Lin, D. C., Yurke, B., Langrana, N. A. Mechanical properties of a reversible, DNA-crosslinked polyacrylamide hydrogel. J Biomech Eng. 126 (1), 104-110 (2004).
- Lin, D. C., Yurke, B., Langrana, N. A. Use of rigid spherical inclusions in young’s moduli determination: Application to DNA-crosslinked gels. J Biomech Eng. 127 (4), 571-579 (2005).
- Luo, Y., Shoichet, M. S. Light-activated immobilization of biomolecules to agarose hydrogels for controlled cellular response. Biomacromolecules. 5 (6), 2315-2323 (2004).
- Marklein, R. A., Burdick, J. A. Spatially controlled hydrogel mechanics to modulate stem cell interactions. Soft Matter. 6 (1), 136-143 (2010).
- Pelham Jr, R. J., Wang, Y. Cell locomotion and focal adhesions are regulated by substrate flexibility. Proc Natl Acad Sci U S A. 94 (25), 13661-13665 (1997).
- Previtera, M. L., Chippada, U., Schloss, R. S., Yurke, B., Langrana, N. A. Mechanical properties of DNA-crosslinked polyacrylamide hydrogels with increasing crosslinker density. BioResearch Open Access. 1 (5), 256-259 (2012).
- Previtera, M. L., Langhammer, C. G., Firestein, B. L. Effects of substrate stiffness and cell density on primary hippocampal cultures. J Biosci Bioeng. 110 (4), 459-470 (2010).
- Previtera, M. L., Langhammer, C. G., Langrana, N. A., Firestein, B. L. Regulation of dendrite arborization by substrate stiffness is mediated by glutamate receptors. Ann Biomed Eng. 38 (12), 3733-3743 (2010).
- Previtera, M. L., Trout, K. L., Verma, D., Chippada, U., Schloss, R. S., Langrana, N. A. Fibroblast morphology on dynamic softening of hydrogels. Ann Biomed Eng. 40 (5), 1061-1072 (2012).
- Saxena, T., Gilbert, J., Stelzner, D., Hasenwinkel, J. Mechanical characterization of the injured spinal cord after lateral spinal hemisection injury in the rat. J Neurotrauma. 29 (9), 1747-1757 (2012).
- Sundararaghavan, H. G., Monteiro, G. A., Firestein, B. L., Shreiber, D. I. Neurite growth in 3d collagen gels with gradients of mechanical properties. Biotechnol Bioeng. 102 (2), 632-643 (2009).
- Wozniak, M. A., Chen, C. S. Mechanotransduction in development A growing role for contractility. Nat Rev Mol Cell Biol. 10 (1), 34-43 (2009).
- Wuerfel, J., et al. Mr-elastography reveals degradation of tissue integrity in multiple sclerosis. Neuroimage. 49 (3), 2520-2525 (2012).
- Zaari, N., Rajagopalan, P., Kim, S. K., Engler, A. J., Wong, J. Y. Photopolymerization in microfluidic gradient generators Microscale control of substrate compliance to manipulate cell response. Adv Mater. 16 (23-24), 2133-2137 (2004).

